CBGBENCH: Benchmark grafik pengikat kompleks adalah tolok ukur untuk desain molekul sadar target generatif.
Kertas [DiffBP] | Kertas [D3FG] | Benchmark
Catatan
Ini adalah repositori kode resmi dari makalah 'CBGBench: Isi bagian dari grafik pengikat molekul protein', yang bertujuan untuk menyatukan desain molekul sadar target dengan implementasi kode tunggal. Sampai sekarang, kami telah memasukkan 7 metode seperti yang ditunjukkan di bawah ini:
| Model | Tautan kertas | GitHub |
|---|---|---|
| Saku2mol | https://arxiv.org/abs/2205.07249 | https://github.com/pengxingang/pocket2mol |
| Graphbp | https://arxiv.org/abs/2204.09410 | https://github.com/divelab/graphbp |
| Diffsbdd | https://arxiv.org/abs/2210.13695 | https://github.com/arneschneuing/diffsbdd |
| Difbp | https://arxiv.org/abs/2211.11214 | Inilah implementasi resmi. |
| TargetDiff | https://arxiv.org/abs/2303.03543 | https://github.com/guanjq/targetdiff |
| BENDERA | https://openreview.net/forum?id=rq13idf0f73 | https://github.com/zaixizhang/flag |
| D3FG | https://arxiv.org/abs/2306.13769 | Inilah implementasi resmi. |
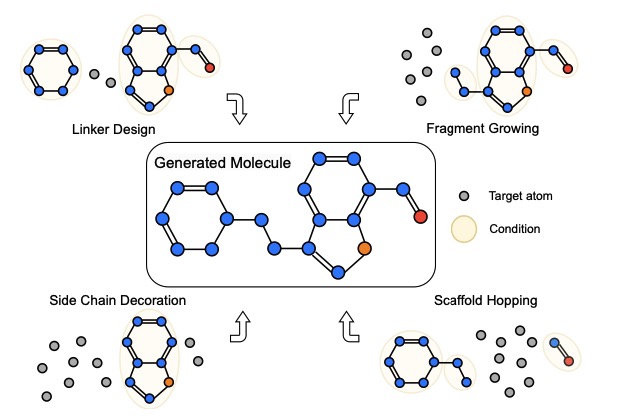
Model -model ini awalnya didirikan untuk generasi molekul de novo , dan kami memperluas lebih banyak tugas termasuk linker design , fragment growing , scaffold hopping , dan side chain decoration .
conda env create -f environment.yml
conda activate cbgbench
conda install pytorch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2 pytorch-cuda=11.8 -c pytorch -c nvidia
conda install pyg pytorch-scatter pytorch-cluster -c pyg
# install rdkit, efgs, obabel, etc.
pip install --use-pep517 EFGs
pip install biopython
pip install lxml
conda install rdkit openbabel tensorboard tqdm pyyaml easydict python-lmdb -c conda-forge
# install plip
mkdir tools
cd tools
git clone https://github.com/pharmai/plip.git
cd plip
python setup.py install
alias plip='python plip/plip/plipcmd.py'
cd ..
### Note that if there is an error in setup.py, it can be ignored as long as openbabel is installed.
# install docking tools
conda install -c conda-forge numpy swig boost-cpp sphinx sphinx_rtd_theme
python -m pip install git+https://github.com/Valdes-Tresanco-MS/AutoDockTools_py3
pip install meeko==0.1.dev3 scipy pdb2pqr vina
# If you are unable to install vina, you can try: conda install vina
# If you encounter the following error:
# ImportError: libtiff.so.5: cannot open shared object file: No such file or directory
# Please try the following steps to resolve it:
pip uninstall pillow
pip install pillow
(i) Unduh dataset yang diproses dari Google Drive. Untuk setiap dataset, ini sesuai dengan metode dan tugas yang berbeda. Silakan merujuk ke tabel di bawah ini untuk menemukan data yang diproses yang perlu Anda gunakan. Perhatikan bahwa untuk melatih D3FG, ini adalah model dua tahap yang pertama-tama menghasilkan kelompok fungsional dan kemudian menghasilkan atom linker, jadi kami menamai model dua tahap 'D3FG_FG' dan 'D3FG_LINKER', dan data pelatihan berbeda untuk mereka.
Tabel: Nama file data yang sesuai dengan berbagai metode dan tugas.
| model | tugas | Nama file |
|---|---|---|
| Saku2mol | de novo | Data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| Graphbp | de novo | Data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| Diffsbdd | de novo | Data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| Difbp | de novo | Data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| TargetDiff | de novo | Data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| BENDERA | de novo | Data/pl_arfg/crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb |
| D3FG_FG | de novo | Data/pl_fg/crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb |
| D3fg_linker | de novo | Data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Saku2mol | fragmen | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Graphbp | fragmen | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Diffsbdd | fragmen | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Difbp | fragmen | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| TargetDiff | fragmen | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Saku2mol | tautan | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Graphbp | tautan | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Diffsbdd | tautan | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Difbp | tautan | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| TargetDiff | tautan | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Saku2mol | perancah | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| Graphbp | perancah | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| Diffsbdd | perancah | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| Difbp | perancah | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| TargetDiff | perancah | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| Saku2mol | rantai samping | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Graphbp | rantai samping | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Diffsbdd | rantai samping | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Difbp | rantai samping | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| TargetDiff | rantai samping | Data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
(ii) Salin dataset ke ./data dir, di mana direktori file data lengkap akan terlihat seperti
- CGBBench
- data
- pl
- crossdocked_name2id.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- pl_arfg
- crossdocked_name2id_arfuncgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb
- pl_decomp
- crossdocked_name2id_frag.pt
- crossdocked_name2id_linker.pt
- crossdocked_name2id_scaffold.pt
- crossdocked_name2id_sidechain.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb
- pl_fg
- crossdocked_name2id_funcgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb
(i) Unduh crossdocked_v1.1_rmsd1.0.tar.gz dari TargetDiff Drive, dan salin ke ./raw_data/ dengan
mkdir raw_data
tar -xzvf crossdocked_v1.1_rmsd1.0.tar.gz ./raw_data
(ii) Jalankan yang berikut:
python ./scripts/extract_pockets.py --source raw_data/crossdocked_v1.1_rmsd1.0 --dest raw_data/crossdocked_v1.1_rmsd1.0_pocket10
(iii) Dalam pelatihan, dataset akan disiapkan untuk setiap tugas. Silakan merujuk pada Training .
Anda juga dapat mengunduh proses target file case_study dari Google drive, dan menyalinnya ke direktori ./data , yang akan mengarahkan dir ke
- CGBBench
- data
- case_study
- processed
- case_study_processed_fullatom.lmdb
- case_study_processed_funcgroup.lmdb
- case_study_name2id.pt
...
python train.py --config ./configs/{task}/train/{method}.yml --logdir ./logs/{task}/{method}
{Tugas} dapat diganti dengan denovo , linker , frag , scaffold dan sidechain , dan {Method} dapat diganti dengan nama model. Tabel berikut memberikan pasangan tugas metode terperinci dan penggantiannya.
Tabel: Pasangan metode-tugas yang digunakan untuk berlatih dari awal.
| Metode | Tugas | {Method} + {Tugas} |
|---|---|---|
| Saku2mol | de novo | Pocket2mol + Denovo |
| Graphbp | de novo | Graphbp + Denovo |
| Diffsbdd | de novo | DiffsBDD + Denovo |
| Difbp | de novo | DiffBP + Denovo |
| TargetDiff | de novo | TargetDiff + Denovo |
| BENDERA | de novo | Bendera + Denovo |
| D3FG | de novo | D3FG_FG + Denovo; D3FG_LINKER + DENOVO |
| Saku2mol | Desain Linker | Pocket2mol + linker |
| Graphbp | Desain Linker | GraphBP + Linker |
| Diffsbdd | Desain Linker | DiffsBDD + Linker |
| Difbp | Desain Linker | DiffBP + Linker |
| TargetDiff | Desain Linker | targetDiff + linker |
| Saku2mol | Fragmen tumbuh | saku2mol + frag |
| Graphbp | Fragmen tumbuh | graphbp + frag |
| Diffsbdd | Fragmen tumbuh | Diffsbdd + frag |
| Difbp | Fragmen tumbuh | DiffBP + Frag |
| TargetDiff | Fragmen tumbuh | targetDiff + frag |
| Saku2mol | Scaffold Hopping | Pocket2mol + scaffold |
| Graphbp | Scaffold Hopping | Graphbp + Scaffold |
| Diffsbdd | Scaffold Hopping | Diffsbdd + scaffold |
| Difbp | Scaffold Hopping | DiffBP + perancah |
| TargetDiff | Scaffold Hopping | targetDiff + scaffold |
| Saku2mol | dekorasi rantai samping | Pocket2mol + sidechain |
| Graphbp | dekorasi rantai samping | Graphbp + Sidechain |
| Diffsbdd | dekorasi rantai samping | Diffsbdd + sidechain |
| Difbp | dekorasi rantai samping | DiffBP + Sidechain |
| TargetDiff | dekorasi rantai samping | TargetDiff + Sidechain |
Perhatikan bahwa D3FG dan Flag tidak kompatibel dengan tugas yang diperluas, dan D3FG menggunakan strategi pelatihan 2-tahap, jadi jika Anda ingin melatih D3FG sendiri, Anda perlu menjalankan:
python train.py --config ./configs/denovo/train/d3fg_fg.yml --logdir ./logs/denovo/d3fg_fg
python train.py --config ./configs/denovo/train/d3fg_linker.yml --logdir ./logs/denovo/d3fg_linker
Todo: Tabel dengan model pretrained
Unduh pos pemeriksaan .pt pretrained yang Anda butuhkan, dan salin ke ./logs/ , yang akan mengarah ke struktur direktori berikut:
# TODO
- CBGBench
- logs
- denovo
- d3fg_fg
- pretrain
- checkpoints
- 951000.pt
- d3fg_linker
- pretrain
- checkpoints
- 4840000.pt
- diffbp
- pretrain
- checkpoints
- 4848000.pt
...
- frag
- diffbp
- pretrain
- checkpoints
- 476000.pt
...
...
Setelah model dilatih, Anda dapat menggambar sampel dari mereka di kantong uji, dengan yang berikut:
bash generate.sh --method {method} --task {task} --tag {tag} --checkpoint {ckpt_number}
Dalam perintah, {Method} dan {Task} pasangan dapat ditemukan di tabel sebelumnya pada pasangan metode-tugas. {TAG} harus diganti dengan selftrain atau pretrain , sesuai dengan pos pemeriksaan yang Anda gunakan. Jika parameter --CheckPoint disediakan tanpa nomor, itu akan secara otomatis menemukan file .pt terbaru. Jika nomor disediakan, itu akan menggunakan file pos pemeriksaan yang ditentukan.
Misalnya, jika Anda ingin menghasilkan sampel pada set tes untuk desain de novo dengan model targetDiff yang terlatih sendiri , Anda dapat menjalankan:
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint
Atau jika Anda ingin menguji pos pemeriksaan 100000-th.
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint 100000
Perhatikan bahwa D3FG menggunakan strategi generasi dua langkah, jadi perintahnya harus
bash generate.sh --method d3fg_fg --task denovo --tag {tag} --checkpoint # generate the functional groups
bash generate.sh --method d3fg_linker --task denovo --tag {tag} --checkpoint # generate the linkers
Jalankan berikut:
cd evaluate_scripts
bash evaluate.sh --method {method} --task {task} --tag {tag}
Misalnya, jika Anda telah melatih TargetDiff sendiri, jalankan
bash evaluate.sh --method targetdiff --task denovo --tag selftrain
bash evaluate.sh --method targetdiff --task frag --tag selftrain
bash evaluate.sh --method targetdiff --task linker --tag selftrain
bash evaluate.sh --method targetdiff --task scaffold --tag selftrain
bash evaluate.sh --method targetdiff --task sidechain --tag selftrain
Atau jika Anda telah mengunduh model pretrained, silakan jalankan
cd evaluate_scripts
bash bash evaluate.sh --method {method} --task {task} --tag pretrained
Gunakan model pretrained untuk menghasilkan molekul pada target dunia nyata secara fleksibel.
Sertakan lebih banyak model, seperti 3DSBDD, Decompdiff, Voxmol, dan Ligan.
Lepaskan model pretrained, uji kelayakan dan reproduktif begitu makalah ini diterbitkan.
Basis kode diinisialisasi oleh Haitao Lin, dengan CBGBench V1 sebagai motivasi kami; Pengoperasian Repositori Kode berikutnya akan dikelola oleh Guojiang Zhao dan tim Teknologi DP -nya.
Jika Anda menemukan repositori bermanfaat untuk penelitian atau proyek Anda, silakan mengutip kertas benchmark kami sebagai referensi:
@misc{lin2024cbgbench,
title={CBGBench: Fill in the Blank of Protein-Molecule Complex Binding Graph},
author={Haitao Lin and Guojiang Zhao and Odin Zhang and Yufei Huang and Lirong Wu and Zicheng Liu and Siyuan Li and Cheng Tan and Zhifeng Gao and Stan Z. Li},
year={2024},
eprint={2406.10840},
archivePrefix={arXiv},
}
Dan karya -karya bekas kami di DiffBP dan D3FG:
@misc{lin2024diffbpgenerativediffusion3d,
title={DiffBP: Generative Diffusion of 3D Molecules for Target Protein Binding},
author={Haitao Lin and Yufei Huang and Odin Zhang and Siqi Ma and Meng Liu and Xuanjing Li and Lirong Wu and Jishui Wang and Tingjun Hou and Stan Z. Li},
year={2024},
eprint={2211.11214},
archivePrefix={arXiv},
primaryClass={q-bio.BM},
url={https://arxiv.org/abs/2211.11214},
}
@inproceedings{
lin2023functionalgroupbased,
title={Functional-Group-Based Diffusion for Pocket-Specific Molecule Generation and Elaboration},
author={Haitao Lin and Yufei Huang and Odin Zhang and Yunfan Liu and Lirong Wu and Siyuan Li and Zhiyuan Chen and Stan Z. Li},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=lRG11M91dx}
}
Proyek ini dilisensikan berdasarkan ketentuan lisensi GPL-3.0.