CBGBench: el complejo de referencia de referencia de gráfico de enlace complejo es un punto de referencia para el diseño generativo de la molécula con el objetivo.
Papel [Diffbp] | Papel [D3FG] | Punto de referencia
Nota
Este es el repositorio de código oficial del documento 'CBGBench: complete el gráfico en blanco del gráfico de unión a la molécula de proteínas', cuyo objetivo es unificar el diseño de la molécula con el objetivo de la implementación de código único. Hasta ahora, hemos incluido 7 métodos como se muestra a continuación:
| Modelo | Enlace de papel | Github |
|---|---|---|
| Bolsillo2mol | https://arxiv.org/abs/2205.07249 | https://github.com/pengxingang/pocket2mol |
| GraphBP | https://arxiv.org/abs/2204.09410 | https://github.com/divelab/graphbp |
| Diffsbdd | https://arxiv.org/abs/2210.13695 | https://github.com/arneschneing/diffsbdd |
| Diffbp | https://arxiv.org/abs/2211.11214 | Aquí está la implementación oficial. |
| Targetdiff | https://arxiv.org/abs/2303.03543 | https://github.com/guanjq/targetdiff |
| BANDERA | https://openreview.net/forum?id=rq13idf0f73 | https://github.com/zaixizhang/flag |
| D3fg | https://arxiv.org/abs/2306.13769 | Aquí está la implementación oficial. |
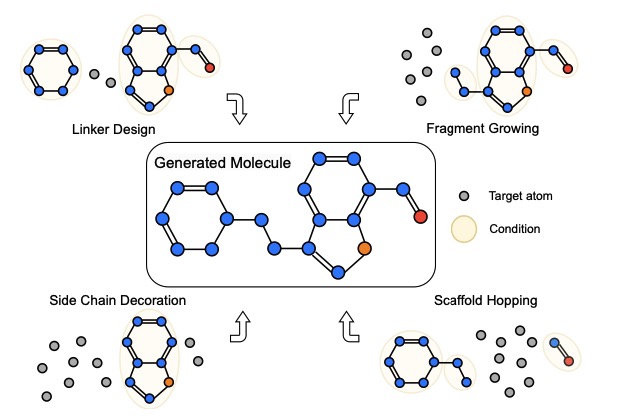
Estos modelos se establecen inicialmente para la generación de moléculas de novo , y ampliamos más tareas, incluyendo linker design , fragment growing , scaffold hopping y side chain decoration .
conda env create -f environment.yml
conda activate cbgbench
conda install pytorch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2 pytorch-cuda=11.8 -c pytorch -c nvidia
conda install pyg pytorch-scatter pytorch-cluster -c pyg
# install rdkit, efgs, obabel, etc.
pip install --use-pep517 EFGs
pip install biopython
pip install lxml
conda install rdkit openbabel tensorboard tqdm pyyaml easydict python-lmdb -c conda-forge
# install plip
mkdir tools
cd tools
git clone https://github.com/pharmai/plip.git
cd plip
python setup.py install
alias plip='python plip/plip/plipcmd.py'
cd ..
### Note that if there is an error in setup.py, it can be ignored as long as openbabel is installed.
# install docking tools
conda install -c conda-forge numpy swig boost-cpp sphinx sphinx_rtd_theme
python -m pip install git+https://github.com/Valdes-Tresanco-MS/AutoDockTools_py3
pip install meeko==0.1.dev3 scipy pdb2pqr vina
# If you are unable to install vina, you can try: conda install vina
# If you encounter the following error:
# ImportError: libtiff.so.5: cannot open shared object file: No such file or directory
# Please try the following steps to resolve it:
pip uninstall pillow
pip install pillow
(i) Descargue el conjunto de datos procesado de Google Drive. Para cada conjunto de datos, corresponde a diferentes métodos y tareas. Consulte la tabla a continuación para encontrar los datos procesados que necesita usar. Tenga en cuenta que para entrenar D3FG, es un modelo de dos etapas que genera primero grupos funcionales y luego genera átomos de enlace, por lo que nombramos el modelo de dos etapas 'D3FG_FG' y 'D3FG_Linker', y los datos de entrenamiento son diferentes para ellos.
Tabla: el nombre del archivo de datos correspondiente a diferentes métodos y tareas.
| modelo | tarea | Nombre del archivo |
|---|---|---|
| Bolsillo2mol | de novo | data/pl/Crossdocked_v1.1_rmsd1.0_pocket10_proced_fullatom.lmdb |
| GraphBP | de novo | data/pl/Crossdocked_v1.1_rmsd1.0_pocket10_proced_fullatom.lmdb |
| Diffsbdd | de novo | data/pl/Crossdocked_v1.1_rmsd1.0_pocket10_proced_fullatom.lmdb |
| Diffbp | de novo | data/pl/Crossdocked_v1.1_rmsd1.0_pocket10_proced_fullatom.lmdb |
| Targetdiff | de novo | data/pl/Crossdocked_v1.1_rmsd1.0_pocket10_proced_fullatom.lmdb |
| BANDERA | de novo | Data/pl_arfg/Crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb |
| D3fg_fg | de novo | data/pl_fg/Crossdocked_v1.1_rmsd1.0_pocket10_proced_funcgroup.lmdb |
| D3fg_linker | de novo | Data/PL/Crossdocked_v1.1_rmsd1.0_pocket10_proced_linker.lmdb |
| Bolsillo2mol | fragmento | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_proced_frag.lmdb |
| GraphBP | fragmento | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_proced_frag.lmdb |
| Diffsbdd | fragmento | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_proced_frag.lmdb |
| Diffbp | fragmento | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_proced_frag.lmdb |
| Targetdiff | fragmento | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_proced_frag.lmdb |
| Bolsillo2mol | enlazador | data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_proced_linker.lmdb |
| GraphBP | enlazador | data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_proced_linker.lmdb |
| Diffsbdd | enlazador | data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_proced_linker.lmdb |
| Diffbp | enlazador | data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_proced_linker.lmdb |
| Targetdiff | enlazador | data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_proced_linker.lmdb |
| Bolsillo2mol | andamio | data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_proced_scaffold.lmdb |
| GraphBP | andamio | data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_proced_scaffold.lmdb |
| Diffsbdd | andamio | data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_proced_scaffold.lmdb |
| Diffbp | andamio | data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_proced_scaffold.lmdb |
| Targetdiff | andamio | data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_proced_scaffold.lmdb |
| Bolsillo2mol | cadena lateral | Data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| GraphBP | cadena lateral | Data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Diffsbdd | cadena lateral | Data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Diffbp | cadena lateral | Data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Targetdiff | cadena lateral | Data/pl_decomp/Crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
(ii) Copie los conjuntos de datos a ./data dir, en el que se verá el directorio de archivos de datos completos
- CGBBench
- data
- pl
- crossdocked_name2id.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- pl_arfg
- crossdocked_name2id_arfuncgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb
- pl_decomp
- crossdocked_name2id_frag.pt
- crossdocked_name2id_linker.pt
- crossdocked_name2id_scaffold.pt
- crossdocked_name2id_sidechain.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb
- pl_fg
- crossdocked_name2id_funcgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb
(i) Descargue crossdocked_v1.1_rmsd1.0.tar.gz desde TargetDiff Drive, y cópielo a ./raw_data/ con
mkdir raw_data
tar -xzvf crossdocked_v1.1_rmsd1.0.tar.gz ./raw_data
(ii) Ejecute lo siguiente:
python ./scripts/extract_pockets.py --source raw_data/crossdocked_v1.1_rmsd1.0 --dest raw_data/crossdocked_v1.1_rmsd1.0_pocket10
(iii) En la capacitación, el conjunto de datos se preparará para cada tarea. Consulte Training .
Además, puede descargar el archivo de objetivos de procesos case_study desde Google Drive y copiarlo en el directorio ./data , lo que llevará al DIR a
- CGBBench
- data
- case_study
- processed
- case_study_processed_fullatom.lmdb
- case_study_processed_funcgroup.lmdb
- case_study_name2id.pt
...
python train.py --config ./configs/{task}/train/{method}.yml --logdir ./logs/{task}/{method}
{tarea} se puede reemplazar con denovo , linker , frag , scaffold y sidechain , y {método} se puede reemplazar con el nombre del modelo. La siguiente tabla proporciona los pares detallados de tareas de método y el reemplazo.
Tabla: pares de tareas de método utilizados para entrenar desde cero.
| Método | Tarea | {método} + {tarea} |
|---|---|---|
| Bolsillo2mol | de novo | Pocket2mol + Denovo |
| GraphBP | de novo | GraphBP + Denovo |
| Diffsbdd | de novo | diffsbdd + denovo |
| Diffbp | de novo | diffbp + denovo |
| Targetdiff | de novo | TargetDiff + Denovo |
| BANDERA | de novo | Bandera + Denovo |
| D3fg | de novo | d3fg_fg + denovo; D3FG_LINKER + DENOVO |
| Bolsillo2mol | diseño de enlazador | Pocket2mol + enlazador |
| GraphBP | diseño de enlazador | GraphBP + enlazador |
| Diffsbdd | diseño de enlazador | diffsbdd + enlazador |
| Diffbp | diseño de enlazador | Diffbp + enlazador |
| Targetdiff | diseño de enlazador | TargetDiff + enlazador |
| Bolsillo2mol | Fragmento de crecimiento | bolsillo2mol + frag |
| GraphBP | Fragmento de crecimiento | GraphBP + Frag |
| Diffsbdd | Fragmento de crecimiento | diffsbdd + frag |
| Diffbp | Fragmento de crecimiento | diffbp + frag |
| Targetdiff | Fragmento de crecimiento | TargetDiff + Frag |
| Bolsillo2mol | salto de andamio | Pocket2mol + andamio |
| GraphBP | salto de andamio | GraphBP + andamio |
| Diffsbdd | salto de andamio | diffsbdd + andamio |
| Diffbp | salto de andamio | Diffbp + andamio |
| Targetdiff | salto de andamio | Targetdiff + andamio |
| Bolsillo2mol | decoración de la cadena lateral | Pocket2mol + Sidechain |
| GraphBP | decoración de la cadena lateral | GraphBP + Sidechain |
| Diffsbdd | decoración de la cadena lateral | diffsbdd + sidechain |
| Diffbp | decoración de la cadena lateral | diffbp + sidechain |
| Targetdiff | decoración de la cadena lateral | TargetDiff + Sidechain |
Tenga en cuenta que D3FG y Flag no son compatibles con las tareas extendidas, y D3FG utiliza estrategias de entrenamiento de 2 etapas, por lo que si desea entrenar D3FG usted mismo, debe ejecutar:
python train.py --config ./configs/denovo/train/d3fg_fg.yml --logdir ./logs/denovo/d3fg_fg
python train.py --config ./configs/denovo/train/d3fg_linker.yml --logdir ./logs/denovo/d3fg_linker
TODO: Tabla con modelos previos a la aparición
Descargue los puntos de control .pt que necesita, y cottelas en ./logs/ , lo que conducirá a la siguiente estructura del directorio:
# TODO
- CBGBench
- logs
- denovo
- d3fg_fg
- pretrain
- checkpoints
- 951000.pt
- d3fg_linker
- pretrain
- checkpoints
- 4840000.pt
- diffbp
- pretrain
- checkpoints
- 4848000.pt
...
- frag
- diffbp
- pretrain
- checkpoints
- 476000.pt
...
...
Una vez que el modelo está entrenado, puede extraer muestras de ellas en los bolsillos de prueba, con lo siguiente:
bash generate.sh --method {method} --task {task} --tag {tag} --checkpoint {ckpt_number}
En el comando, el par {método} y {tarea} se pueden encontrar en la tabla anterior en pares de tareas de método. {etiqueta} debe reemplazarse con selftrain o pretrain , de acuerdo con los puntos de control que usa. Si se proporciona el parámetro --Checkpoint sin un número, encontrará automáticamente el último archivo .pt . Si se proporciona un número, usará el archivo de punto de control especificado.
Por ejemplo, si desea generar muestras en el conjunto de pruebas para el diseño de novo con el modelo TargetDiff autónomo , puede ejecutar:
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint
O si desea probar el punto de control del 100000-Th.
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint 100000
Tenga en cuenta que D3FG utiliza una estrategia de generación de dos pasos, por lo que el comando debe ser
bash generate.sh --method d3fg_fg --task denovo --tag {tag} --checkpoint # generate the functional groups
bash generate.sh --method d3fg_linker --task denovo --tag {tag} --checkpoint # generate the linkers
Ejecute lo siguiente:
cd evaluate_scripts
bash evaluate.sh --method {method} --task {task} --tag {tag}
Por ejemplo, si ha entrenado TargetDiff por usted mismo, corra
bash evaluate.sh --method targetdiff --task denovo --tag selftrain
bash evaluate.sh --method targetdiff --task frag --tag selftrain
bash evaluate.sh --method targetdiff --task linker --tag selftrain
bash evaluate.sh --method targetdiff --task scaffold --tag selftrain
bash evaluate.sh --method targetdiff --task sidechain --tag selftrain
O si ha descargado modelos previos a la aparición, ejecute
cd evaluate_scripts
bash bash evaluate.sh --method {method} --task {task} --tag pretrained
Use los modelos previos a la pretrada para generar moléculas en objetivos del mundo real de manera flexible.
Incluya más modelos, como 3DSBDD, Decompdiff, Voxmol y Ligan.
Libere los modelos previos a la petróleo, pruebe la viabilidad y la reproductividad una vez que se publica el documento.
La base de código es inicializada por Haitao Lin, con el Cbgbench V1 como nuestra motivación; La ejecución posterior del repositorio de código será administrado por Guojiang Zhao y su equipo de tecnología DP.
Si encuentra que el repositorio es útil para su investigación o proyectos, cite a nuestro documento de referencia como referencia:
@misc{lin2024cbgbench,
title={CBGBench: Fill in the Blank of Protein-Molecule Complex Binding Graph},
author={Haitao Lin and Guojiang Zhao and Odin Zhang and Yufei Huang and Lirong Wu and Zicheng Liu and Siyuan Li and Cheng Tan and Zhifeng Gao and Stan Z. Li},
year={2024},
eprint={2406.10840},
archivePrefix={arXiv},
}
Y nuestras obras anteriores de DIFFBP y D3FG:
@misc{lin2024diffbpgenerativediffusion3d,
title={DiffBP: Generative Diffusion of 3D Molecules for Target Protein Binding},
author={Haitao Lin and Yufei Huang and Odin Zhang and Siqi Ma and Meng Liu and Xuanjing Li and Lirong Wu and Jishui Wang and Tingjun Hou and Stan Z. Li},
year={2024},
eprint={2211.11214},
archivePrefix={arXiv},
primaryClass={q-bio.BM},
url={https://arxiv.org/abs/2211.11214},
}
@inproceedings{
lin2023functionalgroupbased,
title={Functional-Group-Based Diffusion for Pocket-Specific Molecule Generation and Elaboration},
author={Haitao Lin and Yufei Huang and Odin Zhang and Yunfan Liu and Lirong Wu and Siyuan Li and Zhiyuan Chen and Stan Z. Li},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=lRG11M91dx}
}
Este proyecto tiene licencia bajo los términos de la licencia GPL-3.0.