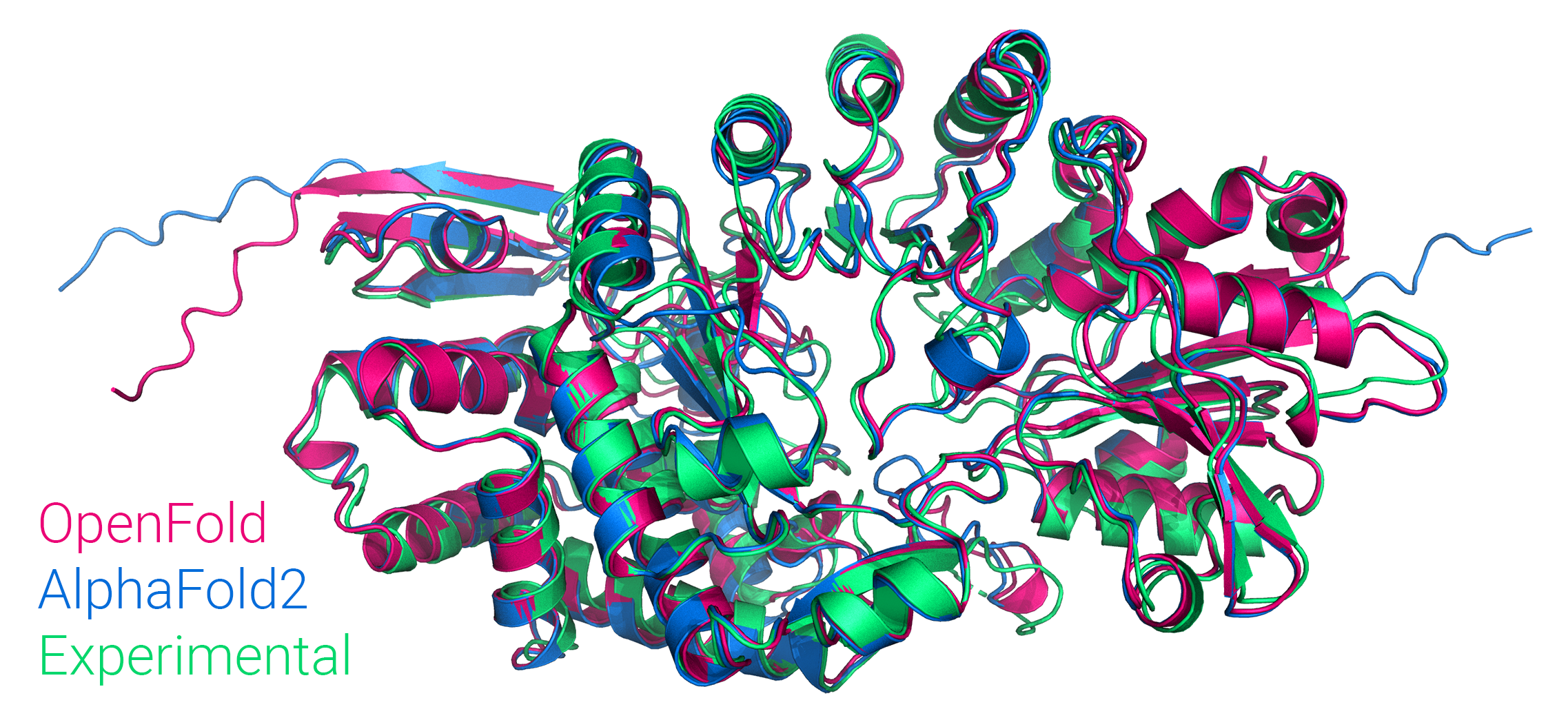
 Figura: Comparação de previsões de dobramento aberto e alphafold2 com a estrutura experimental do PDB 7KDX, cadeia B.
Figura: Comparação de previsões de dobramento aberto e alphafold2 com a estrutura experimental do PDB 7KDX, cadeia B.
Uma reprodução de Pytorch fiel, mas treinável, do Alphafold 2 de Deepmind.
Consulte nosso novo lar para documentos em openfold.readthedocs.io, com instruções para instalação e inferência/treinamento de modelos.
Grande parte do conteúdo desta página pode ser encontrada aqui.
Enquanto o Alphafold e, por extensão, o código -fonte do Openfold é licenciado sob a licença Apache permissiva, versão 2.0, parâmetros pré -treinados do DeepMind se enquadram no CC por licença 4.0, cuja cópia é baixada para openfold/resources/params pelo script de instalação. Observe que o último substitui a licença original e mais restritiva do CC BY-NC 4.0 a partir de janeiro de 2022.
Se você encontrar problemas usando o OpenFold, sinta -se à vontade para criar um problema! Também recebemos pedidos de puxão da comunidade.
Cite nosso artigo:
@article { Ahdritz2022.11.20.517210 ,
author = { Ahdritz, Gustaf and Bouatta, Nazim and Floristean, Christina and Kadyan, Sachin and Xia, Qinghui and Gerecke, William and O{textquoteright}Donnell, Timothy J and Berenberg, Daniel and Fisk, Ian and Zanichelli, Niccolò and Zhang, Bo and Nowaczynski, Arkadiusz and Wang, Bei and Stepniewska-Dziubinska, Marta M and Zhang, Shang and Ojewole, Adegoke and Guney, Murat Efe and Biderman, Stella and Watkins, Andrew M and Ra, Stephen and Lorenzo, Pablo Ribalta and Nivon, Lucas and Weitzner, Brian and Ban, Yih-En Andrew and Sorger, Peter K and Mostaque, Emad and Zhang, Zhao and Bonneau, Richard and AlQuraishi, Mohammed } ,
title = { {O}pen{F}old: {R}etraining {A}lpha{F}old2 yields new insights into its learning mechanisms and capacity for generalization } ,
elocation-id = { 2022.11.20.517210 } ,
year = { 2022 } ,
doi = { 10.1101/2022.11.20.517210 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/10.1101/2022.11.20.517210 } ,
eprint = { https://www.biorxiv.org/content/early/2022/11/22/2022.11.20.517210.full.pdf } ,
journal = { bioRxiv }
}Se você usa o OpenProteInset, cite também:
@misc { ahdritz2023openproteinset ,
title = { {O}pen{P}rotein{S}et: {T}raining data for structural biology at scale } ,
author = { Gustaf Ahdritz and Nazim Bouatta and Sachin Kadyan and Lukas Jarosch and Daniel Berenberg and Ian Fisk and Andrew M. Watkins and Stephen Ra and Richard Bonneau and Mohammed AlQuraishi } ,
year = { 2023 } ,
eprint = { 2308.05326 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
}Qualquer trabalho que cite o OpenFold também deve citar o alfafold e o multimador de alfafold, se aplicável.