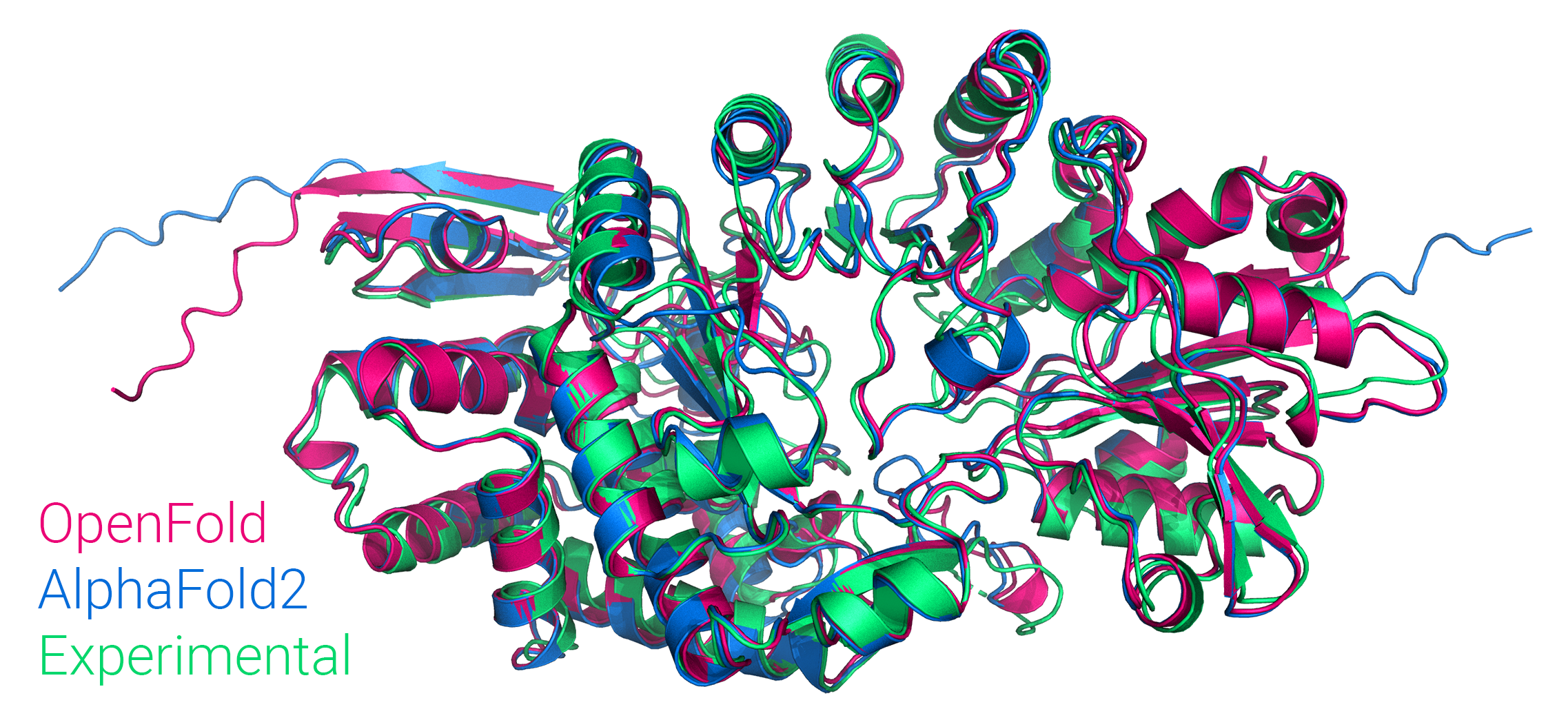
 Figura: Comparación de predicciones abiertas y alfafold2 con la estructura experimental de PDB 7KDX, cadena B.
Figura: Comparación de predicciones abiertas y alfafold2 con la estructura experimental de PDB 7KDX, cadena B.
Una reproducción de Pytorch fiel pero entrenable del Alfafold 2 de Deepmind.
Vea nuestro nuevo hogar para documentos en Openfold.ReadThedocs.io, con instrucciones de instalación e inferencia/capacitación del modelo.
Gran parte del contenido de esta página se puede encontrar aquí.
Mientras que el código fuente de Alfafold y, por extensión, el código fuente de Openfold se encuentra bajo la licencia Permisiva Apache, la versión 2.0, los parámetros previos a los pretrados de DeepMind se encuentran bajo la licencia CC por 4.0, una copia de la cual se descarga a openfold/resources/params mediante el script de instalación. Tenga en cuenta que este último reemplaza la licencia original y más restrictiva CC BY-NC 4.0 a partir de enero de 2022.
Si encuentra problemas con Openfold, ¡no dude en crear un problema! También damos la bienvenida a las solicitudes de la comunidad.
Por favor cita nuestro documento:
@article { Ahdritz2022.11.20.517210 ,
author = { Ahdritz, Gustaf and Bouatta, Nazim and Floristean, Christina and Kadyan, Sachin and Xia, Qinghui and Gerecke, William and O{textquoteright}Donnell, Timothy J and Berenberg, Daniel and Fisk, Ian and Zanichelli, Niccolò and Zhang, Bo and Nowaczynski, Arkadiusz and Wang, Bei and Stepniewska-Dziubinska, Marta M and Zhang, Shang and Ojewole, Adegoke and Guney, Murat Efe and Biderman, Stella and Watkins, Andrew M and Ra, Stephen and Lorenzo, Pablo Ribalta and Nivon, Lucas and Weitzner, Brian and Ban, Yih-En Andrew and Sorger, Peter K and Mostaque, Emad and Zhang, Zhao and Bonneau, Richard and AlQuraishi, Mohammed } ,
title = { {O}pen{F}old: {R}etraining {A}lpha{F}old2 yields new insights into its learning mechanisms and capacity for generalization } ,
elocation-id = { 2022.11.20.517210 } ,
year = { 2022 } ,
doi = { 10.1101/2022.11.20.517210 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/10.1101/2022.11.20.517210 } ,
eprint = { https://www.biorxiv.org/content/early/2022/11/22/2022.11.20.517210.full.pdf } ,
journal = { bioRxiv }
}Si usa OpenProteinset, también cite:
@misc { ahdritz2023openproteinset ,
title = { {O}pen{P}rotein{S}et: {T}raining data for structural biology at scale } ,
author = { Gustaf Ahdritz and Nazim Bouatta and Sachin Kadyan and Lukas Jarosch and Daniel Berenberg and Ian Fisk and Andrew M. Watkins and Stephen Ra and Richard Bonneau and Mohammed AlQuraishi } ,
year = { 2023 } ,
eprint = { 2308.05326 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
}Cualquier trabajo que cita Openfold también debe citar Alfafold y Alfafold-Multimer si corresponde.