ClusterProfShinyORA
1.0.0
这是一个基于Web的交互式应用程序,它包裹了流行的ClusterProfiler软件包,该软件包实现了分析和可视化基因组坐标,基因和基因簇的功能分布的方法。
用户可以从DESEQ2上传自己的差异基因表达(DGE)数据,也可以从上游DESEQ2SHINY应用程序导入数据。
该应用程序允许对GO-TERMS和KEGG途径进行快速简便的过度代表分析(ORA) 。
它旨在为研究人员提供一个直观的界面,以便在R.中进行交互式上载,分析,可视化和探索单细胞RNA-seq数据,而没有先前的编程知识。
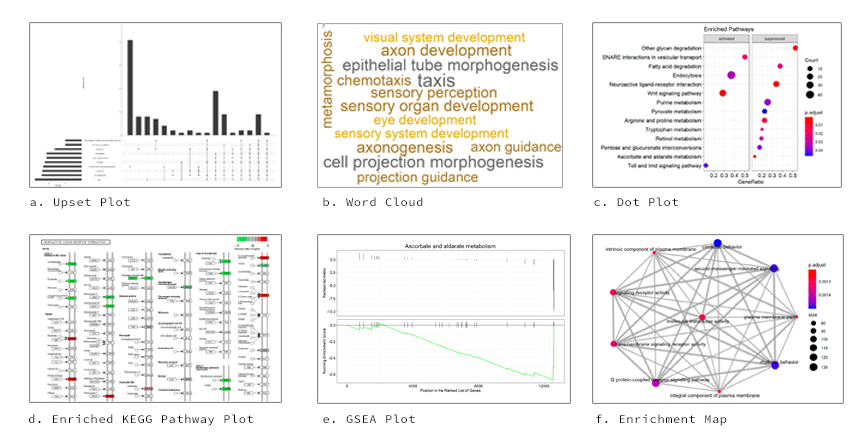
产生的视觉效果包括使用PathView软件包的DOT图,WordClouds,类别网图,富集地图图,GO诱导图,GSEA图和丰富的KEGG途径图。
该申请遵循本教程
请参阅下面的输出图。
本应用程序接受以下类型的输入数据:
出于演示目的,您可以选择“示例数据”
您可以按后按照步骤运行镜像教程的分析,以熟悉该应用
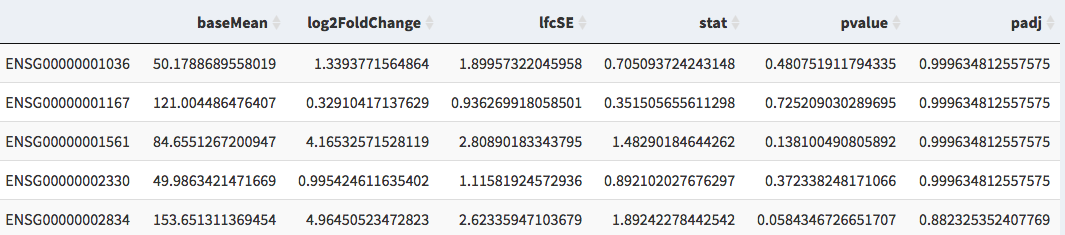
一个.csv/.txt文件,其中包含一个差分基因表达式(DGE)数据表
例如。 DESEQ2的输出
该文件可以是逗号或选项卡
所需的列是基因名称/ID,log2折叠更改,p调整值
您将必须选择匹配上面必需列的列名
对于示例文件,请单击此处
图2:例如。 DGE数据文件
输出结果将显示和/或可下载
对于go/kegg,包括各种形式的可视化:
广琴/群集生产商
穆罕默德·哈尔法(Mohammed Khalfan) - 代表分析