นี่คือแอปพลิเคชั่นแบบโต้ตอบบนเว็บที่ห่อหุ้มแพ็คเกจ ClusterProfiler ยอดนิยมซึ่งใช้วิธีการในการวิเคราะห์และแสดงภาพโปรไฟล์การทำงานของพิกัดจีโนม, ยีนและกลุ่มยีน
ผู้ใช้สามารถอัปโหลดข้อมูลการแสดงออกของยีนที่แตกต่างกัน (DGE) จาก DESEQ2 หรือนำเข้าข้อมูลจากแอพ Upstream Deseq2Shiny
แอพนี้ช่วยให้ การวิเคราะห์การเป็นตัวแทนที่รวดเร็วและง่ายดาย (ORA) ของเส้นทาง Go-terms และ Kegg
มันมีไว้เพื่อให้อินเทอร์เฟซที่ใช้งานง่ายสำหรับนักวิจัย ในการอัปโหลดวิเคราะห์แสดงภาพและสำรวจข้อมูล RNA-seq เซลล์เดียวแบบ โต้ตอบโดยไม่มีความรู้การเขียนโปรแกรมก่อนหน้าใน R.
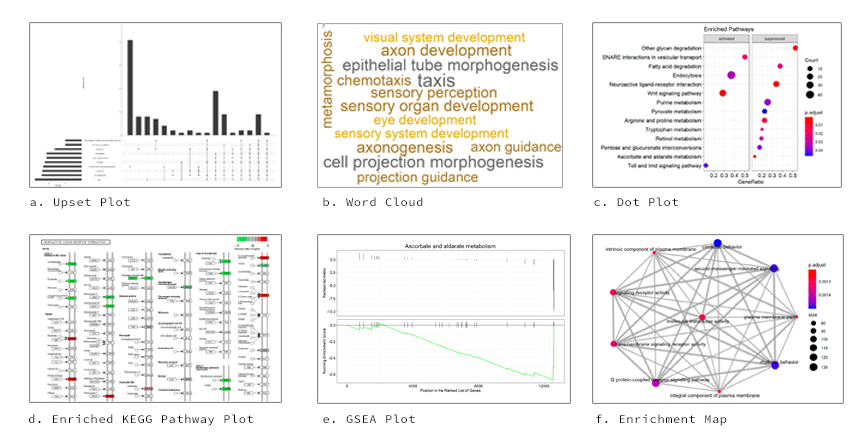
ภาพที่ผลิตได้รวมถึงพล็อต DOT, WordClouds, พล็อตสุทธิหมวดหมู่, พล็อตแผนที่เสริม, กราฟที่เหนี่ยวนำ, GSEA และพล็อตเส้นทาง KEGG ที่ได้รับการตกแต่งโดยใช้ แพ็คเกจ PathView
แอปพลิเคชันติดตามบทช่วยสอนนี้
ดูด้านล่างตัวอย่างเช่นพล็อตเอาท์พุท
แอปพลิเคชันนี้ยอมรับข้อมูลอินพุตประเภทต่อไปนี้:
เพื่อวัตถุประสงค์ในการสาธิตคุณสามารถเลือก "ตัวอย่างข้อมูล"
คุณสามารถทำตามขั้นตอนหลังจากนั้นเพื่อเรียกใช้การวิเคราะห์ที่สะท้อนการสอนเพื่อทำความคุ้นเคยกับแอป
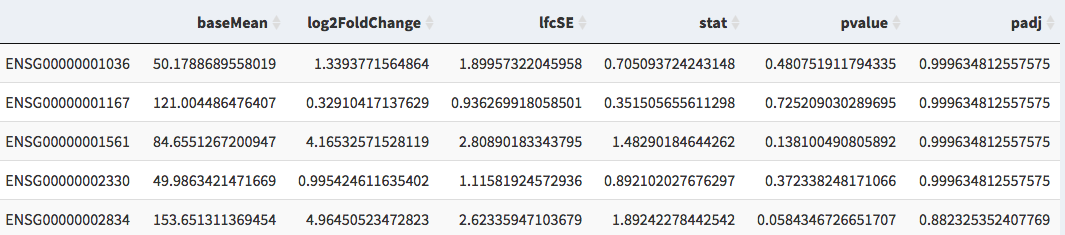
ไฟล์. csv/.txt ที่มี ตารางข้อมูลการแสดงออกของยีน (DGE)
เช่น เอาต์พุตของ deseq2
ไฟล์สามารถเป็น เครื่องหมายจุลภาคหรือแท็บคั่น
คอลัมน์ที่ต้องการคือ ชื่อยีน/id, การเปลี่ยนแปลงการพับ log2, ค่า p-adjusted
คุณจะต้องเลือกชื่อคอลัมน์ที่ตรงกับคอลัมน์ที่ต้องการข้างต้น
สำหรับไฟล์ตัวอย่างคลิกที่นี่
รูปที่ 2: เช่น ไฟล์ข้อมูล DGE
ผลลัพธ์ผลลัพธ์จะปรากฏขึ้นและ/หรือสามารถดาวน์โหลดได้
การสร้างภาพรูปแบบต่าง ๆ รวมอยู่ใน GO/KEGG:
guangchuangyu/clusterprofiler
Mohammed Khalfan - การวิเคราะห์การเป็นตัวแทนมากเกินไป