Este é um aplicativo interativo baseado na Web que envolve o pacote popular do clusterprofiler que implementa métodos para analisar e visualizar perfis funcionais de coordenadas genômicas, clusters de genes e genes.
Os usuários podem fazer upload de seus próprios dados de expressão de genes diferenciais (DGE) do DESEQ2 ou importar dados do aplicativo Upstream DeSeq2Shiny .
Este aplicativo permite uma análise rápida e fácil de representação (ORA) das vias Go-Terms e Kegg.
Destina-se a fornecer uma interface intuitiva para os pesquisadores fazer upload, analisar, visualizar e explorar e explorar facilmente os dados de RNA-seq de células únicas interativamente, sem nenhum conhecimento prévio de programação em R.
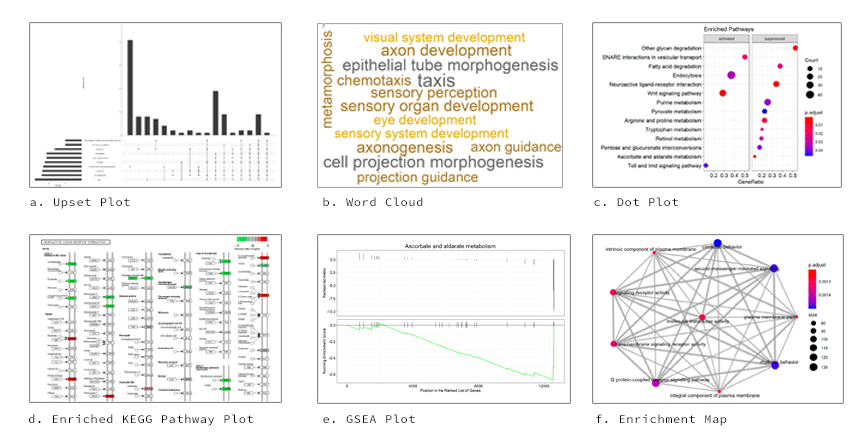
Os visuais produzidos incluem gráficos de pontos, clouds de palavras, gráficos de líquidos de categoria, gráficos de mapas de enriquecimento, gráficos induzidos por GO, gráficos GSEA e gráficos de via KEGG enriquecidos usando o pacote PathView .
O aplicativo segue este tutorial
Veja abaixo, por exemplo, gráficos de saída.
Este aplicativo aceita os seguintes tipos de dados de entrada:
Para fins de demonstração, você pode selecionar "Exemplo de dados"
Você pode seguir as etapas depois para executar a análise espelhando o tutorial para se familiarizar com o aplicativo
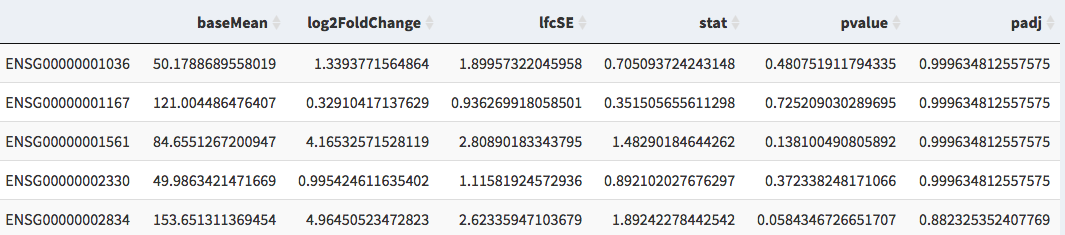
Um arquivo .csv/.txt que contém uma tabela de dados diferenciais de expressão gênica (DGE)
Por exemplo. a saída do deseseq2
O arquivo pode ser delimitado por vírgula ou guia
As colunas necessárias são nome/id de gene, alteração de dobras log2, valores ajustados para P
Você terá que selecionar os nomes das colunas que correspondem às colunas necessárias acima
Para um arquivo de amostra, clique aqui
Figura 2: por exemplo. Arquivo de dados DGE
Os resultados da saída serão exibidos e/ou são para download
Várias formas de visualizações estão incluídas para go/kegg:
Guangchuangyu/ClusterProfiler
Mohammed Khalfan - Análise de excesso de representação