Dies ist eine webbasierte interaktive Anwendung, die das beliebte Clusterprofiler- Paket umhüllt, das Methoden zur Analyse und Visualisierung funktionaler Profile von Genomkoordinaten, Gen- und Genclustern implementiert.
Benutzer können ihre eigenen DGE -Daten (Differential Gene Expression) aus DESEQ2 hochladen oder Daten aus der vorgelagerten DeSeq2Shiny -App importieren.
Diese App ermöglicht eine schnelle und einfache Überrepräsentationsanalyse (ORA) von GO-TERMS und KEGG-Wegen.
Es soll den Forschern eine intuitive Schnittstelle zur Verfügung stellen
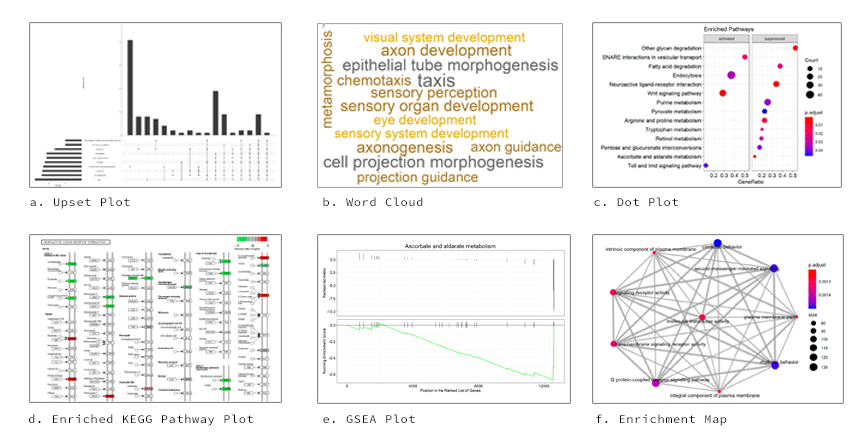
Zu den hergestellten Visuals gehören Punktdiagramme, Wordclouds, Kategorie -Netzdiagramme, Anreicherungskartendiagramme, GO -induzierte Diagramme, GSEA -Diagramme und angereicherte Kegg -Pfaddiagramme unter Verwendung des PathView -Pakets .
Die Anwendung folgt diesem Tutorial
Siehe unten, z. B. Ausgabediagramme.
Diese Anwendung akzeptiert die folgenden Arten von Eingabedaten:
Für Demo -Zwecke können Sie "Beispieldaten" auswählen
Sie können die Schritte danach befolgen, um die Analyse zu betreiben, die das Tutorial widerspiegelt, um mit der App vertraut zu werden
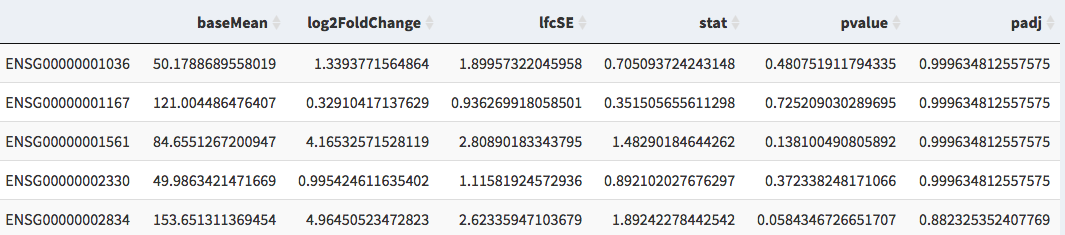
A .csv/.txt -Datei, die eine Tabelle der DGE -Daten der Differential -Genexpression (DGE) enthält
Z.B. die Ausgabe von DeSeq2
Die Datei kann entweder Komma oder Registerkarte abgegrenzt werden
Die erforderlichen Spalten sind Genname/ID, log2-falten Änderung, p-angepasste Werte
Sie müssen die Spaltennamen auswählen, die den oben genannten Spalten übereinstimmen
Klicken Sie für eine Beispieldatei hier
Abbildung 2: z. DGE -Datendatei
Die Ausgabergebnisse werden entweder angezeigt und/oder können heruntergeladen werden
Verschiedene Formen von Visualisierungen sind für GO/KEGG enthalten:
Guangchuangyu/Clusterprofiler
Mohammed Khalfan - Überrepräsentationsanalyse