ClusterProfShinyORA
1.0.0
這是一個基於Web的交互式應用程序,它包裹了流行的ClusterProfiler軟件包,該軟件包實現了分析和可視化基因組坐標,基因和基因簇的功能分佈的方法。
用戶可以從DESEQ2上傳自己的差異基因表達(DGE)數據,也可以從上游DESEQ2SHINY應用程序導入數據。
該應用程序允許對GO-TERMS和KEGG途徑進行快速簡便的過度代表分析(ORA) 。
它旨在為研究人員提供一個直觀的界面,以便在R.中進行交互式上載,分析,可視化和探索單細胞RNA-seq數據,而沒有先前的編程知識。
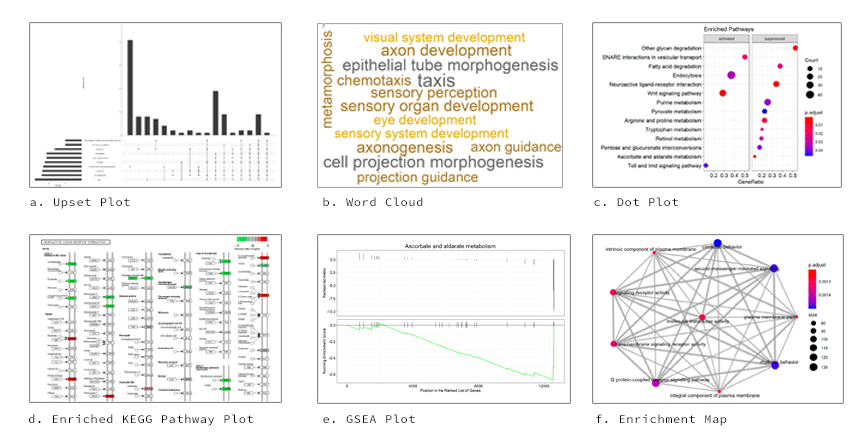
產生的視覺效果包括使用PathView軟件包的DOT圖,WordClouds,類別網圖,富集地圖圖,GO誘導圖,GSEA圖和豐富的KEGG途徑圖。
該申請遵循本教程
請參閱下面的輸出圖。
本應用程序接受以下類型的輸入數據:
出於演示目的,您可以選擇“示例數據”
您可以按後按照步驟運行鏡像教程的分析,以熟悉該應用
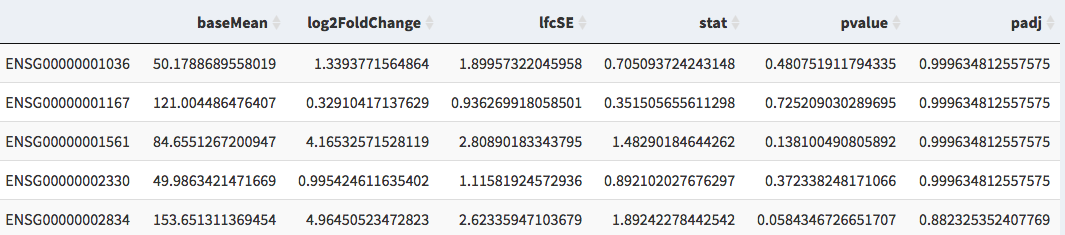
一個.csv/.txt文件,其中包含一個差分基因表達式(DGE)數據表
例如。 DESEQ2的輸出
該文件可以是逗號或選項卡
所需的列是基因名稱/ID,log2折疊更改,p調整值
您將必須選擇匹配上面必需列的列名
對於示例文件,請單擊此處
圖2:例如。 DGE數據文件
輸出結果將顯示和/或可下載
對於go/kegg,包括各種形式的可視化:
廣琴/群集生產商
穆罕默德·哈爾法(Mohammed Khalfan) - 代表分析