ตอนนี้กระดาษออนไลน์! https://arxiv.org/abs/2111.00595
เอกสารตอนนี้ออนไลน์! https://mlmed.org/torchxrayvision/
| (? วิดีโอโปรโมชั่น) - |
|---|
ห้องสมุดสำหรับชุดข้อมูลและรุ่นเอ็กซ์เรย์ทรวงอก รวมถึงรุ่นที่ผ่านการฝึกอบรมมาก่อน
TorchxrayVision เป็นไลบรารีซอฟต์แวร์โอเพ่นซอร์สสำหรับการทำงานกับชุดข้อมูล X-ray ทรวงอกและโมเดลการเรียนรู้ลึก มันมีอินเทอร์เฟซทั่วไปและห่วงโซ่การประมวลผลก่อนการประมวลผลทั่วไปสำหรับชุดข้อมูล X-ray หน้าอกที่เปิดเผยต่อสาธารณะ นอกจากนี้ยังมีรูปแบบการจำแนกประเภทและการเป็นตัวแทนจำนวนมากที่มีสถาปัตยกรรมที่แตกต่างกันซึ่งได้รับการฝึกฝนเกี่ยวกับการผสมผสานข้อมูลที่แตกต่างกันนั้นมีให้บริการผ่านห้องสมุดเพื่อใช้เป็นเส้นฐานหรือสารสกัดจากคุณลักษณะ
Twitter: @torchxrayvision
$ pip install torchxrayvision
import torchxrayvision as xrv
import skimage , torch , torchvision
# Prepare the image:
img = skimage . io . imread ( "16747_3_1.jpg" )
img = xrv . datasets . normalize ( img , 255 ) # convert 8-bit image to [-1024, 1024] range
img = img . mean ( 2 )[ None , ...] # Make single color channel
transform = torchvision . transforms . Compose ([ xrv . datasets . XRayCenterCrop (), xrv . datasets . XRayResizer ( 224 )])
img = transform ( img )
img = torch . from_numpy ( img )
# Load model and process image
model = xrv . models . DenseNet ( weights = "densenet121-res224-all" )
outputs = model ( img [ None ,...]) # or model.features(img[None,...])
# Print results
dict ( zip ( model . pathologies , outputs [ 0 ]. detach (). numpy ()))
{ 'Atelectasis' : 0.32797316 ,
'Consolidation' : 0.42933336 ,
'Infiltration' : 0.5316924 ,
'Pneumothorax' : 0.28849724 ,
'Edema' : 0.024142697 ,
'Emphysema' : 0.5011832 ,
'Fibrosis' : 0.51887786 ,
'Effusion' : 0.27805611 ,
'Pneumonia' : 0.18569896 ,
'Pleural_Thickening' : 0.24489835 ,
'Cardiomegaly' : 0.3645515 ,
'Nodule' : 0.68982 ,
'Mass' : 0.6392845 ,
'Hernia' : 0.00993878 ,
'Lung Lesion' : 0.011150705 ,
'Fracture' : 0.51916164 ,
'Lung Opacity' : 0.59073937 ,
'Enlarged Cardiomediastinum' : 0.27218717 }ตัวอย่างสคริปต์เพื่อประมวลผลภาพการใช้งานแบบจำลองที่ถูกกำหนดไว้คือ process_image.py
$ python3 process_image.py ../tests/00000001_000.png
{'preds': {'Atelectasis': 0.50500506,
'Cardiomegaly': 0.6600903,
'Consolidation': 0.30575264,
'Edema': 0.274184,
'Effusion': 0.4026162,
'Emphysema': 0.5036339,
'Enlarged Cardiomediastinum': 0.40989172,
'Fibrosis': 0.53293407,
'Fracture': 0.32376793,
'Hernia': 0.011924741,
'Infiltration': 0.5154413,
'Lung Lesion': 0.22231922,
'Lung Opacity': 0.2772148,
'Mass': 0.32237658,
'Nodule': 0.5091847,
'Pleural_Thickening': 0.5102617,
'Pneumonia': 0.30947986,
'Pneumothorax': 0.24847917}}
ระบุน้ำหนักสำหรับรุ่นที่ผ่านการฝึกอบรม (ปัจจุบันทั้งหมด densenet121) หมายเหตุ: แต่ละรุ่นที่ผ่านการฝึกอบรมมีเอาต์พุต 18 เอาต์พุต all รุ่นมีการฝึกอบรมทุกรูปแบบ อย่างไรก็ตามสำหรับน้ำหนักอื่น ๆ เป้าหมายบางอย่างไม่ได้รับการฝึกฝนและจะทำนายแบบสุ่มเนื่องจากไม่มีอยู่ในชุดข้อมูลการฝึกอบรม เอาต์พุตที่ถูกต้องเพียงรายการเดียวจะแสดงอยู่ในฟิลด์ {dataset}.pathologies บนชุดข้อมูลที่สอดคล้องกับน้ำหนัก
## 224x224 models
model = xrv . models . DenseNet ( weights = "densenet121-res224-all" )
model = xrv . models . DenseNet ( weights = "densenet121-res224-rsna" ) # RSNA Pneumonia Challenge
model = xrv . models . DenseNet ( weights = "densenet121-res224-nih" ) # NIH chest X-ray8
model = xrv . models . DenseNet ( weights = "densenet121-res224-pc" ) # PadChest (University of Alicante)
model = xrv . models . DenseNet ( weights = "densenet121-res224-chex" ) # CheXpert (Stanford)
model = xrv . models . DenseNet ( weights = "densenet121-res224-mimic_nb" ) # MIMIC-CXR (MIT)
model = xrv . models . DenseNet ( weights = "densenet121-res224-mimic_ch" ) # MIMIC-CXR (MIT)
# 512x512 models
model = xrv . models . ResNet ( weights = "resnet50-res512-all" )
# DenseNet121 from JF Healthcare for the CheXpert competition
model = xrv . baseline_models . jfhealthcare . DenseNet ()
# Official Stanford CheXpert model
model = xrv . baseline_models . chexpert . DenseNet ( weights_zip = "chexpert_weights.zip" )
# Emory HITI lab race prediction model
model = xrv . baseline_models . emory_hiti . RaceModel ()
model . targets - > [ "Asian" , "Black" , "White" ]
# Riken age prediction model
model = xrv . baseline_models . riken . AgeModel ()มาตรฐานของโหมดอยู่ที่นี่: benchmarks.md และประสิทธิภาพของบางรุ่นสามารถดูได้ในบทความนี้ arxiv.org/abs/2002.02497
นอกจากนี้คุณยังสามารถโหลดอัตโนมัติที่ผ่านการฝึกอบรมมาล่วงหน้าซึ่งได้รับการฝึกฝนบนชุดข้อมูล Padchest, NIH, Chexpert และเลียนแบบ
ae = xrv . autoencoders . ResNetAE ( weights = "101-elastic" )
z = ae . encode ( image )
image2 = ae . decode ( z )คุณสามารถโหลดแบบจำลองการแบ่งส่วนทางกายวิภาคที่ผ่านการฝึกอบรม สมุดบันทึกการสาธิต
seg_model = xrv . baseline_models . chestx_det . PSPNet ()
output = seg_model ( image )
output . shape # [1, 14, 512, 512]
seg_model . targets # ['Left Clavicle', 'Right Clavicle', 'Left Scapula', 'Right Scapula',
# 'Left Lung', 'Right Lung', 'Left Hilus Pulmonis', 'Right Hilus Pulmonis',
# 'Heart', 'Aorta', 'Facies Diaphragmatica', 'Mediastinum', 'Weasand', 'Spine'] 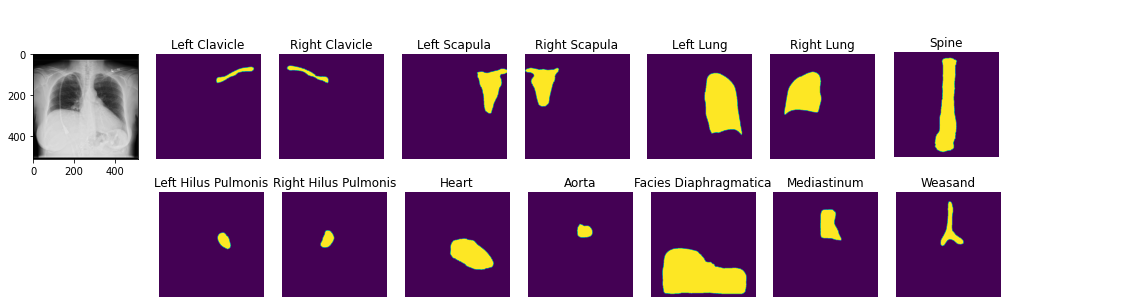
ดู docStrings สำหรับรายละเอียดเพิ่มเติมเกี่ยวกับแต่ละชุดข้อมูลและสมุดบันทึกตัวอย่างและสคริปต์การโหลดตัวอย่าง
transform = torchvision . transforms . Compose ([ xrv . datasets . XRayCenterCrop (),
xrv . datasets . XRayResizer ( 224 )])
# RSNA Pneumonia Detection Challenge. https://pubs.rsna.org/doi/full/10.1148/ryai.2019180041
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "path to stage_2_train_images_jpg" ,
transform = transform )
# CheXpert: A Large Chest Radiograph Dataset with Uncertainty Labels and Expert Comparison. https://arxiv.org/abs/1901.07031
d_chex = xrv . datasets . CheX_Dataset ( imgpath = "path to CheXpert-v1.0-small" ,
csvpath = "path to CheXpert-v1.0-small/train.csv" ,
transform = transform )
# National Institutes of Health ChestX-ray8 dataset. https://arxiv.org/abs/1705.02315
d_nih = xrv . datasets . NIH_Dataset ( imgpath = "path to NIH images" )
# A relabelling of a subset of NIH images from: https://pubs.rsna.org/doi/10.1148/radiol.2019191293
d_nih2 = xrv . datasets . NIH_Google_Dataset ( imgpath = "path to NIH images" )
# PadChest: A large chest x-ray image dataset with multi-label annotated reports. https://arxiv.org/abs/1901.07441
d_pc = xrv . datasets . PC_Dataset ( imgpath = "path to image folder" )
# COVID-19 Image Data Collection. https://arxiv.org/abs/2006.11988
d_covid19 = xrv . datasets . COVID19_Dataset () # specify imgpath and csvpath for the dataset
# SIIM Pneumothorax Dataset. https://www.kaggle.com/c/siim-acr-pneumothorax-segmentation
d_siim = xrv . datasets . SIIM_Pneumothorax_Dataset ( imgpath = "dicom-images-train/" ,
csvpath = "train-rle.csv" )
# VinDr-CXR: An open dataset of chest X-rays with radiologist's annotations. https://arxiv.org/abs/2012.15029
d_vin = xrv . datasets . VinBrain_Dataset ( imgpath = ".../train" ,
csvpath = ".../train.csv" )
# National Library of Medicine Tuberculosis Datasets. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4256233/
d_nlmtb = xrv . datasets . NLMTB_Dataset ( imgpath = "path to MontgomerySet or ChinaSet_AllFiles" )ชุดข้อมูลแต่ละชุดมีจำนวนฟิลด์จำนวนมาก ฟิลด์เหล่านี้จะได้รับการดูแลเมื่อ xrv.datasets.subset_dataset และ xrv.datasets.merge_dataset ใช้
.pathologies ฟิลด์นี้เป็นรายการของโรคที่มีอยู่ในชุดข้อมูลนี้ที่จะอยู่ในฟิลด์ .labels labels]
.labels ฟิลด์นี้มี 1,0 หรือ NAN สำหรับแต่ละฉลากที่กำหนดไว้ใน .pathologies
.csv ฟิลด์นี้เป็น dataframe pandas ของไฟล์เมตาดาต้า CSV ที่มาพร้อมกับข้อมูล แต่ละแถวสอดคล้องกับองค์ประกอบของชุดข้อมูลดังนั้นการจัดทำดัชนีโดยใช้ .iloc จะทำงาน
ถ้าเป็นไปได้. .csv ของแต่ละชุดข้อมูลจะมีฟิลด์ทั่วไปของ CSV สิ่งเหล่านี้จะได้รับการจัดตำแหน่งเมื่อรายการมีดังนี้:
csv.patientid id ที่ไม่ซ้ำกันซึ่งจะระบุตัวอย่างในชุดข้อมูลนี้
csv.offset_day_int ชดเชยเวลาจำนวนเต็มสำหรับภาพในหน่วยของวัน สิ่งนี้คาดว่าจะเป็นเวลาที่สัมพันธ์กันและไม่มีความหมายที่แน่นอนแม้ว่าสำหรับชุดข้อมูลบางชุดเป็นเวลายุค
csv.age_years อายุของผู้ป่วยในปี
csv.sex_male ถ้าผู้ป่วยเป็นผู้ชาย
csv.sex_female ถ้าผู้ป่วยเป็นเพศหญิง
Relabel_Dataset จะจัดตำแหน่งป้ายกำกับให้มีลำดับเช่นเดียวกับอาร์กิวเมนต์ของโรค
xrv . datasets . relabel_dataset ( xrv . datasets . default_pathologies , d_nih ) # has side effectsระบุชุดย่อยของมุมมอง (Demo Notebook)
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "..." ,
views = [ "PA" , "AP" , "AP Supine" ])ระบุเพียง 1 ภาพต่อผู้ป่วย
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "..." ,
unique_patients = True )รับสถิติสรุปต่อชุดข้อมูล
d_chex = xrv . datasets . CheX_Dataset ( imgpath = "CheXpert-v1.0-small" ,
csvpath = "CheXpert-v1.0-small/train.csv" ,
views = [ "PA" , "AP" ], unique_patients = False )
CheX_Dataset num_samples = 191010 views = [ 'PA' , 'AP' ]
{ 'Atelectasis' : { 0.0 : 17621 , 1.0 : 29718 },
'Cardiomegaly' : { 0.0 : 22645 , 1.0 : 23384 },
'Consolidation' : { 0.0 : 30463 , 1.0 : 12982 },
'Edema' : { 0.0 : 29449 , 1.0 : 49674 },
'Effusion' : { 0.0 : 34376 , 1.0 : 76894 },
'Enlarged Cardiomediastinum' : { 0.0 : 26527 , 1.0 : 9186 },
'Fracture' : { 0.0 : 18111 , 1.0 : 7434 },
'Lung Lesion' : { 0.0 : 17523 , 1.0 : 7040 },
'Lung Opacity' : { 0.0 : 20165 , 1.0 : 94207 },
'Pleural Other' : { 0.0 : 17166 , 1.0 : 2503 },
'Pneumonia' : { 0.0 : 18105 , 1.0 : 4674 },
'Pneumothorax' : { 0.0 : 54165 , 1.0 : 17693 },
'Support Devices' : { 0.0 : 21757 , 1.0 : 99747 }}มาสก์มีอยู่ในชุดข้อมูลต่อไปนี้:
xrv . datasets . RSNA_Pneumonia_Dataset () # for Lung Opacity
xrv . datasets . SIIM_Pneumothorax_Dataset () # for Pneumothorax
xrv . datasets . NIH_Dataset () # for Cardiomegaly, Mass, Effusion, ...ตัวอย่างการใช้งาน:
d_rsna = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "stage_2_train_images_jpg" ,
views = [ "PA" , "AP" ],
pathology_masks = True )
# The has_masks column will let you know if any masks exist for that sample
d_rsna . csv . has_masks . value_counts ()
False 20672
True 6012
# Each sample will have a pathology_masks dictionary where the index
# of each pathology will correspond to a mask of that pathology (if it exists).
# There may be more than one mask per sample. But only one per pathology.
sample [ "pathology_masks" ][ d_rsna . pathologies . index ( "Lung Opacity" )] นอกจากนี้ยังทำงานร่วมกับ data_augmentation หากคุณผ่าน data_aug=data_transforms ไปยัง dataloader เมล็ดสุ่มถูกจับคู่เพื่อจัดแนวการเรียกใช้ภาพและหน้ากาก
คลาส xrv.datasets.CovariateDataset ใช้ชุดข้อมูลสองชุดและสองอาร์เรย์ที่แสดงฉลาก ตัวอย่างจะถูกส่งกลับด้วยอัตราส่วนที่ต้องการของภาพจากแต่ละไซต์ เป้าหมายที่นี่คือการจำลองการเปลี่ยนแปลง covariate เพื่อให้โมเดลมุ่งเน้นไปที่คุณสมบัติที่ไม่ถูกต้อง จากนั้นการเปลี่ยนแปลงสามารถย้อนกลับในข้อมูลการตรวจสอบทำให้เกิดความล้มเหลวหายนะในประสิทธิภาพการวางนัยทั่วไป
อัตราส่วน = 0.0 หมายถึงภาพจาก D1 จะมีอัตราส่วนฉลากเป็นบวก = 0.5 หมายถึงภาพจาก D1 จะมีครึ่งหนึ่งของอัตราส่วนฉลากบวก = 1.0 หมายถึงภาพจาก D1 จะไม่มีฉลากบวก
ด้วยอัตราส่วนใด ๆ จำนวนตัวอย่างที่ส่งคืนจะเหมือนกัน
d = xrv . datasets . CovariateDataset ( d1 = # dataset1 with a specific condition
d1_target = #target label to predict,
d2 = # dataset2 with a specific condition
d2_target = #target label to predict,
mode = "train" , # train, valid, and test
ratio = 0.9 )กระดาษหลัก TorchxrayVision: https://arxiv.org/abs/2111.00595
Joseph Paul Cohen, Joseph D. Viviano, Paul Bertin, Paul Morrison, Parsa Torabian, Matteo Guarrera, Matthew P Lungren, Akshay Chaudhari, Rupert Brooks, Mohammad Hashir, Hadrien Bertrand
TorchXRayVision: A library of chest X-ray datasets and models.
Medical Imaging with Deep Learning
https://github.com/mlmed/torchxrayvision, 2020
@inproceedings{Cohen2022xrv,
title = {{TorchXRayVision: A library of chest X-ray datasets and models}},
author = {Cohen, Joseph Paul and Viviano, Joseph D. and Bertin, Paul and Morrison, Paul and Torabian, Parsa and Guarrera, Matteo and Lungren, Matthew P and Chaudhari, Akshay and Brooks, Rupert and Hashir, Mohammad and Bertrand, Hadrien},
booktitle = {Medical Imaging with Deep Learning},
url = {https://github.com/mlmed/torchxrayvision},
arxivId = {2111.00595},
year = {2022}
}
และบทความนี้ซึ่งเริ่มการพัฒนาห้องสมุด: https://arxiv.org/abs/2002.02497
Joseph Paul Cohen and Mohammad Hashir and Rupert Brooks and Hadrien Bertrand
On the limits of cross-domain generalization in automated X-ray prediction.
Medical Imaging with Deep Learning 2020 (Online: https://arxiv.org/abs/2002.02497)
@inproceedings{cohen2020limits,
title={On the limits of cross-domain generalization in automated X-ray prediction},
author={Cohen, Joseph Paul and Hashir, Mohammad and Brooks, Rupert and Bertrand, Hadrien},
booktitle={Medical Imaging with Deep Learning},
year={2020},
url={https://arxiv.org/abs/2002.02497}
}
CIFAR (สถาบันวิจัยขั้นสูงของแคนาดา) | MILA, สถาบันควิเบก AI, มหาวิทยาลัยมอนทรีออล |
|---|---|
ศูนย์ของมหาวิทยาลัยสแตนฟอร์ด ปัญญาประดิษฐ์ด้านการแพทย์และการถ่ายภาพ | สุขภาพของ Carestream |