지금 종이 온라인! https://arxiv.org/abs/2111.00595
지금 문서화 온라인! https://mlmed.org/torchxrayvision/
| (? 프로모션 비디오) )) |
|---|
흉부 X- 레이 데이터 세트 및 모델을위한 라이브러리. 미리 훈련 된 모델을 포함하여.
TorchxRayvision은 흉부 X- 레이 데이터 세트 및 딥 러닝 모델로 작업하기위한 오픈 소스 소프트웨어 라이브러리입니다. 공개적으로 사용 가능한 흉부 X- 레이 데이터 세트를위한 공통 인터페이스 및 공통 사전 처리 체인을 제공합니다. 또한 다양한 데이터 조합에 대해 교육을받은 다양한 아키텍처를 갖춘 여러 분류 및 표현 학습 모델을 라이브러리를 통해 기준선 또는 기능 추출기 역할을 수행 할 수 있습니다.
트위터 : @TorchxRayVision
$ pip install torchxrayvision
import torchxrayvision as xrv
import skimage , torch , torchvision
# Prepare the image:
img = skimage . io . imread ( "16747_3_1.jpg" )
img = xrv . datasets . normalize ( img , 255 ) # convert 8-bit image to [-1024, 1024] range
img = img . mean ( 2 )[ None , ...] # Make single color channel
transform = torchvision . transforms . Compose ([ xrv . datasets . XRayCenterCrop (), xrv . datasets . XRayResizer ( 224 )])
img = transform ( img )
img = torch . from_numpy ( img )
# Load model and process image
model = xrv . models . DenseNet ( weights = "densenet121-res224-all" )
outputs = model ( img [ None ,...]) # or model.features(img[None,...])
# Print results
dict ( zip ( model . pathologies , outputs [ 0 ]. detach (). numpy ()))
{ 'Atelectasis' : 0.32797316 ,
'Consolidation' : 0.42933336 ,
'Infiltration' : 0.5316924 ,
'Pneumothorax' : 0.28849724 ,
'Edema' : 0.024142697 ,
'Emphysema' : 0.5011832 ,
'Fibrosis' : 0.51887786 ,
'Effusion' : 0.27805611 ,
'Pneumonia' : 0.18569896 ,
'Pleural_Thickening' : 0.24489835 ,
'Cardiomegaly' : 0.3645515 ,
'Nodule' : 0.68982 ,
'Mass' : 0.6392845 ,
'Hernia' : 0.00993878 ,
'Lung Lesion' : 0.011150705 ,
'Fracture' : 0.51916164 ,
'Lung Opacity' : 0.59073937 ,
'Enlarged Cardiomediastinum' : 0.27218717 }이미지를 처리하기위한 샘플 스크립트를 사용하여 사전 치료 된 모델은 Process_image.py입니다.
$ python3 process_image.py ../tests/00000001_000.png
{'preds': {'Atelectasis': 0.50500506,
'Cardiomegaly': 0.6600903,
'Consolidation': 0.30575264,
'Edema': 0.274184,
'Effusion': 0.4026162,
'Emphysema': 0.5036339,
'Enlarged Cardiomediastinum': 0.40989172,
'Fibrosis': 0.53293407,
'Fracture': 0.32376793,
'Hernia': 0.011924741,
'Infiltration': 0.5154413,
'Lung Lesion': 0.22231922,
'Lung Opacity': 0.2772148,
'Mass': 0.32237658,
'Nodule': 0.5091847,
'Pleural_Thickening': 0.5102617,
'Pneumonia': 0.30947986,
'Pneumothorax': 0.24847917}}
사전에 사전 된 모델에 대한 가중치를 지정하십시오 (현재 모든 DensenET121) 참고 : 각 사방 모델에는 18 개의 출력이 있습니다. all 모델에는 모든 출력이 훈련되었습니다. 그러나 다른 가중치의 경우 일부 대상은 훈련되지 않으며 교육 데이터 세트에 존재하지 않기 때문에 무작위로 예측할 것입니다. 유효한 유효한 출력은 가중치에 해당하는 데이터 세트의 필드 {dataset}.pathologies 에 나열되어 있습니다.
## 224x224 models
model = xrv . models . DenseNet ( weights = "densenet121-res224-all" )
model = xrv . models . DenseNet ( weights = "densenet121-res224-rsna" ) # RSNA Pneumonia Challenge
model = xrv . models . DenseNet ( weights = "densenet121-res224-nih" ) # NIH chest X-ray8
model = xrv . models . DenseNet ( weights = "densenet121-res224-pc" ) # PadChest (University of Alicante)
model = xrv . models . DenseNet ( weights = "densenet121-res224-chex" ) # CheXpert (Stanford)
model = xrv . models . DenseNet ( weights = "densenet121-res224-mimic_nb" ) # MIMIC-CXR (MIT)
model = xrv . models . DenseNet ( weights = "densenet121-res224-mimic_ch" ) # MIMIC-CXR (MIT)
# 512x512 models
model = xrv . models . ResNet ( weights = "resnet50-res512-all" )
# DenseNet121 from JF Healthcare for the CheXpert competition
model = xrv . baseline_models . jfhealthcare . DenseNet ()
# Official Stanford CheXpert model
model = xrv . baseline_models . chexpert . DenseNet ( weights_zip = "chexpert_weights.zip" )
# Emory HITI lab race prediction model
model = xrv . baseline_models . emory_hiti . RaceModel ()
model . targets - > [ "Asian" , "Black" , "White" ]
# Riken age prediction model
model = xrv . baseline_models . riken . AgeModel ()모드의 벤치 마크는 다음과 같습니다. Benchmarks.MD 및 일부 모델의 성능은이 논문에서 볼 수 있습니다. arxiv.org/abs/2002.02497.
또한 Padchest, NIH, Chexpert 및 Mimic 데이터 세트에서 교육을받은 미리 훈련 된 자동 인코더를로드 할 수도 있습니다.
ae = xrv . autoencoders . ResNetAE ( weights = "101-elastic" )
z = ae . encode ( image )
image2 = ae . decode ( z )사전에 예방 된 해부학 적 세분화 모델을로드 할 수 있습니다. 데모 노트북
seg_model = xrv . baseline_models . chestx_det . PSPNet ()
output = seg_model ( image )
output . shape # [1, 14, 512, 512]
seg_model . targets # ['Left Clavicle', 'Right Clavicle', 'Left Scapula', 'Right Scapula',
# 'Left Lung', 'Right Lung', 'Left Hilus Pulmonis', 'Right Hilus Pulmonis',
# 'Heart', 'Aorta', 'Facies Diaphragmatica', 'Mediastinum', 'Weasand', 'Spine'] 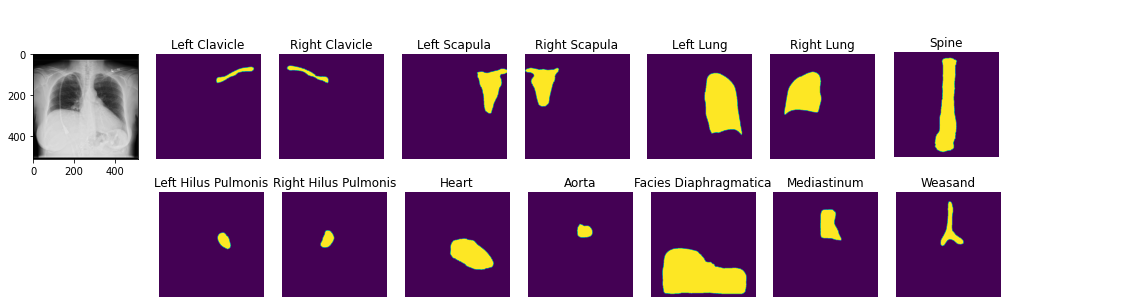
각 데이터 세트 및 데모 노트북 및 예제로드 스크립트에 대한 자세한 내용은 DocStrings보기
transform = torchvision . transforms . Compose ([ xrv . datasets . XRayCenterCrop (),
xrv . datasets . XRayResizer ( 224 )])
# RSNA Pneumonia Detection Challenge. https://pubs.rsna.org/doi/full/10.1148/ryai.2019180041
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "path to stage_2_train_images_jpg" ,
transform = transform )
# CheXpert: A Large Chest Radiograph Dataset with Uncertainty Labels and Expert Comparison. https://arxiv.org/abs/1901.07031
d_chex = xrv . datasets . CheX_Dataset ( imgpath = "path to CheXpert-v1.0-small" ,
csvpath = "path to CheXpert-v1.0-small/train.csv" ,
transform = transform )
# National Institutes of Health ChestX-ray8 dataset. https://arxiv.org/abs/1705.02315
d_nih = xrv . datasets . NIH_Dataset ( imgpath = "path to NIH images" )
# A relabelling of a subset of NIH images from: https://pubs.rsna.org/doi/10.1148/radiol.2019191293
d_nih2 = xrv . datasets . NIH_Google_Dataset ( imgpath = "path to NIH images" )
# PadChest: A large chest x-ray image dataset with multi-label annotated reports. https://arxiv.org/abs/1901.07441
d_pc = xrv . datasets . PC_Dataset ( imgpath = "path to image folder" )
# COVID-19 Image Data Collection. https://arxiv.org/abs/2006.11988
d_covid19 = xrv . datasets . COVID19_Dataset () # specify imgpath and csvpath for the dataset
# SIIM Pneumothorax Dataset. https://www.kaggle.com/c/siim-acr-pneumothorax-segmentation
d_siim = xrv . datasets . SIIM_Pneumothorax_Dataset ( imgpath = "dicom-images-train/" ,
csvpath = "train-rle.csv" )
# VinDr-CXR: An open dataset of chest X-rays with radiologist's annotations. https://arxiv.org/abs/2012.15029
d_vin = xrv . datasets . VinBrain_Dataset ( imgpath = ".../train" ,
csvpath = ".../train.csv" )
# National Library of Medicine Tuberculosis Datasets. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4256233/
d_nlmtb = xrv . datasets . NLMTB_Dataset ( imgpath = "path to MontgomerySet or ChinaSet_AllFiles" )각 데이터 세트에는 여러 필드가 포함되어 있습니다. 이 필드는 xrv.datasets.subset_dataset 및 xrv.datasets.merge_dataset이 사용될 때 유지됩니다.
.pathologies 분야는이 데이터 세트에 포함 된 병리의 목록으로 .labels 필드에 포함될].
.labels 이 필드에는 .pathologies 에 정의 된 각 레이블에 대해 1,0 또는 NAN이 포함되어 있습니다.
.csv 이 필드는 데이터와 함께 제공되는 메타 데이터 CSV 파일의 Pandas 데이터 프레임입니다. 각 행은 데이터 세트의 요소와 정렬되므로 .iloc 사용한 인덱싱이 작동합니다.
가능하면 각 데이터 세트의 .csv 에는 CSV의 공통 필드가 있습니다. 목록이 다음과 같이 정렬됩니다.
csv.patientid 이 데이터 세트에서 샘플을 식별 할 수있는 고유 한 ID
csv.offset_day_int 일일 단위의 이미지에 대한 정수 시간 오프셋. 일부 데이터 세트의 경우 에포크 시간이지만 절대적인 의미는 없습니다.
csv.age_years 몇 년 동안 환자의 나이입니다.
csv.sex_male 환자가 남성 인 경우
csv.sex_female 환자가 여성 인 경우
Relabel_Dataset은 라벨이 병리 논증과 동일한 순서를 갖도록 정렬합니다.
xrv . datasets . relabel_dataset ( xrv . datasets . default_pathologies , d_nih ) # has side effects뷰의 하위 집합 지정 (데모 노트북)
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "..." ,
views = [ "PA" , "AP" , "AP Supine" ])환자 당 1 개의 이미지 만 지정하십시오
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "..." ,
unique_patients = True )데이터 세트 당 요약 통계를 얻습니다
d_chex = xrv . datasets . CheX_Dataset ( imgpath = "CheXpert-v1.0-small" ,
csvpath = "CheXpert-v1.0-small/train.csv" ,
views = [ "PA" , "AP" ], unique_patients = False )
CheX_Dataset num_samples = 191010 views = [ 'PA' , 'AP' ]
{ 'Atelectasis' : { 0.0 : 17621 , 1.0 : 29718 },
'Cardiomegaly' : { 0.0 : 22645 , 1.0 : 23384 },
'Consolidation' : { 0.0 : 30463 , 1.0 : 12982 },
'Edema' : { 0.0 : 29449 , 1.0 : 49674 },
'Effusion' : { 0.0 : 34376 , 1.0 : 76894 },
'Enlarged Cardiomediastinum' : { 0.0 : 26527 , 1.0 : 9186 },
'Fracture' : { 0.0 : 18111 , 1.0 : 7434 },
'Lung Lesion' : { 0.0 : 17523 , 1.0 : 7040 },
'Lung Opacity' : { 0.0 : 20165 , 1.0 : 94207 },
'Pleural Other' : { 0.0 : 17166 , 1.0 : 2503 },
'Pneumonia' : { 0.0 : 18105 , 1.0 : 4674 },
'Pneumothorax' : { 0.0 : 54165 , 1.0 : 17693 },
'Support Devices' : { 0.0 : 21757 , 1.0 : 99747 }}마스크는 다음 데이터 세트에서 사용할 수 있습니다.
xrv . datasets . RSNA_Pneumonia_Dataset () # for Lung Opacity
xrv . datasets . SIIM_Pneumothorax_Dataset () # for Pneumothorax
xrv . datasets . NIH_Dataset () # for Cardiomegaly, Mass, Effusion, ...예제 사용 :
d_rsna = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "stage_2_train_images_jpg" ,
views = [ "PA" , "AP" ],
pathology_masks = True )
# The has_masks column will let you know if any masks exist for that sample
d_rsna . csv . has_masks . value_counts ()
False 20672
True 6012
# Each sample will have a pathology_masks dictionary where the index
# of each pathology will correspond to a mask of that pathology (if it exists).
# There may be more than one mask per sample. But only one per pathology.
sample [ "pathology_masks" ][ d_rsna . pathologies . index ( "Lung Opacity" )] 또한 data_aug=data_transforms dataloader로 전달하면 data_augmentation과 함께 작동합니다. 임의의 씨앗은 이미지와 마스크에 대한 호출을 정렬하기 위해 일치합니다.
클래스 xrv.datasets.CovariateDataset 레이블을 나타내는 두 개의 데이터 세트와 두 개의 배열을 가져옵니다. 샘플은 각 사이트에서 원하는 이미지의 비율로 반환됩니다. 여기서 목표는 공변량 전환을 시뮬레이션하여 잘못된 기능에 모델을 집중시키는 것입니다. 그런 다음 유효성 검사 데이터에서 변화를 되돌릴 수있어 일반화 성능에서 치명적인 실패가 발생합니다.
비율 = 0.0은 D1의 이미지가 양의 레이블 비율을 가짐을 의미합니다 = 0.5는 D1의 이미지가 양의 레이블 비율의 절반을 가짐을 의미합니다. = 1.0은 D1의 이미지가 양의 레이블이 없음을 의미합니다.
비율이면 반환 된 샘플 수가 동일합니다.
d = xrv . datasets . CovariateDataset ( d1 = # dataset1 with a specific condition
d1_target = #target label to predict,
d2 = # dataset2 with a specific condition
d2_target = #target label to predict,
mode = "train" , # train, valid, and test
ratio = 0.9 )기본 TorchxRayvision 용지 : https://arxiv.org/abs/2111.00595
Joseph Paul Cohen, Joseph D. Viviano, Paul Bertin, Paul Morrison, Parsa Torabian, Matteo Guarrera, Matthew P Lungren, Akshay Chaudhari, Rupert Brooks, Mohammad Hashir, Hadrien Bertrand
TorchXRayVision: A library of chest X-ray datasets and models.
Medical Imaging with Deep Learning
https://github.com/mlmed/torchxrayvision, 2020
@inproceedings{Cohen2022xrv,
title = {{TorchXRayVision: A library of chest X-ray datasets and models}},
author = {Cohen, Joseph Paul and Viviano, Joseph D. and Bertin, Paul and Morrison, Paul and Torabian, Parsa and Guarrera, Matteo and Lungren, Matthew P and Chaudhari, Akshay and Brooks, Rupert and Hashir, Mohammad and Bertrand, Hadrien},
booktitle = {Medical Imaging with Deep Learning},
url = {https://github.com/mlmed/torchxrayvision},
arxivId = {2111.00595},
year = {2022}
}
그리고 도서관의 개발을 시작한이 논문 : https://arxiv.org/abs/2002.02497
Joseph Paul Cohen and Mohammad Hashir and Rupert Brooks and Hadrien Bertrand
On the limits of cross-domain generalization in automated X-ray prediction.
Medical Imaging with Deep Learning 2020 (Online: https://arxiv.org/abs/2002.02497)
@inproceedings{cohen2020limits,
title={On the limits of cross-domain generalization in automated X-ray prediction},
author={Cohen, Joseph Paul and Hashir, Mohammad and Brooks, Rupert and Bertrand, Hadrien},
booktitle={Medical Imaging with Deep Learning},
year={2020},
url={https://arxiv.org/abs/2002.02497}
}
Cifar (캐나다 고급 연구 연구소) | Mila, 몬트리올 대학교 퀘벡 AI 연구소 |
|---|---|
스탠포드 대학교의 센터 의학 및 영상의 인공 지능 | 카 스트림 건강 |