Kertas sekarang online! https://arxiv.org/abs/2111.00595
Dokumentasi sekarang online! https://mlmed.org/torchxrayvision/
| (? Video promo) ) |
|---|
Perpustakaan untuk dataset dan model x-ray dada. Termasuk model pra-terlatih.
TorchxRayVision adalah pustaka perangkat lunak open source untuk bekerja dengan dataset x-ray dada dan model pembelajaran yang mendalam. Ini menyediakan antarmuka yang umum dan rantai pra-pemrosesan umum untuk serangkaian dataset x-ray dada yang tersedia untuk umum. Selain itu, sejumlah model pembelajaran klasifikasi dan representasi dengan arsitektur yang berbeda, dilatih pada kombinasi data yang berbeda, tersedia melalui perpustakaan untuk berfungsi sebagai garis dasar atau ekstraktor fitur.
Twitter: @TorChxRayVision
$ pip install torchxrayvision
import torchxrayvision as xrv
import skimage , torch , torchvision
# Prepare the image:
img = skimage . io . imread ( "16747_3_1.jpg" )
img = xrv . datasets . normalize ( img , 255 ) # convert 8-bit image to [-1024, 1024] range
img = img . mean ( 2 )[ None , ...] # Make single color channel
transform = torchvision . transforms . Compose ([ xrv . datasets . XRayCenterCrop (), xrv . datasets . XRayResizer ( 224 )])
img = transform ( img )
img = torch . from_numpy ( img )
# Load model and process image
model = xrv . models . DenseNet ( weights = "densenet121-res224-all" )
outputs = model ( img [ None ,...]) # or model.features(img[None,...])
# Print results
dict ( zip ( model . pathologies , outputs [ 0 ]. detach (). numpy ()))
{ 'Atelectasis' : 0.32797316 ,
'Consolidation' : 0.42933336 ,
'Infiltration' : 0.5316924 ,
'Pneumothorax' : 0.28849724 ,
'Edema' : 0.024142697 ,
'Emphysema' : 0.5011832 ,
'Fibrosis' : 0.51887786 ,
'Effusion' : 0.27805611 ,
'Pneumonia' : 0.18569896 ,
'Pleural_Thickening' : 0.24489835 ,
'Cardiomegaly' : 0.3645515 ,
'Nodule' : 0.68982 ,
'Mass' : 0.6392845 ,
'Hernia' : 0.00993878 ,
'Lung Lesion' : 0.011150705 ,
'Fracture' : 0.51916164 ,
'Lung Opacity' : 0.59073937 ,
'Enlarged Cardiomediastinum' : 0.27218717 }Skrip sampel untuk memproses gambar menggunakan model pretrained adalah process_image.py
$ python3 process_image.py ../tests/00000001_000.png
{'preds': {'Atelectasis': 0.50500506,
'Cardiomegaly': 0.6600903,
'Consolidation': 0.30575264,
'Edema': 0.274184,
'Effusion': 0.4026162,
'Emphysema': 0.5036339,
'Enlarged Cardiomediastinum': 0.40989172,
'Fibrosis': 0.53293407,
'Fracture': 0.32376793,
'Hernia': 0.011924741,
'Infiltration': 0.5154413,
'Lung Lesion': 0.22231922,
'Lung Opacity': 0.2772148,
'Mass': 0.32237658,
'Nodule': 0.5091847,
'Pleural_Thickening': 0.5102617,
'Pneumonia': 0.30947986,
'Pneumothorax': 0.24847917}}
Tentukan bobot untuk model pretrained (saat ini semua densenet121) Catatan: Setiap model pretrain memiliki 18 output. all model memiliki setiap keluaran yang dilatih. Namun, untuk bobot lain beberapa target tidak dilatih dan akan memprediksi secara acak karena mereka tidak ada dalam dataset pelatihan. Satu -satunya output yang valid tercantum di bidang {dataset}.pathologies pada dataset yang sesuai dengan bobot.
## 224x224 models
model = xrv . models . DenseNet ( weights = "densenet121-res224-all" )
model = xrv . models . DenseNet ( weights = "densenet121-res224-rsna" ) # RSNA Pneumonia Challenge
model = xrv . models . DenseNet ( weights = "densenet121-res224-nih" ) # NIH chest X-ray8
model = xrv . models . DenseNet ( weights = "densenet121-res224-pc" ) # PadChest (University of Alicante)
model = xrv . models . DenseNet ( weights = "densenet121-res224-chex" ) # CheXpert (Stanford)
model = xrv . models . DenseNet ( weights = "densenet121-res224-mimic_nb" ) # MIMIC-CXR (MIT)
model = xrv . models . DenseNet ( weights = "densenet121-res224-mimic_ch" ) # MIMIC-CXR (MIT)
# 512x512 models
model = xrv . models . ResNet ( weights = "resnet50-res512-all" )
# DenseNet121 from JF Healthcare for the CheXpert competition
model = xrv . baseline_models . jfhealthcare . DenseNet ()
# Official Stanford CheXpert model
model = xrv . baseline_models . chexpert . DenseNet ( weights_zip = "chexpert_weights.zip" )
# Emory HITI lab race prediction model
model = xrv . baseline_models . emory_hiti . RaceModel ()
model . targets - > [ "Asian" , "Black" , "White" ]
# Riken age prediction model
model = xrv . baseline_models . riken . AgeModel ()Tolok ukur mode ada di sini: tolok ukur.md dan kinerja beberapa model dapat dilihat dalam makalah ini arxiv.org/abs/2002.02497.
Anda juga dapat memuat autoencoder pra-terlatih yang dilatih pada dataset Padchest, NIH, Chexpert, dan Mimic.
ae = xrv . autoencoders . ResNetAE ( weights = "101-elastic" )
z = ae . encode ( image )
image2 = ae . decode ( z )Anda dapat memuat model segmentasi anatomi pretrain. Demo Notebook
seg_model = xrv . baseline_models . chestx_det . PSPNet ()
output = seg_model ( image )
output . shape # [1, 14, 512, 512]
seg_model . targets # ['Left Clavicle', 'Right Clavicle', 'Left Scapula', 'Right Scapula',
# 'Left Lung', 'Right Lung', 'Left Hilus Pulmonis', 'Right Hilus Pulmonis',
# 'Heart', 'Aorta', 'Facies Diaphragmatica', 'Mediastinum', 'Weasand', 'Spine'] 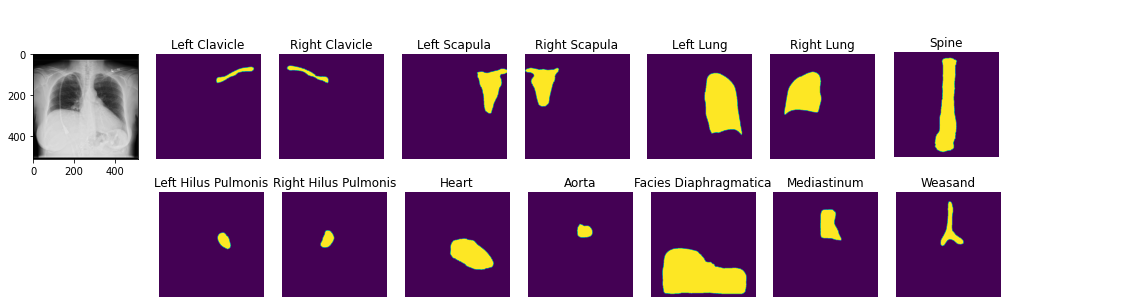
Lihat Docstrings Untuk detail lebih lanjut pada setiap dataset dan notebook demo dan contoh memuat skrip
transform = torchvision . transforms . Compose ([ xrv . datasets . XRayCenterCrop (),
xrv . datasets . XRayResizer ( 224 )])
# RSNA Pneumonia Detection Challenge. https://pubs.rsna.org/doi/full/10.1148/ryai.2019180041
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "path to stage_2_train_images_jpg" ,
transform = transform )
# CheXpert: A Large Chest Radiograph Dataset with Uncertainty Labels and Expert Comparison. https://arxiv.org/abs/1901.07031
d_chex = xrv . datasets . CheX_Dataset ( imgpath = "path to CheXpert-v1.0-small" ,
csvpath = "path to CheXpert-v1.0-small/train.csv" ,
transform = transform )
# National Institutes of Health ChestX-ray8 dataset. https://arxiv.org/abs/1705.02315
d_nih = xrv . datasets . NIH_Dataset ( imgpath = "path to NIH images" )
# A relabelling of a subset of NIH images from: https://pubs.rsna.org/doi/10.1148/radiol.2019191293
d_nih2 = xrv . datasets . NIH_Google_Dataset ( imgpath = "path to NIH images" )
# PadChest: A large chest x-ray image dataset with multi-label annotated reports. https://arxiv.org/abs/1901.07441
d_pc = xrv . datasets . PC_Dataset ( imgpath = "path to image folder" )
# COVID-19 Image Data Collection. https://arxiv.org/abs/2006.11988
d_covid19 = xrv . datasets . COVID19_Dataset () # specify imgpath and csvpath for the dataset
# SIIM Pneumothorax Dataset. https://www.kaggle.com/c/siim-acr-pneumothorax-segmentation
d_siim = xrv . datasets . SIIM_Pneumothorax_Dataset ( imgpath = "dicom-images-train/" ,
csvpath = "train-rle.csv" )
# VinDr-CXR: An open dataset of chest X-rays with radiologist's annotations. https://arxiv.org/abs/2012.15029
d_vin = xrv . datasets . VinBrain_Dataset ( imgpath = ".../train" ,
csvpath = ".../train.csv" )
# National Library of Medicine Tuberculosis Datasets. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4256233/
d_nlmtb = xrv . datasets . NLMTB_Dataset ( imgpath = "path to MontgomerySet or ChinaSet_AllFiles" )Setiap dataset berisi sejumlah bidang. Bidang -bidang ini dipertahankan ketika xrv.datasets.subset_dataset dan xrv.datasets.merge_dataset digunakan.
.pathologies Bidang ini adalah daftar patologi yang terkandung dalam dataset ini yang akan terkandung dalam bidang .labels ].
.labels bidang ini berisi 1,0, atau NAN untuk setiap label yang ditentukan dalam .pathologies .
.csv Bidang ini adalah basis data panda dari file metadata CSV yang disertakan dengan data. Setiap baris selaras dengan elemen dataset sehingga pengindeksan menggunakan .iloc akan berfungsi.
Jika memungkinkan, setiap dataset .csv akan memiliki beberapa bidang umum CSV. Ini akan disejajarkan saat daftar adalah sebagai berikut:
csv.patientid ID unik yang secara unikely mengidentifikasi sampel dalam dataset ini
csv.offset_day_int Offset waktu integer untuk gambar dalam satuan hari. Ini diharapkan untuk waktu relatif dan tidak memiliki makna absolut meskipun untuk beberapa dataset itu adalah waktu zaman.
csv.age_years Usia pasien selama bertahun -tahun.
csv.sex_male jika pasien laki -laki
csv.sex_female jika pasien adalah perempuan
Relabel_dataset akan menyelaraskan label untuk memiliki urutan yang sama dengan argumen patologi.
xrv . datasets . relabel_dataset ( xrv . datasets . default_pathologies , d_nih ) # has side effectsTentukan subset tampilan (Demo Notebook)
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "..." ,
views = [ "PA" , "AP" , "AP Supine" ])Tentukan hanya 1 gambar per pasien
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "..." ,
unique_patients = True )Dapatkan ringkasan statistik per dataset
d_chex = xrv . datasets . CheX_Dataset ( imgpath = "CheXpert-v1.0-small" ,
csvpath = "CheXpert-v1.0-small/train.csv" ,
views = [ "PA" , "AP" ], unique_patients = False )
CheX_Dataset num_samples = 191010 views = [ 'PA' , 'AP' ]
{ 'Atelectasis' : { 0.0 : 17621 , 1.0 : 29718 },
'Cardiomegaly' : { 0.0 : 22645 , 1.0 : 23384 },
'Consolidation' : { 0.0 : 30463 , 1.0 : 12982 },
'Edema' : { 0.0 : 29449 , 1.0 : 49674 },
'Effusion' : { 0.0 : 34376 , 1.0 : 76894 },
'Enlarged Cardiomediastinum' : { 0.0 : 26527 , 1.0 : 9186 },
'Fracture' : { 0.0 : 18111 , 1.0 : 7434 },
'Lung Lesion' : { 0.0 : 17523 , 1.0 : 7040 },
'Lung Opacity' : { 0.0 : 20165 , 1.0 : 94207 },
'Pleural Other' : { 0.0 : 17166 , 1.0 : 2503 },
'Pneumonia' : { 0.0 : 18105 , 1.0 : 4674 },
'Pneumothorax' : { 0.0 : 54165 , 1.0 : 17693 },
'Support Devices' : { 0.0 : 21757 , 1.0 : 99747 }}Topeng tersedia dalam set data berikut:
xrv . datasets . RSNA_Pneumonia_Dataset () # for Lung Opacity
xrv . datasets . SIIM_Pneumothorax_Dataset () # for Pneumothorax
xrv . datasets . NIH_Dataset () # for Cardiomegaly, Mass, Effusion, ...Contoh Penggunaan:
d_rsna = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "stage_2_train_images_jpg" ,
views = [ "PA" , "AP" ],
pathology_masks = True )
# The has_masks column will let you know if any masks exist for that sample
d_rsna . csv . has_masks . value_counts ()
False 20672
True 6012
# Each sample will have a pathology_masks dictionary where the index
# of each pathology will correspond to a mask of that pathology (if it exists).
# There may be more than one mask per sample. But only one per pathology.
sample [ "pathology_masks" ][ d_rsna . pathologies . index ( "Lung Opacity" )] Ini juga berfungsi dengan data_augmentation jika Anda masuk dalam data_aug=data_transforms ke dataloader. Benih acak dicocokkan untuk menyelaraskan panggilan untuk gambar dan topeng.
Kelas xrv.datasets.CovariateDataset mengambil dua kumpulan data dan dua array yang mewakili label. Sampel akan dikembalikan dengan rasio gambar yang diinginkan dari setiap situs. Tujuannya di sini adalah untuk mensimulasikan pergeseran kovariat untuk membuat model fokus pada fitur yang salah. Maka pergeseran dapat dibalik dalam data validasi yang menyebabkan kegagalan bencana dalam kinerja generalisasi.
Rasio = 0,0 berarti gambar dari D1 akan memiliki rasio label positif = 0,5 berarti gambar dari D1 akan memiliki setengah dari rasio label positif = 1.0 berarti gambar dari D1 tidak akan memiliki label positif
Dengan rasio apa pun, jumlah sampel yang dikembalikan akan sama.
d = xrv . datasets . CovariateDataset ( d1 = # dataset1 with a specific condition
d1_target = #target label to predict,
d2 = # dataset2 with a specific condition
d2_target = #target label to predict,
mode = "train" , # train, valid, and test
ratio = 0.9 )Kertas torchxrayvision utama: https://arxiv.org/abs/2111.00595
Joseph Paul Cohen, Joseph D. Viviano, Paul Bertin, Paul Morrison, Parsa Torabian, Matteo Guarrera, Matthew P Lungren, Akshay Chaudhari, Rupert Brooks, Mohammad Hashir, Hadrien Bertrand
TorchXRayVision: A library of chest X-ray datasets and models.
Medical Imaging with Deep Learning
https://github.com/mlmed/torchxrayvision, 2020
@inproceedings{Cohen2022xrv,
title = {{TorchXRayVision: A library of chest X-ray datasets and models}},
author = {Cohen, Joseph Paul and Viviano, Joseph D. and Bertin, Paul and Morrison, Paul and Torabian, Parsa and Guarrera, Matteo and Lungren, Matthew P and Chaudhari, Akshay and Brooks, Rupert and Hashir, Mohammad and Bertrand, Hadrien},
booktitle = {Medical Imaging with Deep Learning},
url = {https://github.com/mlmed/torchxrayvision},
arxivId = {2111.00595},
year = {2022}
}
dan makalah ini yang memulai pengembangan perpustakaan: https://arxiv.org/abs/2002.02497
Joseph Paul Cohen and Mohammad Hashir and Rupert Brooks and Hadrien Bertrand
On the limits of cross-domain generalization in automated X-ray prediction.
Medical Imaging with Deep Learning 2020 (Online: https://arxiv.org/abs/2002.02497)
@inproceedings{cohen2020limits,
title={On the limits of cross-domain generalization in automated X-ray prediction},
author={Cohen, Joseph Paul and Hashir, Mohammad and Brooks, Rupert and Bertrand, Hadrien},
booktitle={Medical Imaging with Deep Learning},
year={2020},
url={https://arxiv.org/abs/2002.02497}
}
CIFAR (Institut Kanada untuk Penelitian Lanjutan) | Mila, Institut AI Quebec, Universitas Montreal |
|---|---|
Pusat Universitas Stanford Kecerdasan Buatan dalam Kedokteran & Pencitraan | Kesehatan Carestream |