ورقة الآن على الإنترنت! https://arxiv.org/abs/2111.00595
الوثائق الآن على الإنترنت! https://mlmed.org/torchxrayvision/
| (؟ فيديو ترويجي) ) |
|---|
مكتبة لمجموعات البيانات والموديلات بالأشعة السينية الصدر. بما في ذلك النماذج التي تم تدريبها مسبقًا.
TorchxRayVision هي مكتبة برمجيات مفتوحة المصدر للعمل مع مجموعات بيانات الأشعة السينية للصدر ونماذج التعلم العميق. يوفر واجهة مشتركة وسلسلة معالجة مسبقة مشتركة لمجموعة واسعة من مجموعات بيانات الأشعة السينية المتاحة للجمهور. بالإضافة إلى ذلك ، يتوفر عدد من نماذج التعلم في التصنيف والتمثيل مع بنيات مختلفة ، مدربة على مجموعات بيانات مختلفة ، من خلال المكتبة لتكون بمثابة خطوط الأساس أو مستخلصات الميزات.
Twitter: torchxrayvision
$ pip install torchxrayvision
import torchxrayvision as xrv
import skimage , torch , torchvision
# Prepare the image:
img = skimage . io . imread ( "16747_3_1.jpg" )
img = xrv . datasets . normalize ( img , 255 ) # convert 8-bit image to [-1024, 1024] range
img = img . mean ( 2 )[ None , ...] # Make single color channel
transform = torchvision . transforms . Compose ([ xrv . datasets . XRayCenterCrop (), xrv . datasets . XRayResizer ( 224 )])
img = transform ( img )
img = torch . from_numpy ( img )
# Load model and process image
model = xrv . models . DenseNet ( weights = "densenet121-res224-all" )
outputs = model ( img [ None ,...]) # or model.features(img[None,...])
# Print results
dict ( zip ( model . pathologies , outputs [ 0 ]. detach (). numpy ()))
{ 'Atelectasis' : 0.32797316 ,
'Consolidation' : 0.42933336 ,
'Infiltration' : 0.5316924 ,
'Pneumothorax' : 0.28849724 ,
'Edema' : 0.024142697 ,
'Emphysema' : 0.5011832 ,
'Fibrosis' : 0.51887786 ,
'Effusion' : 0.27805611 ,
'Pneumonia' : 0.18569896 ,
'Pleural_Thickening' : 0.24489835 ,
'Cardiomegaly' : 0.3645515 ,
'Nodule' : 0.68982 ,
'Mass' : 0.6392845 ,
'Hernia' : 0.00993878 ,
'Lung Lesion' : 0.011150705 ,
'Fracture' : 0.51916164 ,
'Lung Opacity' : 0.59073937 ,
'Enlarged Cardiomediastinum' : 0.27218717 }نموذج نصي لمعالجة الصور النماذج المسبقة هي process_image.py
$ python3 process_image.py ../tests/00000001_000.png
{'preds': {'Atelectasis': 0.50500506,
'Cardiomegaly': 0.6600903,
'Consolidation': 0.30575264,
'Edema': 0.274184,
'Effusion': 0.4026162,
'Emphysema': 0.5036339,
'Enlarged Cardiomediastinum': 0.40989172,
'Fibrosis': 0.53293407,
'Fracture': 0.32376793,
'Hernia': 0.011924741,
'Infiltration': 0.5154413,
'Lung Lesion': 0.22231922,
'Lung Opacity': 0.2772148,
'Mass': 0.32237658,
'Nodule': 0.5091847,
'Pleural_Thickening': 0.5102617,
'Pneumonia': 0.30947986,
'Pneumothorax': 0.24847917}}
حدد أوزان النماذج المسبقة (حاليًا جميع Densenet121) ملاحظة: يحتوي كل طراز PretRained على 18 مخرجًا. all النموذج لديه كل الإخراج مدرب. ومع ذلك ، بالنسبة للأوزان الأخرى ، لا يتم تدريب بعض الأهداف وسوف تتنبأ بشكل عشوائي لأنها غير موجودة في مجموعة بيانات التدريب. يتم سرد المخرجات الصالحة الوحيدة في الحقل {dataset}.pathologies على مجموعة البيانات التي تتوافق مع الأوزان.
## 224x224 models
model = xrv . models . DenseNet ( weights = "densenet121-res224-all" )
model = xrv . models . DenseNet ( weights = "densenet121-res224-rsna" ) # RSNA Pneumonia Challenge
model = xrv . models . DenseNet ( weights = "densenet121-res224-nih" ) # NIH chest X-ray8
model = xrv . models . DenseNet ( weights = "densenet121-res224-pc" ) # PadChest (University of Alicante)
model = xrv . models . DenseNet ( weights = "densenet121-res224-chex" ) # CheXpert (Stanford)
model = xrv . models . DenseNet ( weights = "densenet121-res224-mimic_nb" ) # MIMIC-CXR (MIT)
model = xrv . models . DenseNet ( weights = "densenet121-res224-mimic_ch" ) # MIMIC-CXR (MIT)
# 512x512 models
model = xrv . models . ResNet ( weights = "resnet50-res512-all" )
# DenseNet121 from JF Healthcare for the CheXpert competition
model = xrv . baseline_models . jfhealthcare . DenseNet ()
# Official Stanford CheXpert model
model = xrv . baseline_models . chexpert . DenseNet ( weights_zip = "chexpert_weights.zip" )
# Emory HITI lab race prediction model
model = xrv . baseline_models . emory_hiti . RaceModel ()
model . targets - > [ "Asian" , "Black" , "White" ]
# Riken age prediction model
model = xrv . baseline_models . riken . AgeModel ()معايير الأوضاع هنا: المعايير.
يمكنك أيضًا تحميل مشفر تلقائي تم تدريبه مسبقًا يتم تدريبه على مجموعات بيانات Padchest و NIH و Chexpert و Rimic.
ae = xrv . autoencoders . ResNetAE ( weights = "101-elastic" )
z = ae . encode ( image )
image2 = ae . decode ( z )يمكنك تحميل نماذج تجزئة تشريحية قبل. دفتر ملاحظات تجريبي
seg_model = xrv . baseline_models . chestx_det . PSPNet ()
output = seg_model ( image )
output . shape # [1, 14, 512, 512]
seg_model . targets # ['Left Clavicle', 'Right Clavicle', 'Left Scapula', 'Right Scapula',
# 'Left Lung', 'Right Lung', 'Left Hilus Pulmonis', 'Right Hilus Pulmonis',
# 'Heart', 'Aorta', 'Facies Diaphragmatica', 'Mediastinum', 'Weasand', 'Spine'] 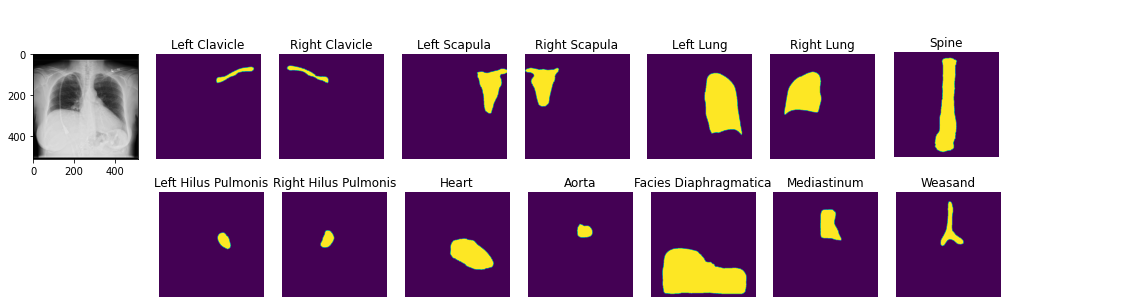
عرض docstrings لمزيد من التفاصيل في كل مجموعة بيانات ودفتر تجريبي ومثال على تحميل البرنامج النصي
transform = torchvision . transforms . Compose ([ xrv . datasets . XRayCenterCrop (),
xrv . datasets . XRayResizer ( 224 )])
# RSNA Pneumonia Detection Challenge. https://pubs.rsna.org/doi/full/10.1148/ryai.2019180041
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "path to stage_2_train_images_jpg" ,
transform = transform )
# CheXpert: A Large Chest Radiograph Dataset with Uncertainty Labels and Expert Comparison. https://arxiv.org/abs/1901.07031
d_chex = xrv . datasets . CheX_Dataset ( imgpath = "path to CheXpert-v1.0-small" ,
csvpath = "path to CheXpert-v1.0-small/train.csv" ,
transform = transform )
# National Institutes of Health ChestX-ray8 dataset. https://arxiv.org/abs/1705.02315
d_nih = xrv . datasets . NIH_Dataset ( imgpath = "path to NIH images" )
# A relabelling of a subset of NIH images from: https://pubs.rsna.org/doi/10.1148/radiol.2019191293
d_nih2 = xrv . datasets . NIH_Google_Dataset ( imgpath = "path to NIH images" )
# PadChest: A large chest x-ray image dataset with multi-label annotated reports. https://arxiv.org/abs/1901.07441
d_pc = xrv . datasets . PC_Dataset ( imgpath = "path to image folder" )
# COVID-19 Image Data Collection. https://arxiv.org/abs/2006.11988
d_covid19 = xrv . datasets . COVID19_Dataset () # specify imgpath and csvpath for the dataset
# SIIM Pneumothorax Dataset. https://www.kaggle.com/c/siim-acr-pneumothorax-segmentation
d_siim = xrv . datasets . SIIM_Pneumothorax_Dataset ( imgpath = "dicom-images-train/" ,
csvpath = "train-rle.csv" )
# VinDr-CXR: An open dataset of chest X-rays with radiologist's annotations. https://arxiv.org/abs/2012.15029
d_vin = xrv . datasets . VinBrain_Dataset ( imgpath = ".../train" ,
csvpath = ".../train.csv" )
# National Library of Medicine Tuberculosis Datasets. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4256233/
d_nlmtb = xrv . datasets . NLMTB_Dataset ( imgpath = "path to MontgomerySet or ChinaSet_AllFiles" )تحتوي كل مجموعة بيانات على عدد من الحقول. يتم الحفاظ على هذه الحقول عند استخدام XRV.Datasets.Subset_Dataset و XRV.Datasets.merge_dataset.
.pathologies .labels
.labels يحتوي هذا الحقل على 1،0 ، أو نان لكل علامة محددة في .pathologies
.csv هذا الحقل هو نظام بيانات Pandas لملف CSV البيانات الوصفية التي تأتي مع البيانات. يتماشى كل صف مع عناصر مجموعة البيانات ، وبالتالي فإن الفهرسة باستخدام .iloc ستعمل.
إذا كان ذلك ممكنًا ، فسيكون لكل مجموعة بيانات .csv بعض الحقول المشتركة من CSV. سيتم محاذاة هذه عندما تكون القائمة كما يلي:
csv.patientid معرف فريد من نوعه من شأنه تحديد العينات في مجموعة البيانات هذه
csv.offset_day_int إزاحة وقت عدد صحيح للصورة في وحدة الأيام. من المتوقع أن يكون هذا في الأوقات النسبية وليس له معنى مطلق على الرغم من أنه بالنسبة لبعض مجموعات البيانات ، فهو وقت الحقبة.
csv.age_years عصر المريض منذ سنوات.
csv.sex_male إذا كان المريض ذكر
csv.sex_female إذا كان المريض أنثى
سوف Relabel_dataset محاذاة الملصقات للحصول على نفس الترتيب مثل وسيطة الأمراض.
xrv . datasets . relabel_dataset ( xrv . datasets . default_pathologies , d_nih ) # has side effectsحدد مجموعة فرعية من المشاهدات (دفتر ملاحظات تجريبي)
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "..." ,
views = [ "PA" , "AP" , "AP Supine" ])حدد صورة واحدة فقط لكل مريض
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "..." ,
unique_patients = True )الحصول على إحصاءات موجزة لكل مجموعة بيانات
d_chex = xrv . datasets . CheX_Dataset ( imgpath = "CheXpert-v1.0-small" ,
csvpath = "CheXpert-v1.0-small/train.csv" ,
views = [ "PA" , "AP" ], unique_patients = False )
CheX_Dataset num_samples = 191010 views = [ 'PA' , 'AP' ]
{ 'Atelectasis' : { 0.0 : 17621 , 1.0 : 29718 },
'Cardiomegaly' : { 0.0 : 22645 , 1.0 : 23384 },
'Consolidation' : { 0.0 : 30463 , 1.0 : 12982 },
'Edema' : { 0.0 : 29449 , 1.0 : 49674 },
'Effusion' : { 0.0 : 34376 , 1.0 : 76894 },
'Enlarged Cardiomediastinum' : { 0.0 : 26527 , 1.0 : 9186 },
'Fracture' : { 0.0 : 18111 , 1.0 : 7434 },
'Lung Lesion' : { 0.0 : 17523 , 1.0 : 7040 },
'Lung Opacity' : { 0.0 : 20165 , 1.0 : 94207 },
'Pleural Other' : { 0.0 : 17166 , 1.0 : 2503 },
'Pneumonia' : { 0.0 : 18105 , 1.0 : 4674 },
'Pneumothorax' : { 0.0 : 54165 , 1.0 : 17693 },
'Support Devices' : { 0.0 : 21757 , 1.0 : 99747 }}الأقنعة متوفرة في مجموعات البيانات التالية:
xrv . datasets . RSNA_Pneumonia_Dataset () # for Lung Opacity
xrv . datasets . SIIM_Pneumothorax_Dataset () # for Pneumothorax
xrv . datasets . NIH_Dataset () # for Cardiomegaly, Mass, Effusion, ...مثال الاستخدام:
d_rsna = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "stage_2_train_images_jpg" ,
views = [ "PA" , "AP" ],
pathology_masks = True )
# The has_masks column will let you know if any masks exist for that sample
d_rsna . csv . has_masks . value_counts ()
False 20672
True 6012
# Each sample will have a pathology_masks dictionary where the index
# of each pathology will correspond to a mask of that pathology (if it exists).
# There may be more than one mask per sample. But only one per pathology.
sample [ "pathology_masks" ][ d_rsna . pathologies . index ( "Lung Opacity" )] يعمل أيضًا مع Data_augmentation إذا قمت بتمرير data_aug=data_transforms إلى dataloader. تتم مطابقة البذور العشوائية لمحاذاة مكالمات الصورة والقناع.
يأخذ Class xrv.datasets.CovariateDataset مجموعتين ومصفوفتين يمثلان الملصقات. سيتم إرجاع العينات مع النسبة المطلوبة للصور من كل موقع. الهدف هنا هو محاكاة تحول المتغير لجعل النموذج يركز على ميزة غير صحيحة. ثم يمكن عكس التحول في بيانات التحقق مما تسبب في فشل كارثي في أداء التعميم.
النسبة = 0.0 تعني الصور من D1 سيكون لها نسبة تسمية موجبة = 0.5 تعني الصور من D1 سيكون لها نصف نسبة التسميات الإيجابية = 1.0 يعني أن الصور من D1 لن يكون لها علامة إيجابية
مع أي نسبة ، سيكون عدد العينات التي تم إرجاعها هو نفسه.
d = xrv . datasets . CovariateDataset ( d1 = # dataset1 with a specific condition
d1_target = #target label to predict,
d2 = # dataset2 with a specific condition
d2_target = #target label to predict,
mode = "train" , # train, valid, and test
ratio = 0.9 )ورقة torchxrayvision الأولية: https://arxiv.org/abs/2111.00595
Joseph Paul Cohen, Joseph D. Viviano, Paul Bertin, Paul Morrison, Parsa Torabian, Matteo Guarrera, Matthew P Lungren, Akshay Chaudhari, Rupert Brooks, Mohammad Hashir, Hadrien Bertrand
TorchXRayVision: A library of chest X-ray datasets and models.
Medical Imaging with Deep Learning
https://github.com/mlmed/torchxrayvision, 2020
@inproceedings{Cohen2022xrv,
title = {{TorchXRayVision: A library of chest X-ray datasets and models}},
author = {Cohen, Joseph Paul and Viviano, Joseph D. and Bertin, Paul and Morrison, Paul and Torabian, Parsa and Guarrera, Matteo and Lungren, Matthew P and Chaudhari, Akshay and Brooks, Rupert and Hashir, Mohammad and Bertrand, Hadrien},
booktitle = {Medical Imaging with Deep Learning},
url = {https://github.com/mlmed/torchxrayvision},
arxivId = {2111.00595},
year = {2022}
}
وهذه الورقة التي بدأت تطوير المكتبة: https://arxiv.org/abs/2002.02497
Joseph Paul Cohen and Mohammad Hashir and Rupert Brooks and Hadrien Bertrand
On the limits of cross-domain generalization in automated X-ray prediction.
Medical Imaging with Deep Learning 2020 (Online: https://arxiv.org/abs/2002.02497)
@inproceedings{cohen2020limits,
title={On the limits of cross-domain generalization in automated X-ray prediction},
author={Cohen, Joseph Paul and Hashir, Mohammad and Brooks, Rupert and Bertrand, Hadrien},
booktitle={Medical Imaging with Deep Learning},
year={2020},
url={https://arxiv.org/abs/2002.02497}
}
CIFAR (المعهد الكندي للبحث المتقدم) | ميلا ، معهد كيبيك منظمة العفو الدولية ، جامعة مونتريال |
|---|---|
مركز جامعة ستانفورد الذكاء الاصطناعي في الطب والتصوير | Carestream Health |