Papier jetzt online! https://arxiv.org/abs/2111.00595
Dokumentation jetzt online! https://mlmed.org/torchxrayvision/
| (? Promo Video) ) |
|---|
Eine Bibliothek für Röntgendatensätze und -modelle für Brust. Einschließlich vorgebliebener Modelle.
Torchxrayvision ist eine Open-Source-Software-Bibliothek für die Arbeit mit Röntgendatensätzen und Deep-Learning-Modellen. Es bietet eine gemeinsame Schnittstelle und gemeinsame Vorverarbeitungskette für eine Vielzahl öffentlich verfügbarer Röntgendatensätze der Brust. Darüber hinaus sind eine Reihe von Lernmodellen für Klassifizierungs- und Darstellungslernmodelle mit unterschiedlichen Architekturen, die auf verschiedenen Datenkombinationen geschult sind, über die Bibliothek verfügbar, um als Baselines oder Feature -Extraktoren zu dienen.
Twitter: @torchxrayvision
$ pip install torchxrayvision
import torchxrayvision as xrv
import skimage , torch , torchvision
# Prepare the image:
img = skimage . io . imread ( "16747_3_1.jpg" )
img = xrv . datasets . normalize ( img , 255 ) # convert 8-bit image to [-1024, 1024] range
img = img . mean ( 2 )[ None , ...] # Make single color channel
transform = torchvision . transforms . Compose ([ xrv . datasets . XRayCenterCrop (), xrv . datasets . XRayResizer ( 224 )])
img = transform ( img )
img = torch . from_numpy ( img )
# Load model and process image
model = xrv . models . DenseNet ( weights = "densenet121-res224-all" )
outputs = model ( img [ None ,...]) # or model.features(img[None,...])
# Print results
dict ( zip ( model . pathologies , outputs [ 0 ]. detach (). numpy ()))
{ 'Atelectasis' : 0.32797316 ,
'Consolidation' : 0.42933336 ,
'Infiltration' : 0.5316924 ,
'Pneumothorax' : 0.28849724 ,
'Edema' : 0.024142697 ,
'Emphysema' : 0.5011832 ,
'Fibrosis' : 0.51887786 ,
'Effusion' : 0.27805611 ,
'Pneumonia' : 0.18569896 ,
'Pleural_Thickening' : 0.24489835 ,
'Cardiomegaly' : 0.3645515 ,
'Nodule' : 0.68982 ,
'Mass' : 0.6392845 ,
'Hernia' : 0.00993878 ,
'Lung Lesion' : 0.011150705 ,
'Fracture' : 0.51916164 ,
'Lung Opacity' : 0.59073937 ,
'Enlarged Cardiomediastinum' : 0.27218717 }Ein Beispielskript zum Verarbeiten von Bildern.
$ python3 process_image.py ../tests/00000001_000.png
{'preds': {'Atelectasis': 0.50500506,
'Cardiomegaly': 0.6600903,
'Consolidation': 0.30575264,
'Edema': 0.274184,
'Effusion': 0.4026162,
'Emphysema': 0.5036339,
'Enlarged Cardiomediastinum': 0.40989172,
'Fibrosis': 0.53293407,
'Fracture': 0.32376793,
'Hernia': 0.011924741,
'Infiltration': 0.5154413,
'Lung Lesion': 0.22231922,
'Lung Opacity': 0.2772148,
'Mass': 0.32237658,
'Nodule': 0.5091847,
'Pleural_Thickening': 0.5102617,
'Pneumonia': 0.30947986,
'Pneumothorax': 0.24847917}}
Geben Sie Gewichte für vorbereitete Modelle an (derzeit alle Densenet121). Hinweis: Jedes vorgespannte Modell verfügt über 18 Ausgänge. Das all hat jeden Ausgang geschult. Für die anderen Gewichte sind einige Ziele jedoch nicht geschult und prognostizieren zufällig, weil sie im Trainingsdatensatz nicht existieren. Die einzigen gültigen Ausgänge sind im Feld {dataset}.pathologies auf dem Datensatz aufgeführt, das den Gewichten entspricht.
## 224x224 models
model = xrv . models . DenseNet ( weights = "densenet121-res224-all" )
model = xrv . models . DenseNet ( weights = "densenet121-res224-rsna" ) # RSNA Pneumonia Challenge
model = xrv . models . DenseNet ( weights = "densenet121-res224-nih" ) # NIH chest X-ray8
model = xrv . models . DenseNet ( weights = "densenet121-res224-pc" ) # PadChest (University of Alicante)
model = xrv . models . DenseNet ( weights = "densenet121-res224-chex" ) # CheXpert (Stanford)
model = xrv . models . DenseNet ( weights = "densenet121-res224-mimic_nb" ) # MIMIC-CXR (MIT)
model = xrv . models . DenseNet ( weights = "densenet121-res224-mimic_ch" ) # MIMIC-CXR (MIT)
# 512x512 models
model = xrv . models . ResNet ( weights = "resnet50-res512-all" )
# DenseNet121 from JF Healthcare for the CheXpert competition
model = xrv . baseline_models . jfhealthcare . DenseNet ()
# Official Stanford CheXpert model
model = xrv . baseline_models . chexpert . DenseNet ( weights_zip = "chexpert_weights.zip" )
# Emory HITI lab race prediction model
model = xrv . baseline_models . emory_hiti . RaceModel ()
model . targets - > [ "Asian" , "Black" , "White" ]
# Riken age prediction model
model = xrv . baseline_models . riken . AgeModel ()Benchmarks der Modi finden Sie hier: Benchmarks.md und die Leistung einiger Modelle ist in diesem Papier zu sehen.
Sie können auch einen vorgeborenen AutoCodierer laden, der auf Padchest, NIH, Chexpert und Mimic Datasets trainiert wird.
ae = xrv . autoencoders . ResNetAE ( weights = "101-elastic" )
z = ae . encode ( image )
image2 = ae . decode ( z )Sie können vorgezogene anatomische Segmentierungsmodelle laden. Demo -Notizbuch
seg_model = xrv . baseline_models . chestx_det . PSPNet ()
output = seg_model ( image )
output . shape # [1, 14, 512, 512]
seg_model . targets # ['Left Clavicle', 'Right Clavicle', 'Left Scapula', 'Right Scapula',
# 'Left Lung', 'Right Lung', 'Left Hilus Pulmonis', 'Right Hilus Pulmonis',
# 'Heart', 'Aorta', 'Facies Diaphragmatica', 'Mediastinum', 'Weasand', 'Spine'] 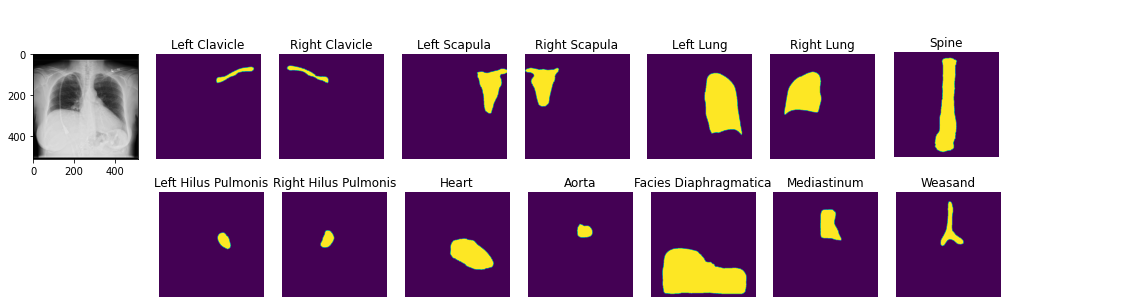
Weitere Informationen zu jedem Datensatz- und Demo -Notizbuch sowie beispielsweise laden Sie detaillierter detaillierte Informationen
transform = torchvision . transforms . Compose ([ xrv . datasets . XRayCenterCrop (),
xrv . datasets . XRayResizer ( 224 )])
# RSNA Pneumonia Detection Challenge. https://pubs.rsna.org/doi/full/10.1148/ryai.2019180041
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "path to stage_2_train_images_jpg" ,
transform = transform )
# CheXpert: A Large Chest Radiograph Dataset with Uncertainty Labels and Expert Comparison. https://arxiv.org/abs/1901.07031
d_chex = xrv . datasets . CheX_Dataset ( imgpath = "path to CheXpert-v1.0-small" ,
csvpath = "path to CheXpert-v1.0-small/train.csv" ,
transform = transform )
# National Institutes of Health ChestX-ray8 dataset. https://arxiv.org/abs/1705.02315
d_nih = xrv . datasets . NIH_Dataset ( imgpath = "path to NIH images" )
# A relabelling of a subset of NIH images from: https://pubs.rsna.org/doi/10.1148/radiol.2019191293
d_nih2 = xrv . datasets . NIH_Google_Dataset ( imgpath = "path to NIH images" )
# PadChest: A large chest x-ray image dataset with multi-label annotated reports. https://arxiv.org/abs/1901.07441
d_pc = xrv . datasets . PC_Dataset ( imgpath = "path to image folder" )
# COVID-19 Image Data Collection. https://arxiv.org/abs/2006.11988
d_covid19 = xrv . datasets . COVID19_Dataset () # specify imgpath and csvpath for the dataset
# SIIM Pneumothorax Dataset. https://www.kaggle.com/c/siim-acr-pneumothorax-segmentation
d_siim = xrv . datasets . SIIM_Pneumothorax_Dataset ( imgpath = "dicom-images-train/" ,
csvpath = "train-rle.csv" )
# VinDr-CXR: An open dataset of chest X-rays with radiologist's annotations. https://arxiv.org/abs/2012.15029
d_vin = xrv . datasets . VinBrain_Dataset ( imgpath = ".../train" ,
csvpath = ".../train.csv" )
# National Library of Medicine Tuberculosis Datasets. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4256233/
d_nlmtb = xrv . datasets . NLMTB_Dataset ( imgpath = "path to MontgomerySet or ChinaSet_AllFiles" )Jeder Datensatz enthält eine Reihe von Feldern. Diese Felder werden beibehalten, wenn xrv.datasets.subset_dataset und xrv.datasets.merge_dataset verwendet werden.
.pathologies Dieses Feld ist eine Liste der in diesem Datensatz enthaltenen Pathologien, die im Feld .labels enthalten sein wird].
.labels Dieses Feld enthält eine 1,0 oder NAN für jedes in .pathologies definierte Etikett.
.csv Dieses Feld ist ein Pandas -Datenframe der Metadaten -CSV -Datei, die mit den Daten geliefert wird. Jede Zeile richtet sich an die Elemente des Datensatzes, sodass die Indexierung mit .iloc funktioniert.
Wenn möglich, verfügt die .csv jedes Datensatzes über einige gemeinsame Felder des CSV. Diese werden ausgerichtet, wenn die Liste wie folgt lautet:
csv.patientid Eine eindeutige ID, die die Stichproben in diesem Datensatz eindeutig identifiziert
csv.offset_day_int Ein ganzzahliger Zeitversatz für das Bild in der Einheit der Tage. Dies wird für relative Zeiten erwartet und hat keine absolute Bedeutung, obwohl es für einige Datensätze die Epochenzeit ist.
csv.age_years das Alter des Patienten in Jahren.
csv.sex_male Wenn der Patient männlich ist
csv.sex_female Wenn die Patientin weiblich ist
relabel_dataset wird die Beschriftungen so ausrichten, dass die gleiche Reihenfolge wie das Pathologies -Argument aufweist.
xrv . datasets . relabel_dataset ( xrv . datasets . default_pathologies , d_nih ) # has side effectsGeben Sie eine Teilmenge von Ansichten an (Demo -Notizbuch).
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "..." ,
views = [ "PA" , "AP" , "AP Supine" ])Geben Sie nur 1 Bild pro Patient an
d_kaggle = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "..." ,
unique_patients = True )Erhalten Sie zusammenfassende Statistiken pro Datensatz
d_chex = xrv . datasets . CheX_Dataset ( imgpath = "CheXpert-v1.0-small" ,
csvpath = "CheXpert-v1.0-small/train.csv" ,
views = [ "PA" , "AP" ], unique_patients = False )
CheX_Dataset num_samples = 191010 views = [ 'PA' , 'AP' ]
{ 'Atelectasis' : { 0.0 : 17621 , 1.0 : 29718 },
'Cardiomegaly' : { 0.0 : 22645 , 1.0 : 23384 },
'Consolidation' : { 0.0 : 30463 , 1.0 : 12982 },
'Edema' : { 0.0 : 29449 , 1.0 : 49674 },
'Effusion' : { 0.0 : 34376 , 1.0 : 76894 },
'Enlarged Cardiomediastinum' : { 0.0 : 26527 , 1.0 : 9186 },
'Fracture' : { 0.0 : 18111 , 1.0 : 7434 },
'Lung Lesion' : { 0.0 : 17523 , 1.0 : 7040 },
'Lung Opacity' : { 0.0 : 20165 , 1.0 : 94207 },
'Pleural Other' : { 0.0 : 17166 , 1.0 : 2503 },
'Pneumonia' : { 0.0 : 18105 , 1.0 : 4674 },
'Pneumothorax' : { 0.0 : 54165 , 1.0 : 17693 },
'Support Devices' : { 0.0 : 21757 , 1.0 : 99747 }}Masken sind in den folgenden Datensätzen verfügbar:
xrv . datasets . RSNA_Pneumonia_Dataset () # for Lung Opacity
xrv . datasets . SIIM_Pneumothorax_Dataset () # for Pneumothorax
xrv . datasets . NIH_Dataset () # for Cardiomegaly, Mass, Effusion, ...Beispiel Verwendung:
d_rsna = xrv . datasets . RSNA_Pneumonia_Dataset ( imgpath = "stage_2_train_images_jpg" ,
views = [ "PA" , "AP" ],
pathology_masks = True )
# The has_masks column will let you know if any masks exist for that sample
d_rsna . csv . has_masks . value_counts ()
False 20672
True 6012
# Each sample will have a pathology_masks dictionary where the index
# of each pathology will correspond to a mask of that pathology (if it exists).
# There may be more than one mask per sample. But only one per pathology.
sample [ "pathology_masks" ][ d_rsna . pathologies . index ( "Lung Opacity" )] Es funktioniert auch mit data_augmentation, wenn Sie data_aug=data_transforms an den Dataloader weitergeben. Der zufällige Saatgut wird an die Ausrichtung des Bildes und die Maske angepasst.
Die Klasse xrv.datasets.CovariateDataset nimmt zwei Datensätze und zwei Arrays, die die Etiketten darstellen. Die Proben werden mit dem gewünschten Verhältnis von Bildern von jeder Stelle zurückgegeben. Das Ziel hier ist es, eine kovariate Verschiebung zu simulieren, um ein Modell auf eine falsche Funktion zu konzentrieren. Anschließend kann die Verschiebung in den Validierungsdaten umgekehrt werden, was zu einem katastrophalen Versagen bei der Generalisierungsleistung führt.
Verhältnis = 0,0 bedeutet, dass Bilder von D1 ein positives Etikettsverhältnis = 0,5 bedeutet, dass Bilder aus D1 die Hälfte des Verhältnisses von positiven Etiketten haben = 1,0 bedeutet Bilder von D1 haben keine positive Etikett
Bei jedem Verhältnis wird die Anzahl der zurückgegebenen Proben gleich sein.
d = xrv . datasets . CovariateDataset ( d1 = # dataset1 with a specific condition
d1_target = #target label to predict,
d2 = # dataset2 with a specific condition
d2_target = #target label to predict,
mode = "train" , # train, valid, and test
ratio = 0.9 )Primäres Torchxrayvision -Papier: https://arxiv.org/abs/2111.00595
Joseph Paul Cohen, Joseph D. Viviano, Paul Bertin, Paul Morrison, Parsa Torabian, Matteo Guarrera, Matthew P Lungren, Akshay Chaudhari, Rupert Brooks, Mohammad Hashir, Hadrien Bertrand
TorchXRayVision: A library of chest X-ray datasets and models.
Medical Imaging with Deep Learning
https://github.com/mlmed/torchxrayvision, 2020
@inproceedings{Cohen2022xrv,
title = {{TorchXRayVision: A library of chest X-ray datasets and models}},
author = {Cohen, Joseph Paul and Viviano, Joseph D. and Bertin, Paul and Morrison, Paul and Torabian, Parsa and Guarrera, Matteo and Lungren, Matthew P and Chaudhari, Akshay and Brooks, Rupert and Hashir, Mohammad and Bertrand, Hadrien},
booktitle = {Medical Imaging with Deep Learning},
url = {https://github.com/mlmed/torchxrayvision},
arxivId = {2111.00595},
year = {2022}
}
und dieses Papier, das die Entwicklung der Bibliothek initiierte: https://arxiv.org/abs/2002.02497
Joseph Paul Cohen and Mohammad Hashir and Rupert Brooks and Hadrien Bertrand
On the limits of cross-domain generalization in automated X-ray prediction.
Medical Imaging with Deep Learning 2020 (Online: https://arxiv.org/abs/2002.02497)
@inproceedings{cohen2020limits,
title={On the limits of cross-domain generalization in automated X-ray prediction},
author={Cohen, Joseph Paul and Hashir, Mohammad and Brooks, Rupert and Bertrand, Hadrien},
booktitle={Medical Imaging with Deep Learning},
year={2020},
url={https://arxiv.org/abs/2002.02497}
}
CIFAR (Canadian Institute for Advanced Research) | Mila, Quebec AI Institute, Universität von Montreal |
|---|---|
Zentrum der Stanford University für Künstliche Intelligenz in Medizin und Bildgebung | CARESTREAM GESUNDHEIT |