(Anciennement connu sous le nom de cwlab.)
Une application Web open source et prêt pour le cloud pour un déploiement simplifié de workflows de Big Data.
CI / CD:
Conditionnement:
Citation et contribution:
Attention: Workflux est dans l'état bêta et des changements de rupture pourraient être introduits à l'avenir. Cependant, si vous aimez le tester ou même exécuter en production, nous vous soutiendrons.
L'installation peut être effectuée à l'aide de PIP:
python3 -m pip install workflux
Veuillez consulter la section "Configuration" pour une discussion des options disponibles.
Démarrez le serveur Web avec votre configuration personnalisée (ou omettez l'indicateur --config pour utiliser celui par défaut):
workflux up --config config.yaml
Si vous aimez utiliser des conteneurs pour la gestion des dépendances, vous devez installer Docker ou une solution de conteneurisation compatible Docker comme Singularity ou Udocker. Pour fonctionner sur Windows ou MacOS, veuillez installer les versions dédiées Docker: Docker pour Windows, Docker pour Mac
L'utilisation de l'interface Web devrait être explicite avec l'instruction de l'engagement. La section suivante donne un aperçu du scénario d'utilisation de base.
WorkFlux est écrit en python d'agnostique de plate-forme et peut donc être exécuté sur:
Tout coureur CWL qui a une interface de ligne de commande peut être intégré dans WorkFlux afin d'exécuter des flux de travail CWL ou des lavers d'outils, tels que:
Par conséquent, WorkFlux peut être utilisé sur toute infrastructure soutenue par ces coureurs CWL, notamment:
* Veuillez noter:
L'exécution sur Windows n'est prise en charge que par CWLTool qui parle à Docker pour Windows. Par conséquent, les outils et les workflows enveloppés de CWL qui, dans l'origine à l'origine pour Linux / MacOS, peuvent être exécutés sur Windows avec une interface graphique fournie par WorkFlux.
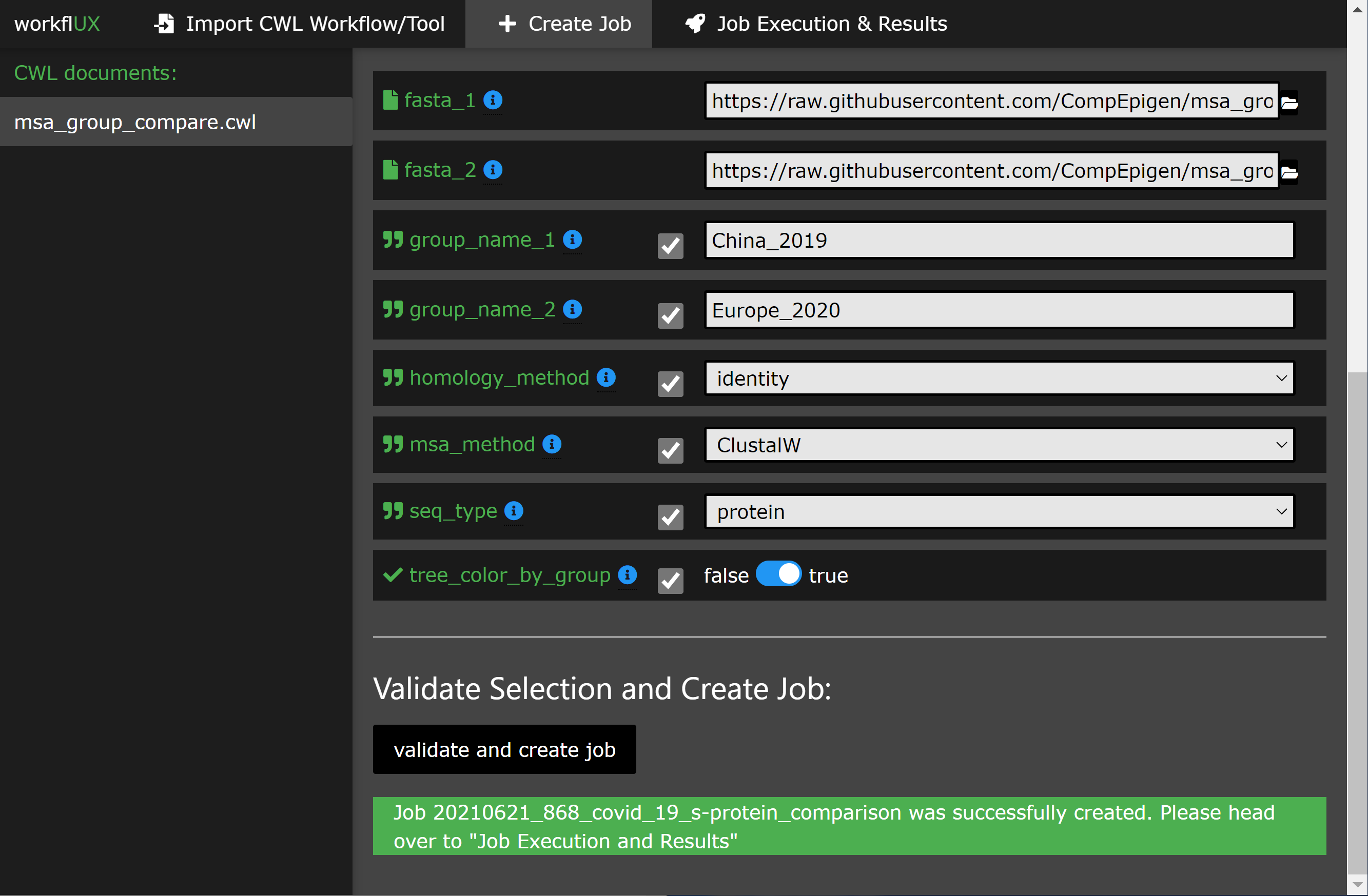
Veuillez consulter notre tutoriel, qui vous guide à travers un exemple simple mais significatif de la façon dont Workflux peut être utilisé pour comparer les séquences de protéines de pointe de Covid-19 dans deux cohortes de patients.
Voici quelques apetiseurs: 
WorkFlux est un package très polyvalent et ne fait presque aucune hypothèse sur votre environnement dur et logiciel utilisé pour l'exécution de CWL. Pour l'adapter à votre système et à votre cas d'utilisation, un ensemble d'options de configuration est disponible:
Toutes les options de configuration peuvent être spécifiées dans un seul fichier YAML qui est fourni à WorkFlux au début:
workflux up --config my_config.yaml
Pour obtenir un exemple de fichier de configuration, exécutez la commande suivante:
workflux print_config > config.yaml (ou voir l'exemple ci-dessous)
Web_server_host :
Spécifiez l'hôte ou l'adresse IP sur laquelle le serveur Web doit s'exécuter. Utilisez localhost pour une utilisation locale sur votre machine uniquement. Utilisez 0.0.0.0 pour permettre l'accessibilité à distance par d'autres machines du même réseau.
Par défaut : localhost
Web_server_port :
Spécifiez le port utilisé par le serveur Web.
Par défaut : 5000
Temp_dir :
Répertoire des fichiers temporaires.
Par défaut : un sous-dossier "Workflux / Temp" dans le répertoire domestique
Workflow_dir :
Répertoire pour enregistrer les documents CWL.
Par défaut : un sous-dossier "Workflux / Temp" dans le répertoire domestique
Exec_dir :
Répertoire pour enregistrer les données d'exécution, y compris les fichiers de sortie.
Par défaut : un sous-dossier "Workflux / Temp" dans le répertoire domestique
Default_input_dir :
Répertoire par défaut où les utilisateurs peuvent rechercher des fichiers d'entrée. Vous pouvez spécifier des répertoires d'entrée supplémentaires à l'aide du paramètre " add_input_dirs ". Par défaut : un sous-dossier "Workflux / Temp" dans le répertoire domestique
Db_dir :
Répertoire des bases de données.
Par défaut : un sous-dossier "Workflux / Temp" dans le répertoire domestique
Add_input_dirs :
En plus de " default_input_dir ", ces répertoires peuvent être recherchés par l'utilisateur pour les fichiers d'entrée.
Veuillez les spécifier dans le format " Nom: Path " comme illustré dans cet exemple:
ADD_INPUT_DIRS:
GENOMES_DIR: '/ngs_share/genomes'
PUBLIC_GEO_DATA: '/datasets/public/geo'
Par défaut : pas de réseaux d'entrée supplémentaires.
Add_input_and_upload_dirs :
Les utilisateurs peuvent rechercher ces répertoires pour les fichiers d'entrée (en plus de " default_input_dir ") et ils peuvent également télécharger leurs fichiers.
Veuillez les spécifier dans le format " Nom: Path " comme illustré dans cet exemple:
ADD_INPUT_AND_UPLOAD_DIRS:
UPLOAD_SCRATCH: '/scratch/upload'
PERMANEN_UPLOAD_STORE: '/datasets/upload'
Par défaut : pas de réseaux d'entrée supplémentaires.
Débogage :
Si elle est définie sur true, le mode de débogage est activé. N'utilisez pas sur les systèmes de production.
Par défaut : faux
C'est là que vous configurez comment exécuter des travaux CWL sur votre système. Un profil se compose de quatre étapes: Préparez, EXEC, EVAL et finalisez (seul EXEC requis, le reste est facultatif). Pour chaque étape, vous pouvez spécifier des commandes qui sont exécutées dans Bash ou CMD Terminal.
Vous pouvez définir plusieurs profil d'exécution comme indiqué dans l'exemple de configuration ci-dessous. Cela permet aux utilisateurs de frontend de choisir entre différentes options d'exécution (par exemple, à l'aide de différents coureurs CWL, différents systèmes de gestion des dépendances, ou même de choisir un entre plusieurs infrastructures d'exécution par lots disponibles comme LSF, PBS, ...). Pour chaque profil d'exécution, les paramètres de configuration suivants sont disponibles (mais seul le type et l'exec sont requis):
taper :
Spécifiez quel shell / interprète utiliser. Pour Linux ou MacOS, utilisez bash . Pour Windows, utilisez powershell .
Requis .
MAX_RETRIES : Spécifiez combien de fois l'exécution (toutes les étapes) est remise avant de marquer une exécution en cas d'échec.
temps mort :
Pour chaque étape du profil d'exécution, vous pouvez définir une limite de délai d'expiration.
Défaut :
prepare : 120
exec : 86400
eval : 120
finalize : 120 préparer *:
Commandes qui sont exécutées avant l'exécution réelle de CWL. Par exemple pour charger les environnements Python / conda requis.
Facultatif .
exec *:
Commandes pour démarrer l'exécution CWL. Habituellement, ce n'est que la ligne de commande pour exécuter le coureur CWL. Le stdout et le stderr du coureur CWL doivent être redirigés vers le fichier journal prédéfini.
Requis .
ev *:
L'état de sortie à la fin de l'étape EXEC est automatiquement vérifié. Ici, vous pouvez spécifier des commandes pour évaluer en outre le contenu du journal d'exécution pour déterminer si l'exécution a réussi. Pour communiquer l'échec de WorkFlux, définissez la variable SUCCESS sur False .
Facultatif .
Finalize *: Commandes qui sont exécutées après Exec et Ev . Par exemple, cela peut être utilisé pour nettoyer les fichiers temporaires.
* Notes supplémentaires concernant les étapes du profil d'exécution:
JOB_IDRUN_ID (veuillez noter: n'est unique que dans un travail)WORKFLOW (le chemin vers le document CWL utilisé)RUN_INPUT (le chemin d'accès au fichier yaml contenant des paramètres d'entrée)OUTPUT_DIR (le chemin du répertoire de sortie spécifique à l'exécution)LOG_FILE (le chemin du fichier journal qui devrait recevoir le stdout et le stderr de CWL Runner)SUCCESS (s'il est défini sur False , la course sera marquée comme échoué et terminé)PYTHON_PATH (le chemin vers l'interprète Python utilisé pour exécuter WorkFlux)SUCCESS sur False .{...} ) doit être en une seule ligne.Ci-dessous, vous pouvez trouver des exemples de configurations pour l'exécution locale de workflows ou d'outils CWL avec CWLTool.
WEB_SERVER_HOST : localhost
WEB_SERVER_PORT : 5000
DEBUG : False
TEMP_DIR : ' /home/workflux_user/workflux/temp '
WORKFLOW_DIR : ' /home/workflux_user/workflux/workflows '
EXEC_DIR : ' /datasets/processing_out/ '
DEFAULT_INPUT_DIR : ' /home/workflux_user/workflux/input '
DB_DIR : ' /home/workflux_user/workflux/db '
ADD_INPUT_DIRS :
GENOMES_DIR : ' /ngs_share/genomes '
PUBLIC_GEO_DATA : ' /datasets/public/geo '
ADD_INPUT_AND_UPLOAD_DIRS :
UPLOAD_SCRATCH : ' /scratch/upload '
PERMANEN_UPLOAD_STORE : ' /datasets/upload '
EXEC_PROFILES :
cwltool_local :
type : bash
max_retries : 2
timeout :
prepare : 120
exec : 86400
eval : 120
finalize : 120
exec : |
cwltool --outdir "${OUTPUT_DIR}" "${WORKFLOW}" "${RUN_INPUT}"
>> "${LOG_FILE}" 2>&1
eval : |
LAST_LINE=$(tail -n 1 ${LOG_FILE})
if [[ "${LAST_LINE}" == *"Final process status is success"* ]]
then
SUCCESS=True
else
SUCCESS=False
ERR_MESSAGE="cwltool failed - ${LAST_LINE}"
fi WEB_SERVER_HOST : localhost
WEB_SERVER_PORT : 5000
DEBUG : False
TEMP_DIR : ' C:Usersworkflux_userworkfluxtemp '
WORKFLOW_DIR : ' C:Usersworkflux_userworkfluxworkflows '
EXEC_DIR : ' D:processing_out '
DEFAULT_INPUT_DIR : ' C:Usersworkflux_userworkfluxinput '
DB_DIR : ' C:Usersworkflux_userworkfluxdb '
ADD_INPUT_DIRS :
GENOMES_DIR : ' E:genomes '
PUBLIC_GEO_DATA : ' D:publicgeo '
ADD_INPUT_AND_UPLOAD_DIRS :
UPLOAD_SCRATCH : ' E:upload '
PERMANEN_UPLOAD_STORE : ' D:upload '
EXEC_PROFILES :
cwltool_windows :
type : powershell
max_retries : 2
timeout :
prepare : 120
exec : 86400
eval : 120
finalize : 120
exec : |
. "${PYTHON_PATH}" -m cwltool --debug --default-container ubuntu:16.04 --outdir "${OUTPUT_DIR}" "${CWL}" "${RUN_INPUT}" > "${LOG_FILE}" 2>&1
eval : |
$LAST_LINES = (Get-Content -Tail 2 "${LOG_FILE}")
if ($LAST_LINES.Contains("Final process status is success")){$SUCCESS="True"}
else {$SUCCESS="False"; $ERR_MESSAGE = "cwltool failed - ${LAST_LINE}"} Ce package est gratuit à utiliser et à modifier sous la licence Apache 2.0.
Merci à ces gens merveilleux (clé emoji):
Kersten Breuer ? | Pavlo lutsik ? ? | Sven Twardziok | Marius ? | Lukas Jelonk | Michael Franklin | Alex Kanitz |
Yoann PageAud | Yassen Assenov ? | Yuyu Lin ? |
Ce projet suit les spécifications de tous les contributeurs. Contributions de toute nature bienvenue!