(Früher bekannt als Cwlab.)
Eine Open-Source-Cloud-fertige Webanwendung für eine vereinfachte Bereitstellung von Big-Data-Workflows.
CI/CD:
Verpackung:
Zitat & Beitrag:
Achtung: WorkFlux befindet sich im Beta -Zustand und es könnte in Zukunft geführt werden. Wenn Sie es jedoch testen oder sogar in der Produktion betreiben möchten, werden wir Sie unterstützen.
Die Installation kann mit PIP durchgeführt werden:
python3 -m pip install workflux
Weitere Informationen finden Sie im Abschnitt "Konfiguration" für die verfügbaren Optionen.
Starten Sie den WebServer mit Ihrer benutzerdefinierten Konfiguration (oder lassen Sie das Flag --config -Flag heraus, um die Standardeinstellung zu verwenden):
workflux up --config config.yaml
Wenn Sie Container für das Abhängigkeitsmanagement verwenden möchten, müssen Sie Docker oder eine Docker-kompatible Containerisierungslösung wie Singularität oder Udocker installieren. Um unter Windows oder MacOS auszuführen, installieren Sie bitte die dedizierten Docker -Versionen: Docker für Windows, Docker für Mac
Die Verwendung der Webschnittstelle sollte mit Build-In-Anweisungen selbsterklärend sein. Der folgende Abschnitt gibt einen Überblick über das Grundnutzungsszenario.
WorkFlux ist in Plattform-Agnostic Python geschrieben und kann daher ausgeführt werden:
Jeder CWL-Läufer mit einer Befehlszeilenschnittstelle kann in WorkFlux integriert werden, um CWL-Workflows oder Tool-Wrapper auszuführen, z. B.:
Daher kann WorkFlux für jede Infrastruktur verwendet werden, die von diesen CWL -Läufern unterstützt wird, darunter:
* Bitte beachten Sie:
Die Ausführung unter Windows wird nur von CWLTOOL unterstützt, das mit Docker für Windows spricht. Daher können CWL-umgestaltete Tools und Workflows, in denen ursprünglich für Linux/MacOS entwickelt wurde, auf Windows mit einer grafischen Schnittstelle von WorkFlux ausgeführt werden.
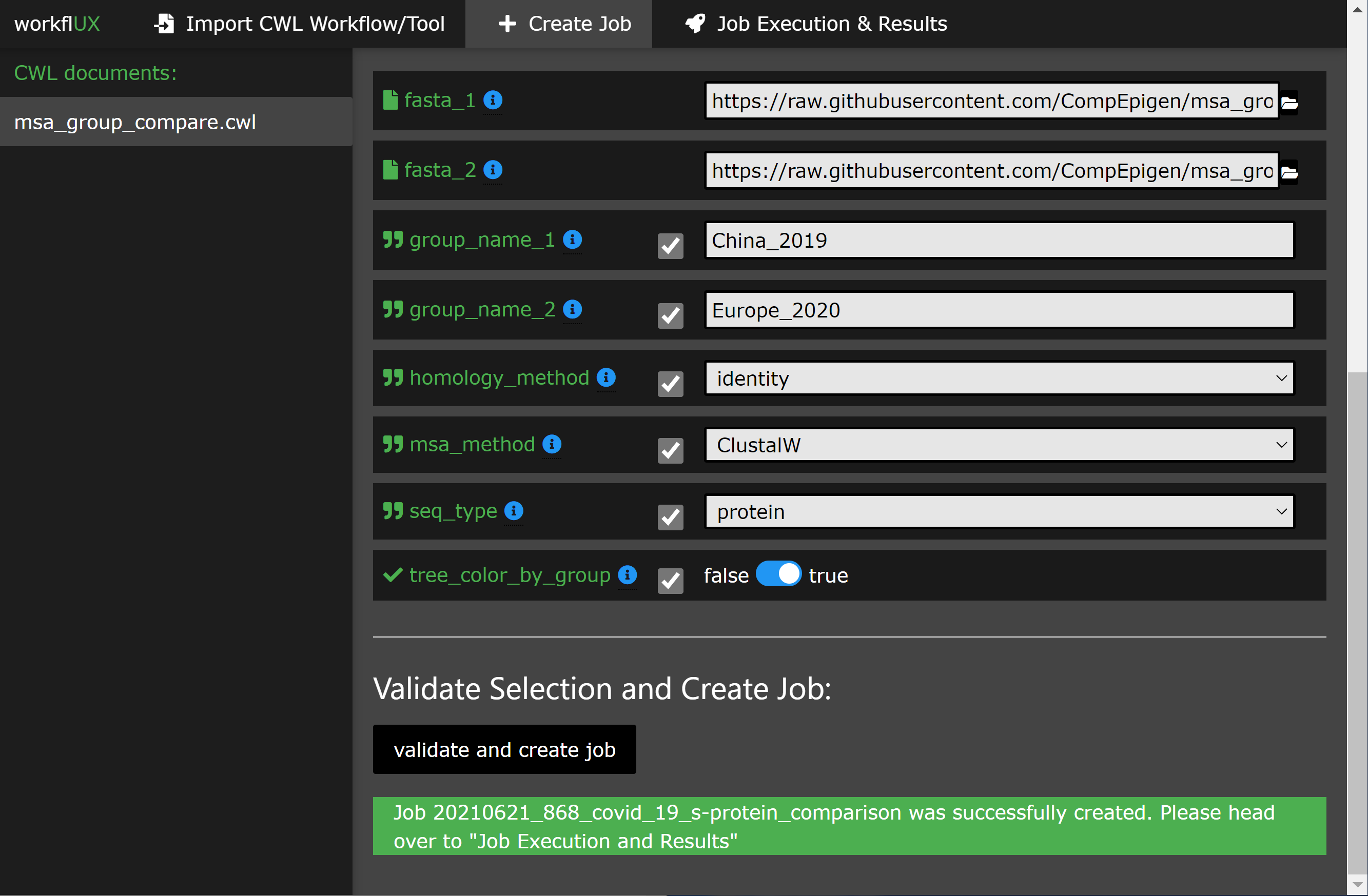
Bitte sehen Sie unser Tutorial, in dem Sie durch ein einfaches, aber aussagekräftiges Beispiel dafür führt, wie Workflux verwendet werden kann, um die Spike-Proteinsequenzen von Covid-19 in zwei Patientenkohorten zu vergleichen.
Hier sind einige Apetizer: 
WorkFlux ist ein sehr vielseitiges Paket und trifft fast keine Annahmen in Ihrer harten und softwarigen Umgebung, die für die Ausführung von CWL verwendet wird. Um es an Ihr System und Ihr Anwendungsfall anzupassen, ist eine Reihe von Konfigurationsoptionen verfügbar:
Alle Konfigurationsoptionen können in einer einzelnen YAML -Datei angegeben werden, die zu Start für WorkFlux zur Verfügung gestellt wird:
workflux up --config my_config.yaml
Um eine Beispielkonfigurationsdatei zu erhalten, führen Sie den folgenden Befehl aus:
workflux print_config > config.yaml (oder siehe das Beispiel unten)
Web_server_host :
Geben Sie die Host- oder IP -Adresse an, auf der der Webserver ausgeführt wird. Verwenden Sie localhost nur für die lokale Nutzung auf Ihrer Maschine. Verwenden Sie 0.0.0.0 , um die Remote -Zugänglichkeit durch andere Maschinen im selben Netzwerk zu ermöglichen.
Standard : localhost
Web_server_port :
Geben Sie den vom Webserver verwendeten Port an.
Standard : 5000
Temp_dir :
Verzeichnis für temporäre Dateien.
Standardeinstellung : Ein Unterordner "Workflux/Temp" im Heimverzeichnis
Workflow_dir :
Verzeichnis zum Speichern von CWL -Dokumenten.
Standardeinstellung : Ein Unterordner "Workflux/Temp" im Heimverzeichnis
Exec_dir :
Verzeichnis zum Speichern von Ausführungsdaten einschließlich Ausgabedateien.
Standardeinstellung : Ein Unterordner "Workflux/Temp" im Heimverzeichnis
Default_input_dir :
Standardverzeichnis, in dem Benutzer nach Eingabedateien suchen können. Sie können zusätzliche Eingabeverzeichnisse mit dem Parameter " add_input_dirs " angeben. Standardeinstellung : Ein Unterordner "Workflux/Temp" im Heimverzeichnis
Db_dir :
Verzeichnis für Datenbanken.
Standardeinstellung : Ein Unterordner "Workflux/Temp" im Heimverzeichnis
Add_input_dirs :
Zusätzlich zu " default_input_dir " können diese Verzeichnisse vom Benutzer nach Eingabedateien durchsucht werden.
Bitte geben Sie sie im Format an " Name: Pfad ", wie in diesem Beispiel gezeigt:
ADD_INPUT_DIRS:
GENOMES_DIR: '/ngs_share/genomes'
PUBLIC_GEO_DATA: '/datasets/public/geo'
Standard : Keine zusätzlichen Eingabediren.
Add_input_and_upload_dirs :
Benutzer können diese Verzeichnisse nach Eingabedateien durchsuchen (zusätzlich zu " default_input_dir ") und sie können auch ihre Einzeldateien hochladen.
Bitte geben Sie sie im Format an " Name: Pfad ", wie in diesem Beispiel gezeigt:
ADD_INPUT_AND_UPLOAD_DIRS:
UPLOAD_SCRATCH: '/scratch/upload'
PERMANEN_UPLOAD_STORE: '/datasets/upload'
Standard : Keine zusätzlichen Eingabediren.
Debugg :
Wenn der Debugging -Modus eingestellt ist, wird eingeschaltet. Verwenden Sie nicht auf Produktionssystemen.
Standard : Falsch
Hier konfigurieren Sie, wie Sie CWL -Jobs in Ihrem System ausführen. Ein Profil besteht aus vier Schritten: Vorbereiten, EXEC, Evaling und Fertigstellung (nur Exec erforderlich, der Rest ist optional). Für jeden Schritt können Sie Befehle angeben, die im Bash- oder CMD -Terminal ausgeführt werden.
Sie können mehrere Ausführungsprofile wie im folgenden Konfigurationsbeispiel definieren. Auf diese Weise können Frontend -Benutzer zwischen verschiedenen Ausführungsoptionen auswählen (z. B. die Verwendung verschiedener CWL -Läufer, unterschiedliche Abhängigkeitsmanagementsysteme oder sogar einen zwischen mehreren verfügbaren Batch -Ausführungsinfrastrukturen wie LSF, PBS, ...). Für jedes Ausführungsprofil sind die folgenden Konfigurationsparameter verfügbar (nur Typ und EXEC sind erforderlich):
Typ :
Geben Sie an, welche Shell/Interpreter zu verwenden ist. Für Linux oder MacOS verwenden Sie bash . Verwenden Sie für Windows powershell .
Erforderlich .
MAX_RETRIES : Geben Sie an, wie oft die Ausführung (alle Schritte) vor dem Markieren eines Laufs als fehlgeschlagen neu markiert wird.
Zeitüberschreitung :
Für jeden Schritt im Ausführungsprofil können Sie ein Zeitüberschreitungslimit festlegen.
Standard :
prepare : 120
exec : 86400
eval : 120
finalize : 120 vorbereiten *:
Befehle, die vor der tatsächlichen CWL -Ausführung ausgeführt werden. Zum Beispiel für die Belastung erforderlich Python/Conda -Umgebungen.
Optional .
Exec *:
Befehle, um die CWL -Ausführung zu starten. Normalerweise ist dies nur die Befehlszeile, um den CWL -Läufer auszuführen. Der Stdout und die Stderr des CWL -Läufers sollten in die vordefinierte Protokolldatei umgeleitet werden.
Erforderlich .
Eval *:
Der Exit -Status am Ende des EXEC -Schritts wird automatisch überprüft. Hier können Sie Befehle angeben, um zusätzlich den Inhalt des Ausführungsprotokolls zu bewerten, um festzustellen, ob die Ausführung erfolgreich ist. Um das Versagen des Workfluxs zu kommunizieren, setzen Sie die SUCCESS auf False .
Optional .
Finalize *: Befehle, die nach Exec und Eval ausgeführt werden. Dies kann beispielsweise zum Reinigen von temporären Dateien verwendet werden.
* Zusätzliche Hinweise zu Ausführungsprofilschritten:
JOB_IDRUN_ID (Bitte beachten Sie: Ist nur innerhalb eines Jobs einzigartig)WORKFLOW (der Pfad zum gebrauchten CWL -Dokument)RUN_INPUT (der Pfad zur YAML -Datei mit Eingabeparametern)OUTPUT_DIR (der Pfad des runspezifischen Ausgangsverzeichnisses)LOG_FILE (der Pfad der Protokolldatei, die den STDOut und den STDERR von CWL Runner empfangen sollte)SUCCESS (falls auf False eingestellt wird der Lauf wird als fehlgeschlagen und beendet markiert)PYTHON_PATH (der Pfad zum Python -Interpreter, der zum Ausführen von Workflux verwendet wird)SUCCESS auf False fest.{...} ) in einer Zeile sein muss.Im Folgenden finden Sie Beispielkonfigurationen für die lokale Ausführung von CWL -Workflows oder Tools mit CWLTool.
WEB_SERVER_HOST : localhost
WEB_SERVER_PORT : 5000
DEBUG : False
TEMP_DIR : ' /home/workflux_user/workflux/temp '
WORKFLOW_DIR : ' /home/workflux_user/workflux/workflows '
EXEC_DIR : ' /datasets/processing_out/ '
DEFAULT_INPUT_DIR : ' /home/workflux_user/workflux/input '
DB_DIR : ' /home/workflux_user/workflux/db '
ADD_INPUT_DIRS :
GENOMES_DIR : ' /ngs_share/genomes '
PUBLIC_GEO_DATA : ' /datasets/public/geo '
ADD_INPUT_AND_UPLOAD_DIRS :
UPLOAD_SCRATCH : ' /scratch/upload '
PERMANEN_UPLOAD_STORE : ' /datasets/upload '
EXEC_PROFILES :
cwltool_local :
type : bash
max_retries : 2
timeout :
prepare : 120
exec : 86400
eval : 120
finalize : 120
exec : |
cwltool --outdir "${OUTPUT_DIR}" "${WORKFLOW}" "${RUN_INPUT}"
>> "${LOG_FILE}" 2>&1
eval : |
LAST_LINE=$(tail -n 1 ${LOG_FILE})
if [[ "${LAST_LINE}" == *"Final process status is success"* ]]
then
SUCCESS=True
else
SUCCESS=False
ERR_MESSAGE="cwltool failed - ${LAST_LINE}"
fi WEB_SERVER_HOST : localhost
WEB_SERVER_PORT : 5000
DEBUG : False
TEMP_DIR : ' C:Usersworkflux_userworkfluxtemp '
WORKFLOW_DIR : ' C:Usersworkflux_userworkfluxworkflows '
EXEC_DIR : ' D:processing_out '
DEFAULT_INPUT_DIR : ' C:Usersworkflux_userworkfluxinput '
DB_DIR : ' C:Usersworkflux_userworkfluxdb '
ADD_INPUT_DIRS :
GENOMES_DIR : ' E:genomes '
PUBLIC_GEO_DATA : ' D:publicgeo '
ADD_INPUT_AND_UPLOAD_DIRS :
UPLOAD_SCRATCH : ' E:upload '
PERMANEN_UPLOAD_STORE : ' D:upload '
EXEC_PROFILES :
cwltool_windows :
type : powershell
max_retries : 2
timeout :
prepare : 120
exec : 86400
eval : 120
finalize : 120
exec : |
. "${PYTHON_PATH}" -m cwltool --debug --default-container ubuntu:16.04 --outdir "${OUTPUT_DIR}" "${CWL}" "${RUN_INPUT}" > "${LOG_FILE}" 2>&1
eval : |
$LAST_LINES = (Get-Content -Tail 2 "${LOG_FILE}")
if ($LAST_LINES.Contains("Final process status is success")){$SUCCESS="True"}
else {$SUCCESS="False"; $ERR_MESSAGE = "cwltool failed - ${LAST_LINE}"} Dieses Paket kann unter der Apache 2.0 -Lizenz kostenlos verwendet und geändert werden.
Vielen Dank an diese wunderbaren Menschen (Emoji -Schlüssel):
Ksten Breuer ? | Pavlo Lutsik ? ? | Sven Twardziok | Marius ? | Lukas Jelonek | Michael Franklin | Alex Kanitz |
Yoann Pageaud | Yassen Assenov ? | Yuyu Lin ? |
Dieses Projekt folgt der All-Contributors-Spezifikation. Beiträge jeglicher Art willkommen!