(Anteriormente conocido como CWLAB).
Una aplicación web de código abierto listos para la nube para la implementación simplificada de flujos de trabajo de Big Data.
CI/CD:
Embalaje:
Cita y contribución:
Atención: WorkFlux está en estado beta y los cambios de ruptura podrían introducirse en el futuro. Sin embargo, si le gusta probarlo o incluso ejecutar en producción, lo apoyaremos.
La instalación se puede realizar con PIP:
python3 -m pip install workflux
Consulte la sección "Configuración" para una discusión de las opciones disponibles.
Inicie el servidor web con su configuración personalizada (o deje de lado el indicador --config para usar el predeterminado):
workflux up --config config.yaml
Si desea utilizar contenedores para la gestión de dependencias, debe instalar Docker o una solución de contenedorización compatible con Docker como Singularity o Udocker. Para ejecutarse en Windows o macOS, instale las versiones de Docker dedicadas: Docker para Windows, Docker para Mac
El uso de la interfaz web debe explicarse por sí mismo con la instrucción incorporada. La siguiente sección ofrece una visión general del escenario de uso básico.
WorkFlux está escrito en Python Agnóstico de plataforma y, por lo tanto, se puede ejecutar en:
Cualquier corredor de CWL que tenga una interfaz de línea de comandos se puede integrar en WorkFlux para ejecutar flujos de trabajo CWL o hábiles de herramientas, como:
Por lo tanto, WorkFlux se puede utilizar en cualquier infraestructura compatible con estos corredores CWL, incluidos:
* Tenga en cuenta:
La ejecución en Windows solo es compatible con CWLTool, que habla con Docker para Windows. Por lo tanto, las herramientas y flujos de trabajo envueltos en CWL que están diseñados originalmente para Linux/macOS se pueden ejecutar en Windows con una interfaz gráfica proporcionada por WorkFlux.
Consulte nuestro tutorial, que lo guía a través de un ejemplo simple pero significativo de cómo se puede usar Workflux para comparar las secuencias de proteínas Spike de Covid-19 en dos cohortes de pacientes.
Aquí hay algunos apetizadores: 
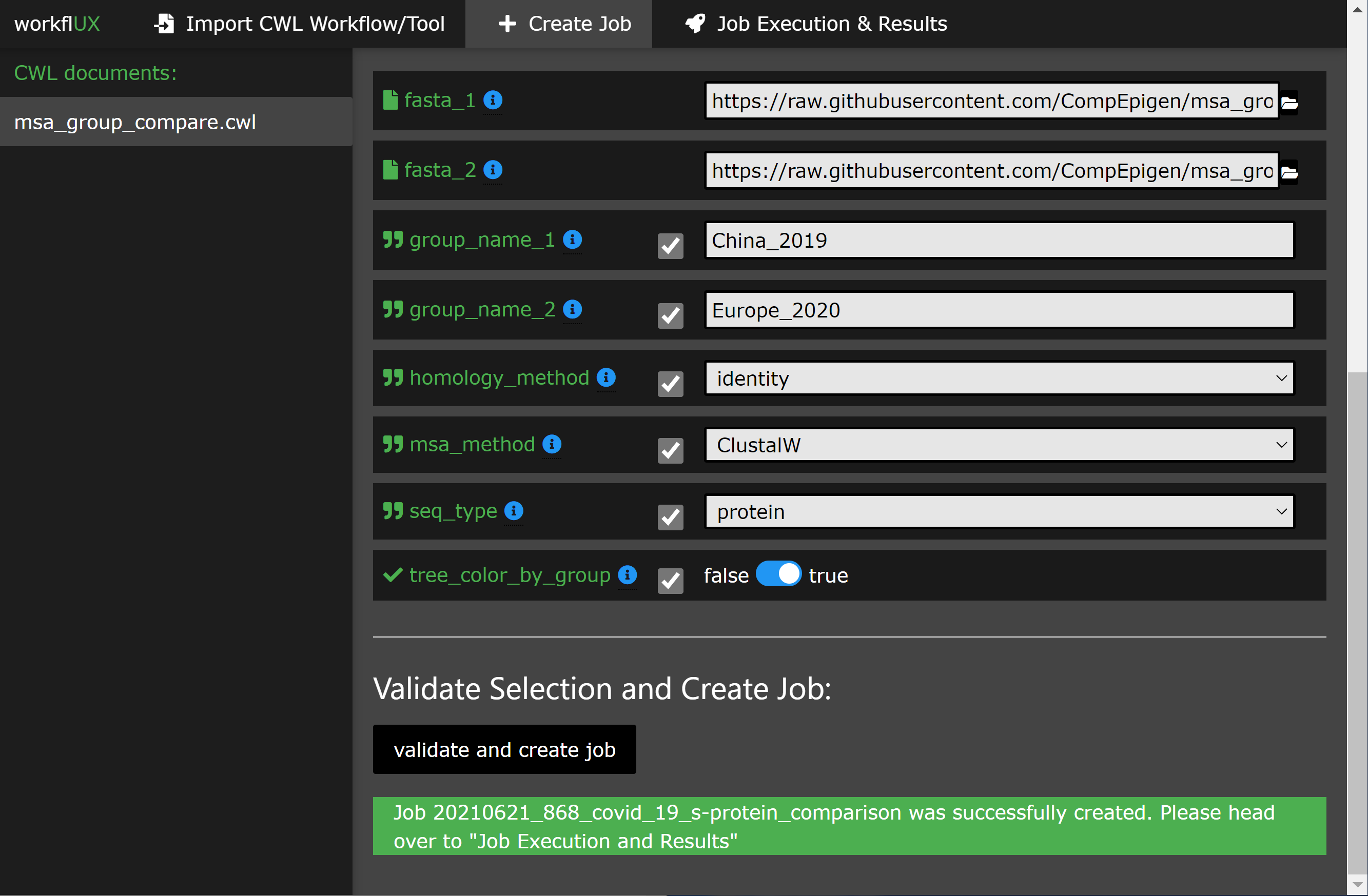
WorkFlux es un paquete altamente versátil y casi no tiene suposiciones en su entorno de software y de software utilizado para la ejecución de CWL. Para adaptarlo a su sistema y caso de uso, está disponible un conjunto de opciones de configuración:
Todas las opciones de configuración se pueden especificar en un solo archivo YAML que se proporciona a WorkFlux al inicio:
workflux up --config my_config.yaml
Para obtener un archivo de configuración de ejemplo, ejecute el siguiente comando:
workflux print_config > config.yaml (o vea el ejemplo a continuación)
Web_server_host :
Especifique la dirección host o IP en la que se ejecutará el servidor web. Use localhost para uso local solo en su máquina. Use 0.0.0.0 para permitir la accesibilidad remota de otras máquinas en la misma red.
Valor predeterminado : localhost
Web_server_port :
Especifique el puerto utilizado por el servidor web.
Valor predeterminado : 5000
Temp_dir :
Directorio para archivos temporales.
Valor predeterminado : una subcarpeta "WorkFlux/Temp" en el directorio de inicio
Workflow_dir :
Directorio para guardar documentos CWL.
Valor predeterminado : una subcarpeta "WorkFlux/Temp" en el directorio de inicio
Exec_dir :
Directorio para guardar los datos de ejecución, incluidos los archivos de salida.
Valor predeterminado : una subcarpeta "WorkFlux/Temp" en el directorio de inicio
Default_input_dir :
Directorio predeterminado donde los usuarios pueden buscar archivos de entrada. Puede especificar directorios de entrada adicionales utilizando el parámetro " add_input_dirs ". Valor predeterminado : una subcarpeta "WorkFlux/Temp" en el directorio de inicio
Db_dir :
Directorio para bases de datos.
Valor predeterminado : una subcarpeta "WorkFlux/Temp" en el directorio de inicio
Add_input_dirs :
Además de " default_input_dir ", el usuario puede buscar estos directorios para los archivos de entrada.
Especifíquelos en el formato " Nombre: ruta " como se muestra en este ejemplo:
ADD_INPUT_DIRS:
GENOMES_DIR: '/ngs_share/genomes'
PUBLIC_GEO_DATA: '/datasets/public/geo'
Valor predeterminado : no hay DIR de entrada adicionales.
Add_input_and_upload_dirs :
Los usuarios pueden buscar estos directorios los archivos de entrada (además de " default_input_dir ") y también pueden cargar sus archivos.
Especifíquelos en el formato " Nombre: ruta " como se muestra en este ejemplo:
ADD_INPUT_AND_UPLOAD_DIRS:
UPLOAD_SCRATCH: '/scratch/upload'
PERMANEN_UPLOAD_STORE: '/datasets/upload'
Valor predeterminado : no hay DIR de entrada adicionales.
Depuración :
Si se establece en True, el modo de depuración está activado. No use en los sistemas de producción.
Valor predeterminado : falso
Aquí es donde configura cómo ejecutar trabajos CWL en su sistema. Un perfil consta de cuatro pasos: preparar, ejecutar, evaluar y finalizar (solo exec requerido, el resto es opcional). Para cada paso, puede especificar comandos que se ejecutan en Bash o Terminal CMD.
Puede definir múltiples perfil de ejecución como se muestra en el ejemplo de configuración a continuación. Esto permite a los usuarios frontend elegir entre diferentes opciones de ejecución (por ejemplo, utilizando diferentes corredores de CWL, diferentes sistemas de gestión de dependencias o incluso elegir un entre múltiples infraestructuras de ejecución por lotes disponibles como LSF, PBS, ...). Para cada perfil de ejecución, los siguientes parámetros de configuración están disponibles (pero solo se requiere Tipo y Exec ):
tipo :
Especifique qué shell/intérprete usar. Para Linux o macOS usan bash . Para Windows, use powershell .
Requerido .
max_retries : especifique cuántas veces la ejecución (todos los pasos) se vuelve a jugar antes de marcar una ejecución como falló.
se acabó el tiempo :
Para cada paso en el perfil de ejecución, puede establecer un límite de tiempo de espera.
Por defecto :
prepare : 120
exec : 86400
eval : 120
finalize : 120 preparar *:
Comandos que se ejecutan antes de la ejecución real de CWL. Por ejemplo, para cargar entornos de pitón/conda requeridos.
Opcional .
ejecutivo *:
Comandos para iniciar la ejecución de CWL. Por lo general, esta es solo la línea de comando para ejecutar el corredor CWL. El stdout y el stderr del corredor CWL deben redirigirse al archivo de registro predefinido.
Requerido .
eval *:
El estado de salida al final del paso EXEC se verifica automáticamente. Aquí puede especificar comandos para evaluar adicionalmente el contenido del registro de ejecución para determinar si la ejecución tuvo éxito. Para comunicar el fracaso a WorkFlux, establezca la variable SUCCESS en False .
Opcional .
Finalizar *: Comandos que se ejecutan después de Exec y Eval . Por ejemplo, esto se puede usar para limpiar archivos temporales.
* Notas adicionales sobre los pasos del perfil de ejecución:
JOB_IDRUN_ID (tenga en cuenta: solo es único dentro de un trabajo)WORKFLOW (la ruta al documento CWL usado)RUN_INPUT (la ruta al archivo YAML que contiene parámetros de entrada)OUTPUT_DIR (la ruta del directorio de salida específico de ejecución)LOG_FILE (la ruta del archivo de registro que debería recibir el stdout y stderr de CWL Runner)SUCCESS (si se establece en False la ejecución se marcará como falló y termina)PYTHON_PATH (el camino hacia el intérprete de Python utilizado para ejecutar Workflux)SUCCESS en False .{...} ) tiene que estar en una línea.A continuación, puede encontrar configuraciones de ejemplo para la ejecución local de flujos de trabajo o herramientas CWL con CWLTool.
WEB_SERVER_HOST : localhost
WEB_SERVER_PORT : 5000
DEBUG : False
TEMP_DIR : ' /home/workflux_user/workflux/temp '
WORKFLOW_DIR : ' /home/workflux_user/workflux/workflows '
EXEC_DIR : ' /datasets/processing_out/ '
DEFAULT_INPUT_DIR : ' /home/workflux_user/workflux/input '
DB_DIR : ' /home/workflux_user/workflux/db '
ADD_INPUT_DIRS :
GENOMES_DIR : ' /ngs_share/genomes '
PUBLIC_GEO_DATA : ' /datasets/public/geo '
ADD_INPUT_AND_UPLOAD_DIRS :
UPLOAD_SCRATCH : ' /scratch/upload '
PERMANEN_UPLOAD_STORE : ' /datasets/upload '
EXEC_PROFILES :
cwltool_local :
type : bash
max_retries : 2
timeout :
prepare : 120
exec : 86400
eval : 120
finalize : 120
exec : |
cwltool --outdir "${OUTPUT_DIR}" "${WORKFLOW}" "${RUN_INPUT}"
>> "${LOG_FILE}" 2>&1
eval : |
LAST_LINE=$(tail -n 1 ${LOG_FILE})
if [[ "${LAST_LINE}" == *"Final process status is success"* ]]
then
SUCCESS=True
else
SUCCESS=False
ERR_MESSAGE="cwltool failed - ${LAST_LINE}"
fi WEB_SERVER_HOST : localhost
WEB_SERVER_PORT : 5000
DEBUG : False
TEMP_DIR : ' C:Usersworkflux_userworkfluxtemp '
WORKFLOW_DIR : ' C:Usersworkflux_userworkfluxworkflows '
EXEC_DIR : ' D:processing_out '
DEFAULT_INPUT_DIR : ' C:Usersworkflux_userworkfluxinput '
DB_DIR : ' C:Usersworkflux_userworkfluxdb '
ADD_INPUT_DIRS :
GENOMES_DIR : ' E:genomes '
PUBLIC_GEO_DATA : ' D:publicgeo '
ADD_INPUT_AND_UPLOAD_DIRS :
UPLOAD_SCRATCH : ' E:upload '
PERMANEN_UPLOAD_STORE : ' D:upload '
EXEC_PROFILES :
cwltool_windows :
type : powershell
max_retries : 2
timeout :
prepare : 120
exec : 86400
eval : 120
finalize : 120
exec : |
. "${PYTHON_PATH}" -m cwltool --debug --default-container ubuntu:16.04 --outdir "${OUTPUT_DIR}" "${CWL}" "${RUN_INPUT}" > "${LOG_FILE}" 2>&1
eval : |
$LAST_LINES = (Get-Content -Tail 2 "${LOG_FILE}")
if ($LAST_LINES.Contains("Final process status is success")){$SUCCESS="True"}
else {$SUCCESS="False"; $ERR_MESSAGE = "cwltool failed - ${LAST_LINE}"} Este paquete es gratuito de usar y modificar bajo la licencia Apache 2.0.
Gracias a estas maravillosas personas (Key Emoji):
Kersten Breuer ? | Pavlo Lutsik ? ? | Sven Twardziok | Mario ? | Lukas Jelonek | Michael Franklin | Alex Kanitz |
Yoann pageaud | Yassen Assenov ? | Yuyu Lin ? |
Este proyecto sigue la especificación de todos los contribuyentes. ¡Contribuciones de cualquier tipo bienvenido!