(以前はCWLABとして知られていました。)
ビッグデータワークフローの簡素化された展開のためのオープンソースのクラウド対応のWebアプリケーション。
CI/CD:
パッケージ:
引用と貢献:
注意:Workfluxはベータ状態にあり、将来的に壊れた変更が導入される可能性があります。ただし、テストしたい場合、または生産で実行したい場合は、サポートします。
インストールは、PIPを使用して実行できます。
python3 -m pip install workflux
利用可能なオプションの説明については、セクション「構成」を参照してください。
カスタム構成でWebServerを起動します(または、デフォルトのフラグを使用するために--configフラグを除外してください):
workflux up --config config.yaml
依存関係管理のためにコンテナを使用したい場合は、DockerまたはSingularityやUdockerなどのDocker互換コンテナ化ソリューションをインストールする必要があります。 WindowsまたはMacOSで実行するには、専用のDockerバージョン:Docker for Windows、Docker for Macをインストールしてください
Webインターフェイスの使用は、ビルドイン命令を使用して自明である必要があります。次のセクションでは、基本的な使用シナリオの概要を示します。
Workfluxはプラットフォームに依存しないPythonで記述されるため、以下で実行できます。
コマンドラインインターフェイスを持つCWLランナーは、次のようなCWLワークフローやツールワッパーを実行するためにワークフルックスに統合できます。
したがって、Workfluxは、次のようなCWLランナーがサポートするインフラストラクチャで使用できます。
*ご注意ください:
Windowsでの実行は、Windowsに対してDockerに話しかけるCWLToolによってのみサポートされています。したがって、CWLで巻きつけられたツールとワークフローは、WorkFluxが提供するグラフィカルインターフェイスを使用して、Linux/macos向けに元々Windowsで実行できます。
私たちのチュートリアルをご覧ください。これは、2つの患者コホートのCovid-19のスパイクタンパク質シーケンスを比較するためにワークフルクスを使用する方法のシンプルでありながら意味のある例をご覧ください。
ここにいくつかのapetizersがあります: 
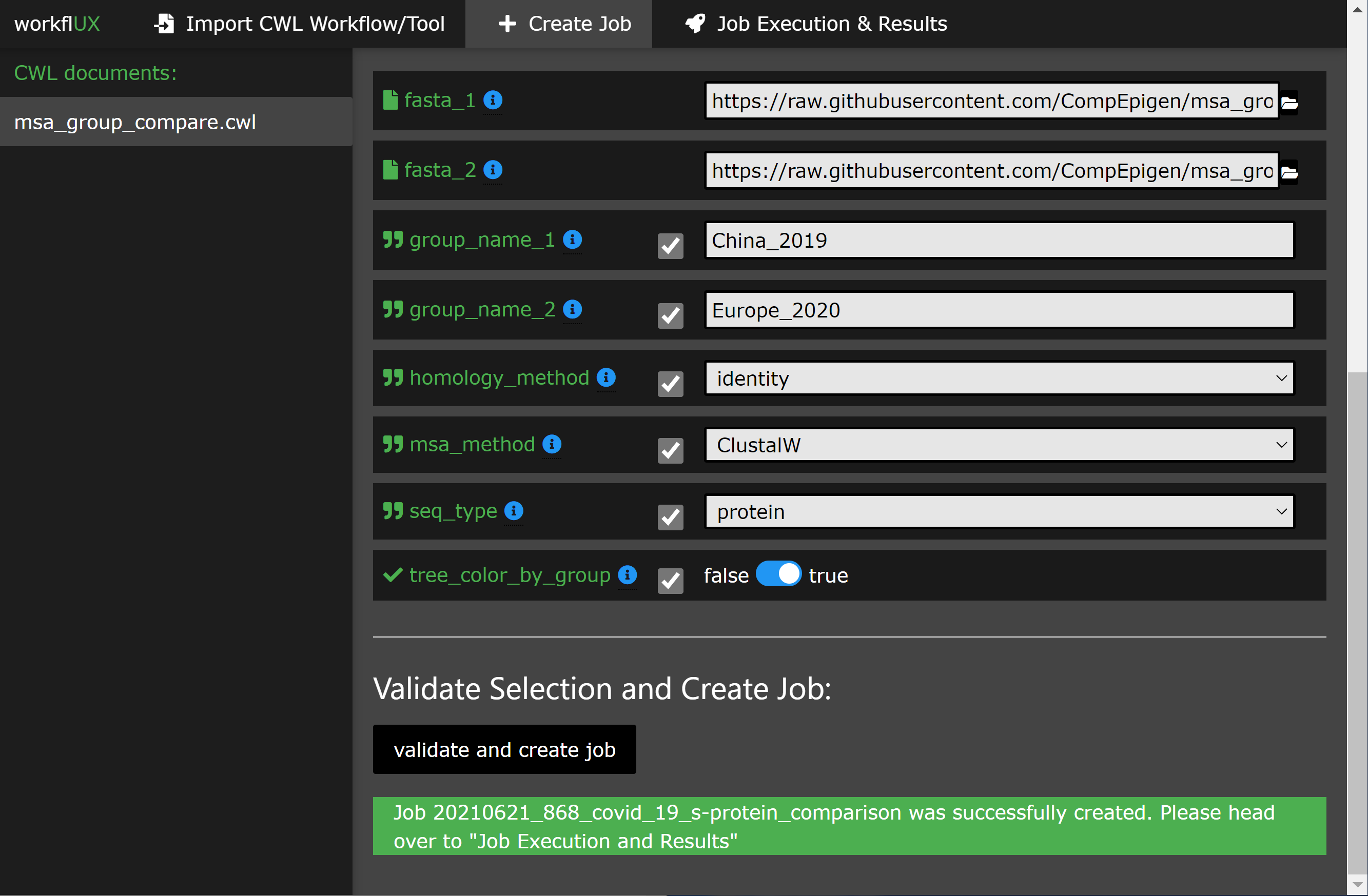
WorkFluxは非常に用途の広いパッケージであり、CWLの実行に使用されるハードおよびソフトウェア環境についてほとんど仮定を行いません。システムとユースケースに適応するために、一連の構成オプションが利用可能です。
すべての構成オプションは、START時にWorkFluxに提供される単一のYAMLファイルで指定できます。
workflux up --config my_config.yaml
configファイルのサンプルを取得するには、次のコマンドを実行します。
workflux print_config > config.yaml (または以下の例を参照)
web_server_host :
Webサーバーが実行されるホストまたはIPアドレスを指定します。マシンでのローカル使用のためにlocalhost使用してください。 0.0.0.0使用して、同じネットワーク内の他のマシンによるリモートアクセシビリティを可能にします。
デフォルト: localhost
web_server_port :
Webサーバーが使用するポートを指定します。
デフォルト:5000
temp_dir :
一時ファイルのディレクトリ。
デフォルト:ホームディレクトリのサブフォルダー「ワークフラックス/テンプ」
workflow_dir :
CWLドキュメントを保存するためのディレクトリ。
デフォルト:ホームディレクトリのサブフォルダー「ワークフラックス/テンプ」
exec_dir :
出力ファイルを含む実行データを保存するためのディレクトリ。
デフォルト:ホームディレクトリのサブフォルダー「ワークフラックス/テンプ」
default_input_dir :
ユーザーが入力ファイルを検索できるデフォルトディレクトリ。 「 add_input_dirs 」パラメーターを使用して、追加の入力ディレクトリを指定できます。デフォルト:ホームディレクトリのサブフォルダー「ワークフラックス/テンプ」
DB_DIR :
データベースのディレクトリ。
デフォルト:ホームディレクトリのサブフォルダー「ワークフラックス/テンプ」
add_input_dirs :
「 default_input_dir 」に加えて、これらのディレクトリはユーザーが入力ファイルを検索できます。
この例に示すように、「名前:パス」という形式で指定してください。
ADD_INPUT_DIRS:
GENOMES_DIR: '/ngs_share/genomes'
PUBLIC_GEO_DATA: '/datasets/public/geo'
デフォルト:追加の入力監督はありません。
add_input_and_upload_dirs :
ユーザーは、これらのディレクトリを[ default_input_dir "に加えて)を検索することもできます。また、1つのファイルをアップロードすることもできます。
この例に示すように、「名前:パス」という形式で指定してください。
ADD_INPUT_AND_UPLOAD_DIRS:
UPLOAD_SCRATCH: '/scratch/upload'
PERMANEN_UPLOAD_STORE: '/datasets/upload'
デフォルト:追加の入力監督はありません。
デバッグ:
Trueに設定すると、デバッグモードがオンになります。生産システムで使用しないでください。
デフォルト:false
これは、システムでCWLジョブを実行する方法を構成する場所です。プロファイルは、準備、exec、評価、および最終化の4つのステップで構成されています(Execのみ、残りはオプションです)。各ステップについて、BASHまたはCMD端子で実行されるコマンドを指定できます。
以下の構成例に示すように、複数の実行プロファイルを定義できます。これにより、FrontEndユーザーは異なる実行オプション(例:異なるCWLランナー、異なる依存関係管理システムを使用するか、LSF、PBSなどの複数の利用可能なバッチ実行インフラストラクチャを選択することもできます。実行プロファイルごとに、次の構成パラメーターが利用可能です(ただし、タイプとexecのみが必要です):
タイプ:
使用するシェル/インタープリターを指定します。 LinuxまたはmacOSの場合はbashを使用します。 Windowsには、 powershellを使用してください。
必須。
MAX_RETRIES :実行が失敗したときに実行をマークする前に、実行(すべてのステップ)が再試行される回数を指定します。
タイムアウト:
実行プロファイルの各ステップについて、タイムアウト制限を設定できます。
デフォルト:
prepare : 120
exec : 86400
eval : 120
finalize : 120準備する*:
実際のCWL実行前に実行されるコマンド。たとえば、必要なPython/Conda環境をロードするために。
オプション。
exec *:
CWL実行を開始するコマンド。通常、これはCWLランナーを実行するコマンドラインにすぎません。 CWLランナーのstdoutとstderrは、事前定義されたログファイルにリダイレクトする必要があります。
必須。
評価*:
execステップの最後にある出口ステータスが自動的にチェックされます。ここでは、コマンドを指定して、実行ログのコンテンツをさらに評価して、実行が成功したかどうかを判断できます。 WorkFluxの失敗を通信するには、 SUCCESS変数をFalseに設定します。
オプション。
実行*: execと評価後に実行されるコマンド。たとえば、これは一時的なファイルをクリーンアップするために使用できます。
*実行プロファイルの手順に関する追加のメモ:
JOB_IDRUN_ID (注:仕事内でのみユニークです)WORKFLOW (使用済みのCWLドキュメントへのパス)RUN_INPUT (入力パラメーターを含むYAMLファイルへのパス)OUTPUT_DIR (実行固有の出力ディレクトリのパス)LOG_FILE (CWLランナーのstdoutとstderrを受信するログファイルのパス)SUCCESS ( Falseに設定されている場合、実行は失敗と終了としてマークされます)PYTHON_PATH (ワークフルを実行するために使用されるpythonインタープリターへのパス)SUCCESS変数をFalseに設定してください。{...}に含まれる)が1行にある必要があることに注意してください。以下に、CWLワークフローまたはCWLToolを使用したツールのローカル実行のための構成の例を見つけることができます。
WEB_SERVER_HOST : localhost
WEB_SERVER_PORT : 5000
DEBUG : False
TEMP_DIR : ' /home/workflux_user/workflux/temp '
WORKFLOW_DIR : ' /home/workflux_user/workflux/workflows '
EXEC_DIR : ' /datasets/processing_out/ '
DEFAULT_INPUT_DIR : ' /home/workflux_user/workflux/input '
DB_DIR : ' /home/workflux_user/workflux/db '
ADD_INPUT_DIRS :
GENOMES_DIR : ' /ngs_share/genomes '
PUBLIC_GEO_DATA : ' /datasets/public/geo '
ADD_INPUT_AND_UPLOAD_DIRS :
UPLOAD_SCRATCH : ' /scratch/upload '
PERMANEN_UPLOAD_STORE : ' /datasets/upload '
EXEC_PROFILES :
cwltool_local :
type : bash
max_retries : 2
timeout :
prepare : 120
exec : 86400
eval : 120
finalize : 120
exec : |
cwltool --outdir "${OUTPUT_DIR}" "${WORKFLOW}" "${RUN_INPUT}"
>> "${LOG_FILE}" 2>&1
eval : |
LAST_LINE=$(tail -n 1 ${LOG_FILE})
if [[ "${LAST_LINE}" == *"Final process status is success"* ]]
then
SUCCESS=True
else
SUCCESS=False
ERR_MESSAGE="cwltool failed - ${LAST_LINE}"
fi WEB_SERVER_HOST : localhost
WEB_SERVER_PORT : 5000
DEBUG : False
TEMP_DIR : ' C:Usersworkflux_userworkfluxtemp '
WORKFLOW_DIR : ' C:Usersworkflux_userworkfluxworkflows '
EXEC_DIR : ' D:processing_out '
DEFAULT_INPUT_DIR : ' C:Usersworkflux_userworkfluxinput '
DB_DIR : ' C:Usersworkflux_userworkfluxdb '
ADD_INPUT_DIRS :
GENOMES_DIR : ' E:genomes '
PUBLIC_GEO_DATA : ' D:publicgeo '
ADD_INPUT_AND_UPLOAD_DIRS :
UPLOAD_SCRATCH : ' E:upload '
PERMANEN_UPLOAD_STORE : ' D:upload '
EXEC_PROFILES :
cwltool_windows :
type : powershell
max_retries : 2
timeout :
prepare : 120
exec : 86400
eval : 120
finalize : 120
exec : |
. "${PYTHON_PATH}" -m cwltool --debug --default-container ubuntu:16.04 --outdir "${OUTPUT_DIR}" "${CWL}" "${RUN_INPUT}" > "${LOG_FILE}" 2>&1
eval : |
$LAST_LINES = (Get-Content -Tail 2 "${LOG_FILE}")
if ($LAST_LINES.Contains("Final process status is success")){$SUCCESS="True"}
else {$SUCCESS="False"; $ERR_MESSAGE = "cwltool failed - ${LAST_LINE}"} このパッケージは、Apache 2.0ライセンスの下で自由に使用および変更できます。
これらの素晴らしい人々に感謝します(絵文字キー):
カーステン・ブルーアー ? | Pavlo lutsik ? ? | Sven twardziok | マリウス ? | ルーカス・ジェロネック | マイケル・フランクリン | アレックス・カニッツ |
ヨアン・パゲー | Yassen Assenov ? | ユユ・リン ? |
このプロジェクトは、全委員会の仕様に従います。あらゆる種類の貢献を歓迎します!