spfy : plataforma para prever subtipos de sequências completas do genoma de E.coli e construir dados gráficos para análises comparativas em toda a população.
Publicado como: Le,KK, Whiteside,MD, Hopkins,JE, Gannon,VPJ, Laing,CR spfy : um banco de dados gráfico integrado para previsão em tempo real de fenótipos bacterianos e análises comparativas downstream. Banco de dados (2018) Vol. 2018: artigo ID bay086; doi:10.1093/database/bay086
Ao vivo: https://lfz.corefacility.ca/superphy/spfy/
git clone --recursive https://github.com/superphy/spfy.gitcd spfy /docker-compose upECTyper:
PanPredic:
Imagem Docker para Conda:
Comparando diferentes grupos populacionais:
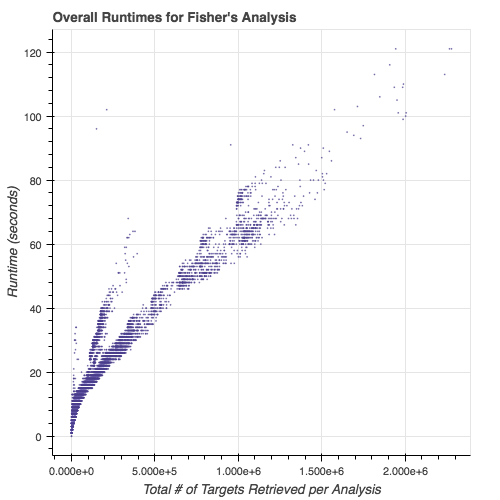
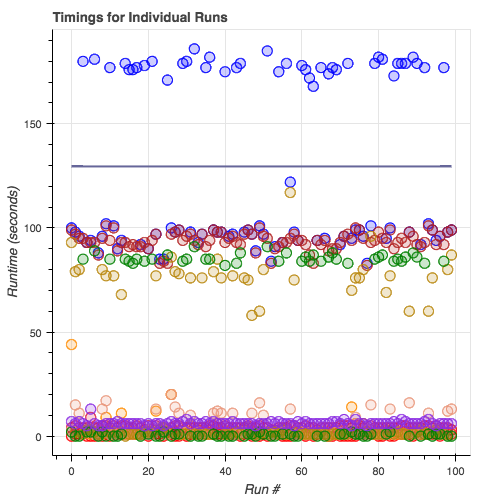
Tempos de execução de módulos de subtipagem:
cd app/python -m modules/savvy -i tests/ecoli/GCA_001894495.1_ASM189449v1_genomic.fna onde o argumento após o -i é o arquivo do seu genoma (FASTA). 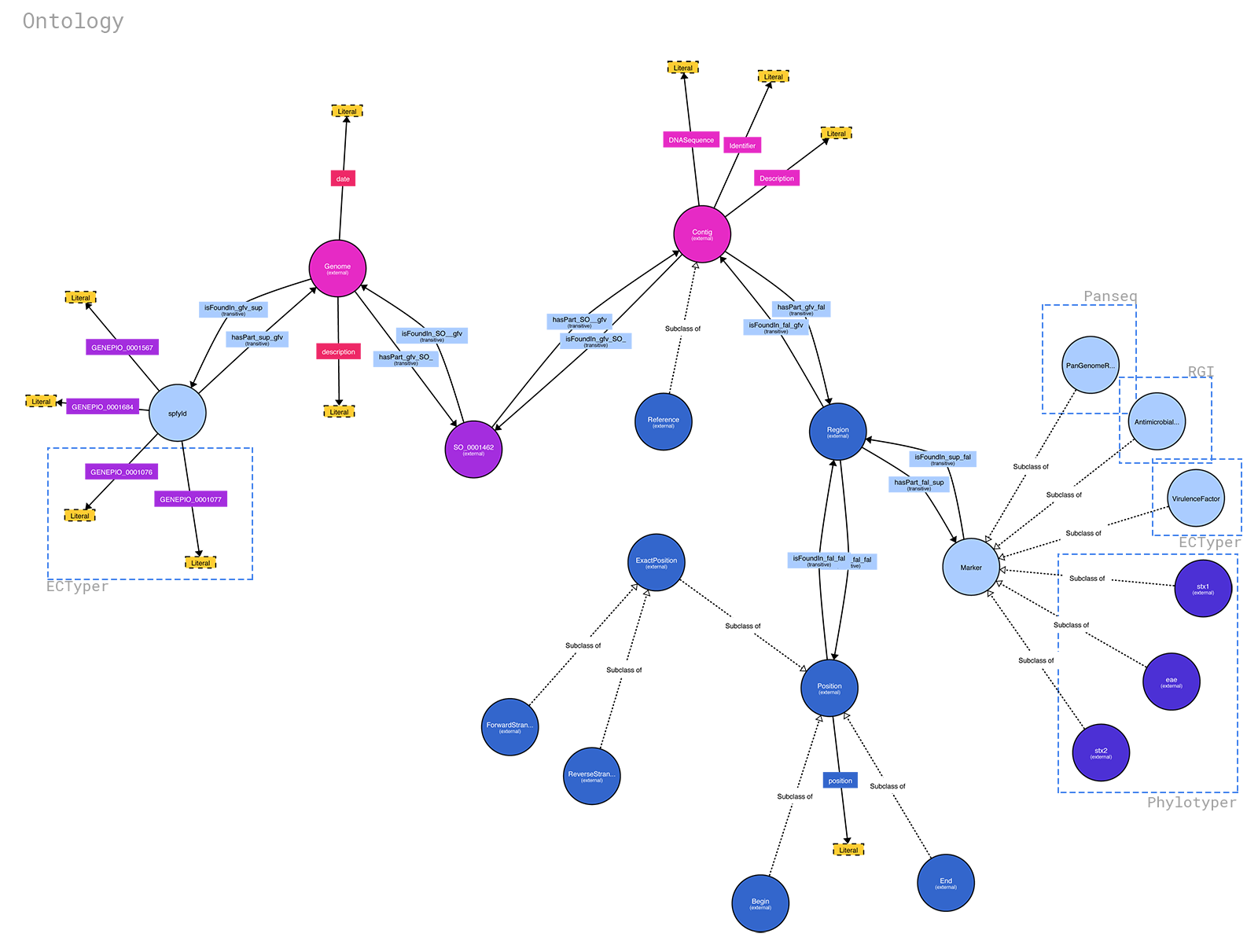
A ontologia para spfy está disponível em: https://raw.githubusercontent.com/superphy/backend/master/app/scripts/spfy_ontology.ttl Foi gerada usando https://raw.githubusercontent.com/superphy/backend/master /app/scripts/generate_ontology.py com funções compartilhadas do código backend de spfy . Se desejar executá-lo, faça: 1. cd app/ 2. python -m scripts/generate_ontology que colocará a ontologia em app/
Você pode gerar um diagrama bonito a partir do arquivo .ttl usando http://www.visualdataweb.de/webvowl/
Observação
atualmente configurado apenas para arquivos .fna
Você pode ignorar o site front-end e ainda enfileirar trabalhos de subtipagem:
/datastore nos contêineres.Por exemplo, se você mantiver seus arquivos em
/home/bob/ecoli-genomes/, você editará o arquivodocker-compose.ymle substituirá:volumes : - /datastorecom:
volumes : - /home/bob/ecoli-genomes:/datastore
docker-compose down docker-compose up -d
docker exec -it backend_webserver_1 sh python -m scripts/sideload exit
Observe que os resíduos podem ser criados na sua pasta genoma.

| Dock er Image | Portas | Nome | Descrição |
|---|---|---|---|
| back-end- rq | 80/tcp, 443/tcp | back-end_trabalhador_1 | os principais trabalhadores da fila de redis |
| back end-rq-b laze gráfico h | 80/tcp, 443/tcp | back end_wor ker-blaz egra ph-i ds_ 1 | esta mão mostra a proporção do gene spfy ID para o banco de dados blaz egra ph |
| back-end | 0,0. 0,0: 8000 ->80 /tcp, 443/tcp | back end_web -ngi nx-u wsgi _1 | o back-end do flask que lida com as tarefas de enfileiramento |
| super rphy /bla zegr aph: 2.1. 4-in fere ncin g | 0,0. 0,0: 8080 ->80 80/t cp | back end_bla zegr aph_1 | Banco de dados Blaz egra ph |
| redis:3. 2 | 6379 /tcp | back end_red é_ 1 | Base de dados do Redi |
| reac tapp | 0,0. 0,0: 8090 ->50 00/t cp | back end_rea ctap p_1 | frente t-en d para spfy |
A imagem superphy/backend-rq:2.0.0 é escalonável : você pode criar quantas instâncias precisar/ter capacidade de processamento. A imagem é responsável por escutar a fila multiples (12 trabalhadores) que trata da maior parte das tarefas, incluindo chamadas RGI . Ele também escuta a fila singles (1 trabalhador) que executa ECTyper . Isso é feito porque RGI é a parte mais lenta da equação. A gestão dos trabalhadores é feita pelo supervisor .
A imagem superphy/backend-rq-blazegraph:2.0.0 não é escalável: ela é responsável por consultar o banco de dados Blazegraph em busca de entradas duplicadas e por atribuir IDs spfy em ordem sequencial . Suas funções são mantidas o mínimo possível para melhorar o desempenho (já que a geração de ID é o único gargalo em pipelines paralelos); as comparações são feitas por hashes sha1 dos arquivos enviados e os não duplicados têm seus IDs reservados vinculando o ID spfy gerado ao hash do arquivo. A gestão dos trabalhadores é feita pelo supervisor .
O superphy/backend:2.0.0 que executa os endpoints Flask usa supervisor para gerenciar processos internos: nginx , uWsgi .
database['blazegraph_url'] em /app/config.py As etapas necessárias para adicionar novos módulos estão documentadas no Guia do desenvolvedor.