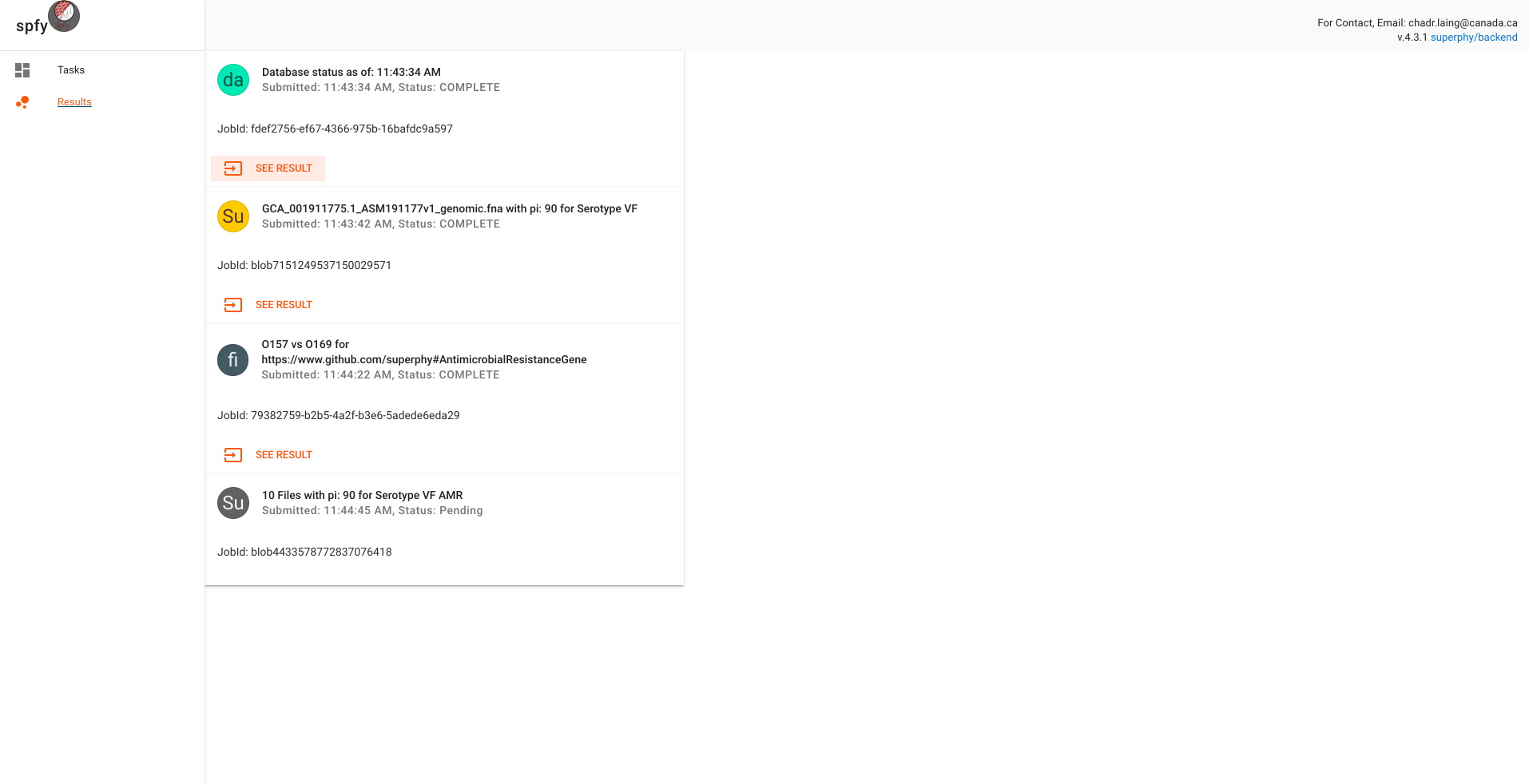
spfy : Plattform zur Vorhersage von Subtypen aus E. coli-Genomsequenzen und zur Erstellung von Diagrammdaten für bevölkerungsweite Vergleichsanalysen.
Veröffentlicht als: Le, KK, Whiteside, MD, Hopkins, JE, Gannon, VPJ, Laing, CR spfy : eine integrierte Diagrammdatenbank zur Echtzeitvorhersage bakterieller Phänotypen und nachgelagerter Vergleichsanalysen. Datenbank (2018) Bd. 2018: Artikel-ID bay086; doi:10.1093/database/bay086
Live: https://lfz.corefacility.ca/superphy/spfy/
git clone --recursive https://github.com/superphy/spfy.gitcd spfy /docker-compose upECTyper:
PanPredic:
Docker-Image für Conda:
Vergleich verschiedener Bevölkerungsgruppen:
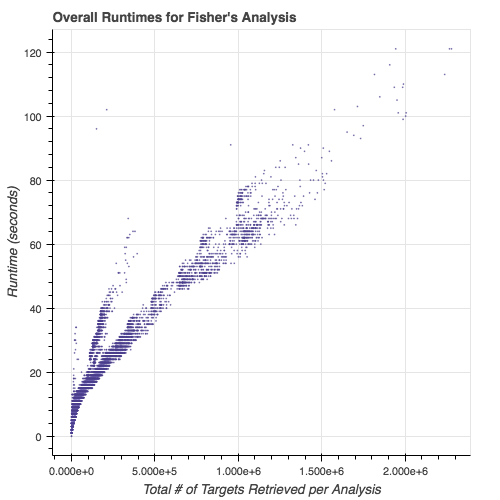
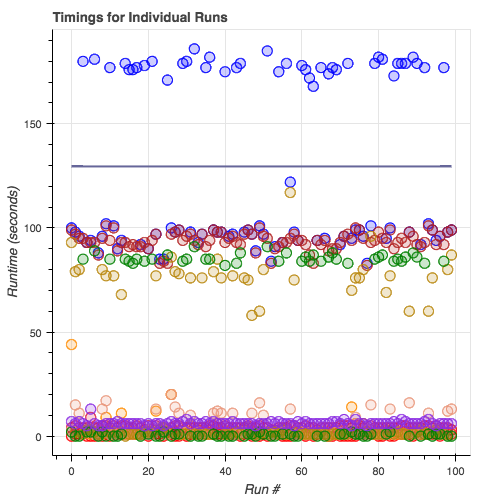
Laufzeiten von Subtypisierungsmodulen:
cd app/python -m modules/savvy -i tests/ecoli/GCA_001894495.1_ASM189449v1_genomic.fna , wobei das Argument nach -i Ihre Genomdatei (FASTA) ist. 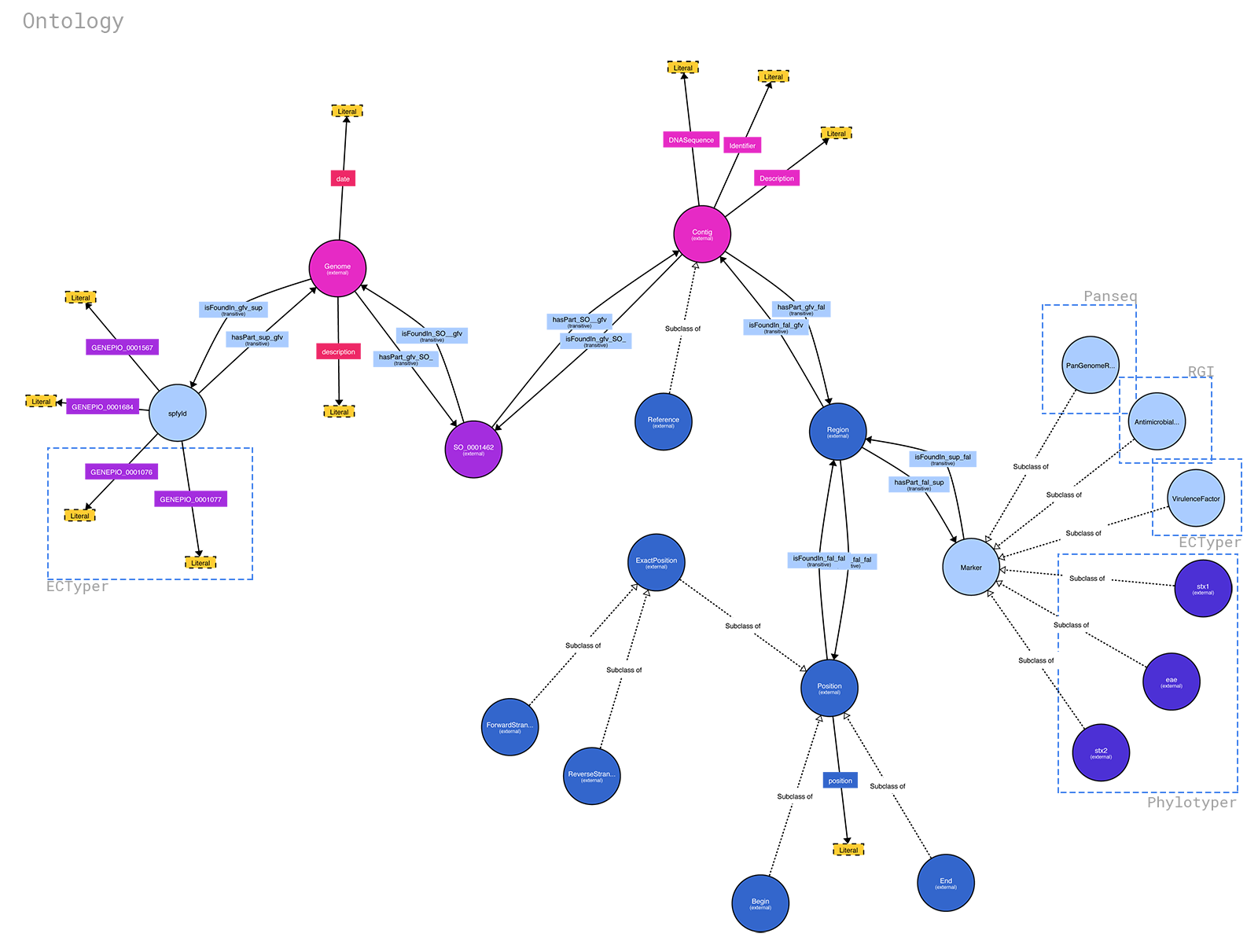
Die Ontologie für spfy ist verfügbar unter: https://raw.githubusercontent.com/superphy/backend/master/app/scripts/spfy_ontology.ttl. Sie wurde mit https://raw.githubusercontent.com/superphy/backend/master generiert /app/scripts/generate_ontology.py mit gemeinsamen Funktionen aus dem Backend-Code von spfy . Wenn Sie es ausführen möchten, gehen Sie wie folgt vor: 1. cd app/ 2. python -m scripts/generate_ontology wodurch die Ontologie in app/ abgelegt wird
Mit http://www.visualdataweb.de/webvowl/ können Sie aus der .ttl-Datei ein hübsches Diagramm generieren.
Notiz
Derzeit nur für .fna-Dateien eingerichtet
Sie können die Front-End-Website umgehen und dennoch Untertypisierungsjobs in die Warteschlange stellen, indem Sie:
/datastore in den Containern ein.Wenn Sie Ihre Dateien beispielsweise unter
/home/bob/ecoli-genomes/aufbewahren, würden Sie die Dateidocker-compose.ymlbearbeiten und Folgendes ersetzen:volumes : - /datastoremit:
volumes : - /home/bob/ecoli-genomes:/datastore
docker-compose down docker-compose up -d
docker exec -it backend_webserver_1 sh python -m scripts/sideload exit
Beachten Sie, dass möglicherweise Rückstände in Ihrem Genomordner erstellt werden.

| Dock er Image e | Hafen s | Name s | Beschreibung |
|---|---|---|---|
| Back-End- rq | 80/t cp, 443/tcp | zurück end_work_1 | Die wichtigsten Redis-Warteschlangenarbeiter |
| Backend-rq-b laze grap h | 80/t cp, 443/tcp | back end_wor ker-blaz egra ph-i ds_ 1 | Dies führt zur Generierung spfy -ID für die Blaz-Egra-Datenbank |
| Backend | 0,0. 0.0: 8000 ->80 /tcp , 443/ tcp | zurück end_web -ngi nx-u wsgi _1 | Das Flask-Backend, das die Abfrageaufgaben übernimmt |
| super rphy /bla zegr aph: 2.1. 4-Zoll-Fertigstellung | 0,0. 0,0: 8080 ->80 80/t cp | zurück end_bla zegr aph_1 | Blaz Egra ph-Datenbank |
| redi s:3. 2 | 6379 /tcp | back end_red ist_ 1 | Redis Datenbank |
| reac tap | 0,0. 0,0: 8090 ->50 00/t cp | back end_rea ctap p_1 | von vorn bis zum spfy |
Das superphy/backend-rq:2.0.0 -Image ist skalierbar : Sie können so viele Instanzen erstellen, wie Sie Rechenleistung benötigen/verfügen. Das Image ist für das Abhören der multiples (12 Worker) verantwortlich, die die meisten Aufgaben, einschließlich RGI -Aufrufe, erledigt. Es überwacht auch die singles -Warteschlange (1 Worker), die ECTyper ausführt. Dies geschieht, da RGI der langsamste Teil der Gleichung ist. Die Mitarbeiterführung erfolgt durch supervisor .
Das Bild superphy/backend-rq-blazegraph:2.0.0 ist nicht skalierbar: Es ist dafür verantwortlich, die Blazegraph-Datenbank nach doppelten Einträgen abzufragen und spfy IDs in sequentieller Reihenfolge zuzuweisen. Seine Funktionen werden so minimal wie möglich gehalten, um die Leistung zu verbessern (da die ID-Generierung den einzigen Engpass in ansonsten parallelen Pipelines darstellt); Vergleiche werden durch SHA1-Hashes der übermittelten Dateien durchgeführt und die IDs von Nicht-Duplikaten werden reserviert, indem die generierte spfy -ID mit dem Datei-Hash verknüpft wird. Die Mitarbeiterführung erfolgt durch supervisor .
Das superphy/backend:2.0.0 , das die Flask-Endpunkte ausführt, verwendet supervisor um innere Prozesse zu verwalten: nginx , uWsgi .
database['blazegraph_url'] in /app/config.py Die zum Hinzufügen neuer Module erforderlichen Schritte sind im Entwicklerhandbuch dokumentiert.