lingress
v3.0.7
Lingress 프로젝트는 일 변량 선형 회귀 모델을 사용하여 NMR (Nuclear Magnetic Resonance) 데이터 세트 분석을위한 간소화 된 파이프 라인을 개발하기위한 이니셔티브입니다. 이 패키지는 일반 최소 제곱 (OLS) 방법을 통한 선형 회귀 분석의 실행을 포함하고 결과 데이터의 시각적 해석을 제공합니다. 특히, 분석 범위에서 모든 NMR 피크의 p- 값을 포함합니다.
기능적 으로이 프로그램은 선형 회귀 적용을 통해 대사 프로파일 모델에 맞게 노력합니다. 설계 및 기능은 대사 연구 영역에서 심층적이고 미묘한 데이터 분석을위한 강력한 도구를 제공합니다.
pip install lingress #Example data
import numpy as np
from lingress import pickie_peak
import pandas as pd
df = pd . read_csv ( "https://raw.githubusercontent.com/aeiwz/example_data/main/dataset/Example_NMR_data.csv" )
spectra = df . iloc [:, 1 :]
ppm = spectra . columns . astype ( float ). to_list ()
#defind plot data and run UI
pickie_peak ( spectra = spectra , ppm = ppm ). run_ui ()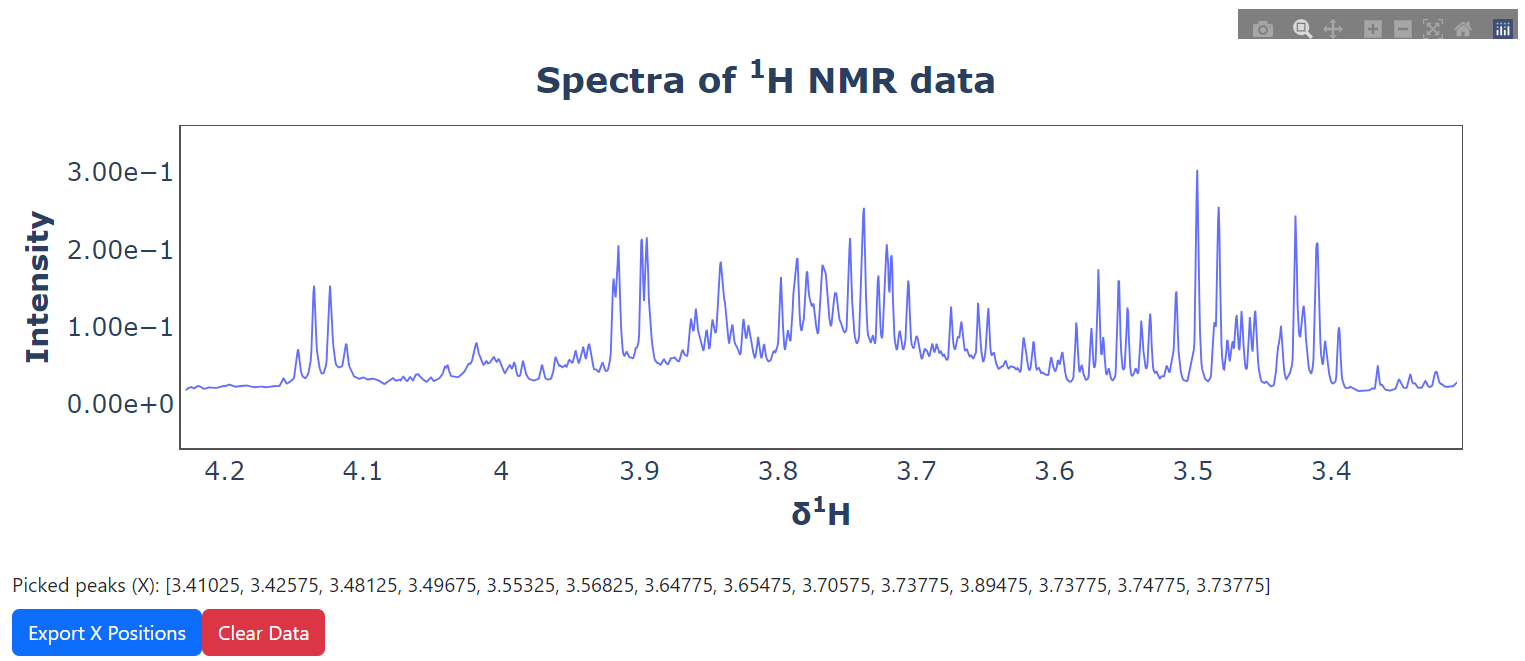
import pandas as pd
from lingress import lin_regression
df = pd . read_csv ( "https://raw.githubusercontent.com/aeiwz/example_data/main/dataset/Example_NMR_data.csv" )
X = df . iloc [:, 1 :]
ppm = spectra . columns . astype ( float ). to_list ()
y = df [ 'Group' ]
mod = lin_regression ( x = X , target = y , label = y , features_name = ppm , adj_method = 'fdr_bh' )
mod . create_dataset ()
mod . fit_model () mod . spec_uniplot ()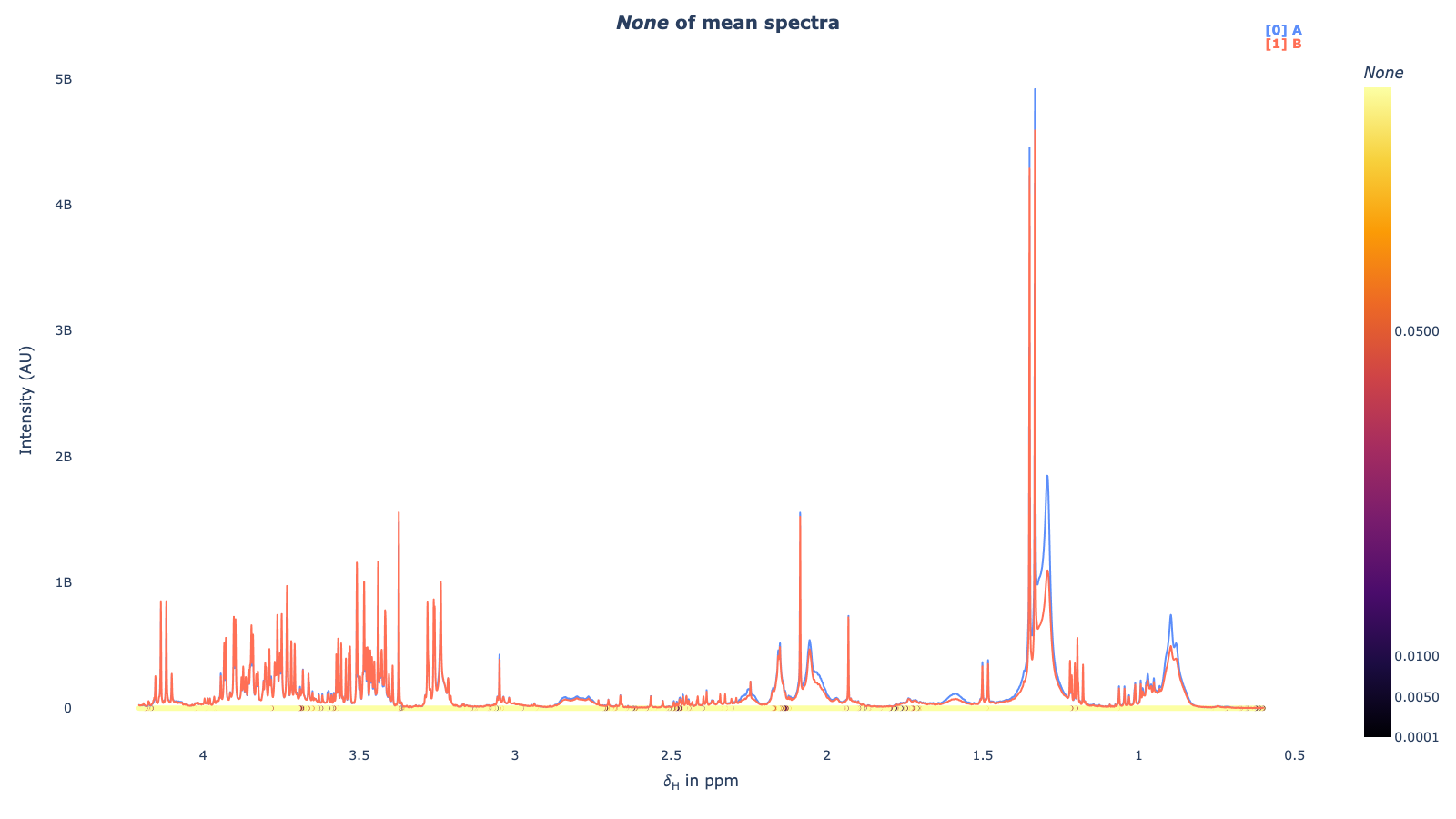
mod . volcano_plot ()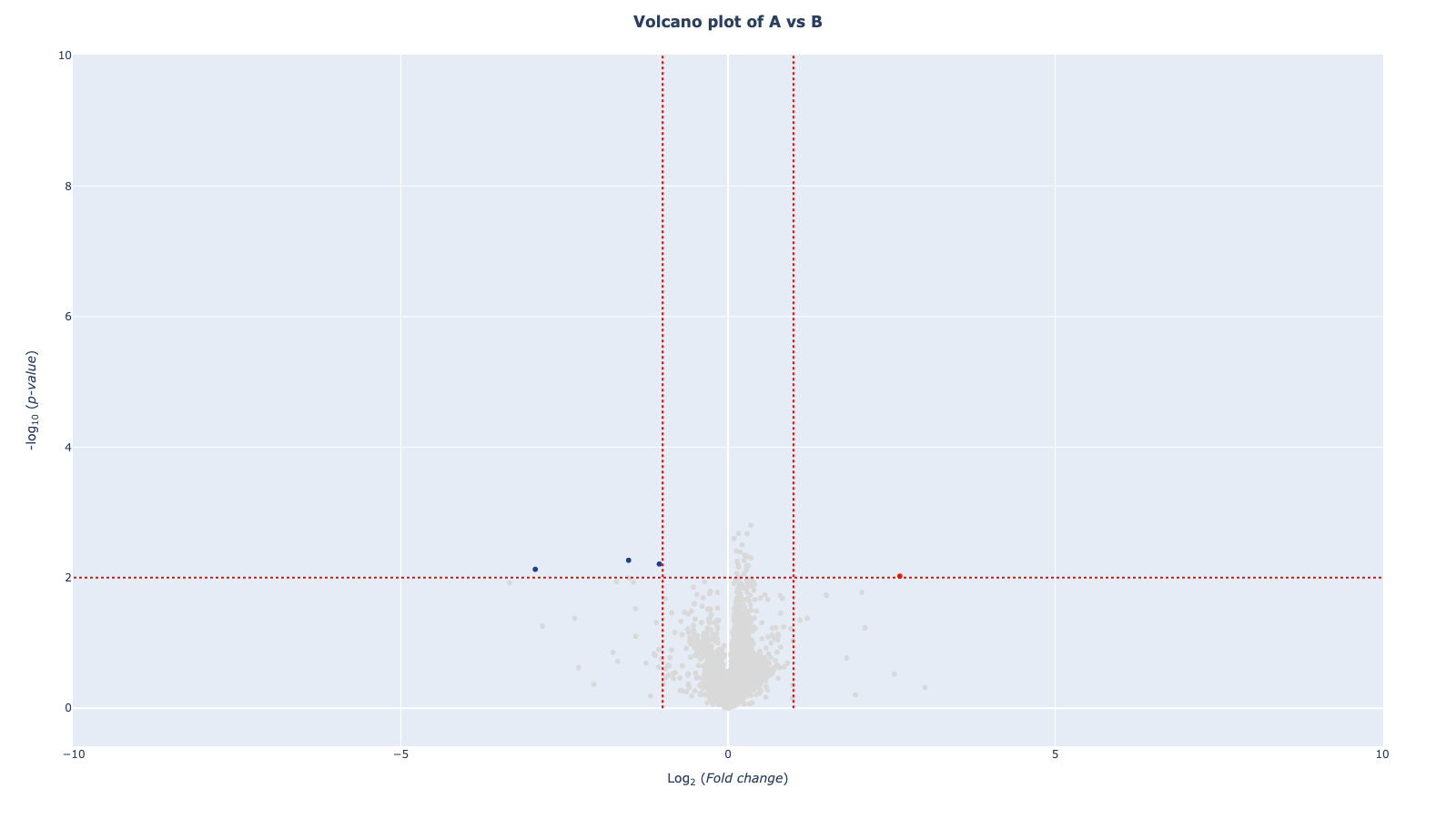
mod . resampling ( n_jobs = - 1 , n_boots = 100 , adj_method = 'fdr_bh' ) [Parallel(n_jobs=-1)]: Using backend LokyBackend with 8 concurrent workers.
[Parallel(n_jobs=-1)]: Done 6 tasks | elapsed: 3.7s
[Parallel(n_jobs=-1)]: Done 60 tasks | elapsed: 6.7s
[Parallel(n_jobs=-1)]: Done 150 tasks | elapsed: 11.2s
[Parallel(n_jobs=-1)]: Done 276 tasks | elapsed: 17.8s
...
[Parallel(n_jobs=-1)]: Done 6486 tasks | elapsed: 5.6min
[Parallel(n_jobs=-1)]: Done 7188 tasks | elapsed: 6.1min
[Parallel(n_jobs=-1)]: Done 7211 out of 7211 | elapsed: 6.1min finished
mod . resampling_df ()| p- 값 | STD P- 값 | 베타 계수 | STD 베타 | 평균 p- 값 (F- 테스트) | STD P- 값 (F- 테스트) | 평균 R- 제곱 | std r-square | R2 | STD R-Square 조정 | q_value |
|---|---|---|---|---|---|---|---|---|---|---|
| 0.60075 | 3.575454E-03 | 1.610523E-02 | 3.673194E+06 | 502596.020205 | 0.434302 | 0.276809 | 0.138650 | 0.156244 | 0.030981 | 4.012856E-03 |
| 0.60125 | 2.327687E-04 | 6.418472E-04 | 4.208365E+06 | 638734.119190 | 난 | 난 | 0.160225 | 0.175463 | 0.056503 | 3.531443E-04 |
| 0.60175 | 1.511846E-04 | 3.690541E-04 | 4.776924E+06 | 582175.023885 | 0.272894 | 0.258094 | 0.250765 | 0.204542 | 0.157111 | 2.443829E-04 |
| 0.60225 | 2.724337E-04 | 7.138873E-04 | 4.450884E+06 | 624407.676115 | 0.132108 | 0.188570 | 0.379931 | 0.198055 | 0.302422 | 4.037237E-04 |
| 0.60275 | 2.271675E-04 | 5.238926E-04 | 3.596622E+06 | 643161.588649 | 0.030732 | 0.056968 | 0.558447 | 0.158948 | 0.503253 | 3.458106E-04 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 4.20375 | 2.542707E-09 | 1.077483E-08 | 2.231841E+07 | 479783.299949 | 난 | 난 | 0.099255 | 0.130321 | -0.010838 | 4.472063E-08 |
| 4.20425 | 4.727199E-10 | 1.269310E-09 | 2.201865E+07 | 631164.491894 | 0.420162 | 0.308196 | 0.163733 | 0.184153 | 0.059199 | 1.940690E-08 |
| 4.20475 | 1.710447E-09 | 4.659603E-09 | 2.285026E+07 | 721568.566334 | 난 | 난 | 0.100927 | 0.138527 | -0.010207 | 3.595928E-08 |
| 4.20525 | 1.043658E-08 | 9.454456E-08 | 2.449345E+07 | 287615.593479 | 0.310386 | 0.301403 | 0.263740 | 0.245996 | 0.171707 | 1.084412E-07 |
| 4.20575 | 1.606948E-08 | 1.123188E-07 | 2.621135E+07 | 246414.620688 | 0.242344 | 0.257300 | 0.299881 | 0.244772 | 0.212366 | 1.457572E-07 |