Le projet Lingress est une initiative visant à développer un pipeline rationalisé pour l'analyse des ensembles de données de résonance magnétique nucléaire (RMN), en utilisant un modèle de régression linéaire univariée. Ce package englobe l'exécution de l'analyse de régression linéaire via la méthode des moindres carrés ordinaires (OLS) et fournit des interprétations visuelles des données résultantes. Il comprend notamment les valeurs p de tous les pics RMN dans sa portée analytique.
Fonctionnellement, ce programme s'efforce d'adapter un modèle de profils métaboliques grâce à l'application de la régression linéaire. Sa conception et ses capacités présentent un outil robuste pour l'analyse approfondie et nuancée des données dans le domaine des études métaboliques.
pip install lingress #Example data
import numpy as np
from lingress import pickie_peak
import pandas as pd
df = pd . read_csv ( "https://raw.githubusercontent.com/aeiwz/example_data/main/dataset/Example_NMR_data.csv" )
spectra = df . iloc [:, 1 :]
ppm = spectra . columns . astype ( float ). to_list ()
#defind plot data and run UI
pickie_peak ( spectra = spectra , ppm = ppm ). run_ui ()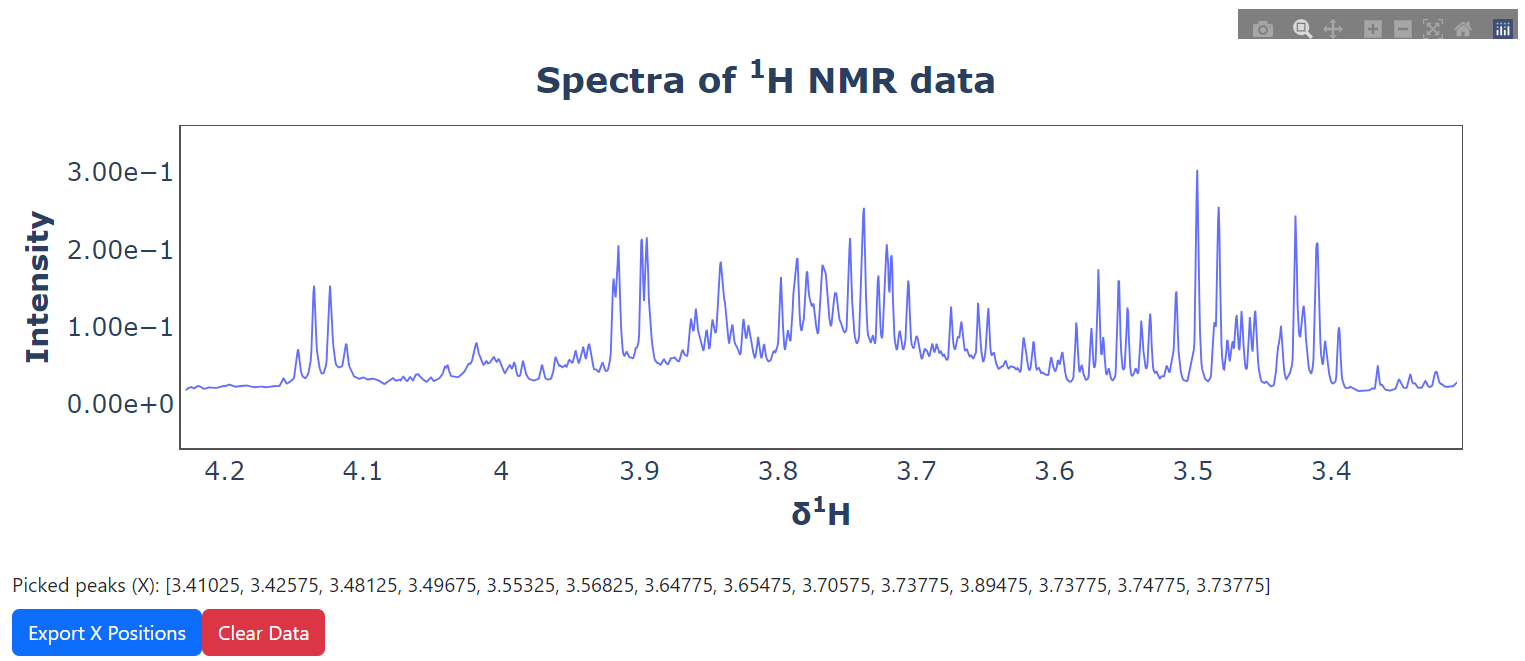
import pandas as pd
from lingress import lin_regression
df = pd . read_csv ( "https://raw.githubusercontent.com/aeiwz/example_data/main/dataset/Example_NMR_data.csv" )
X = df . iloc [:, 1 :]
ppm = spectra . columns . astype ( float ). to_list ()
y = df [ 'Group' ]
mod = lin_regression ( x = X , target = y , label = y , features_name = ppm , adj_method = 'fdr_bh' )
mod . create_dataset ()
mod . fit_model () mod . spec_uniplot ()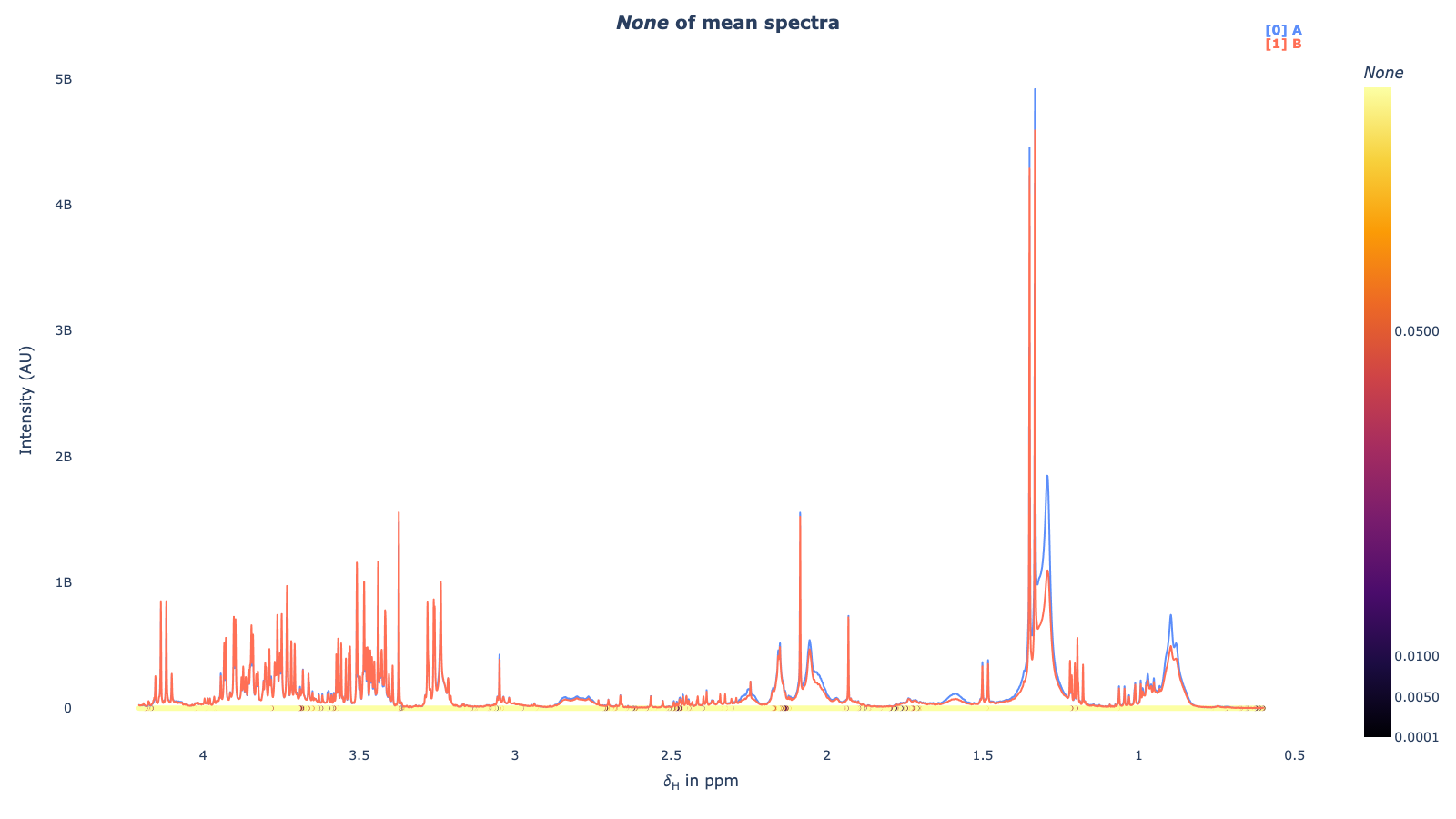
mod . volcano_plot ()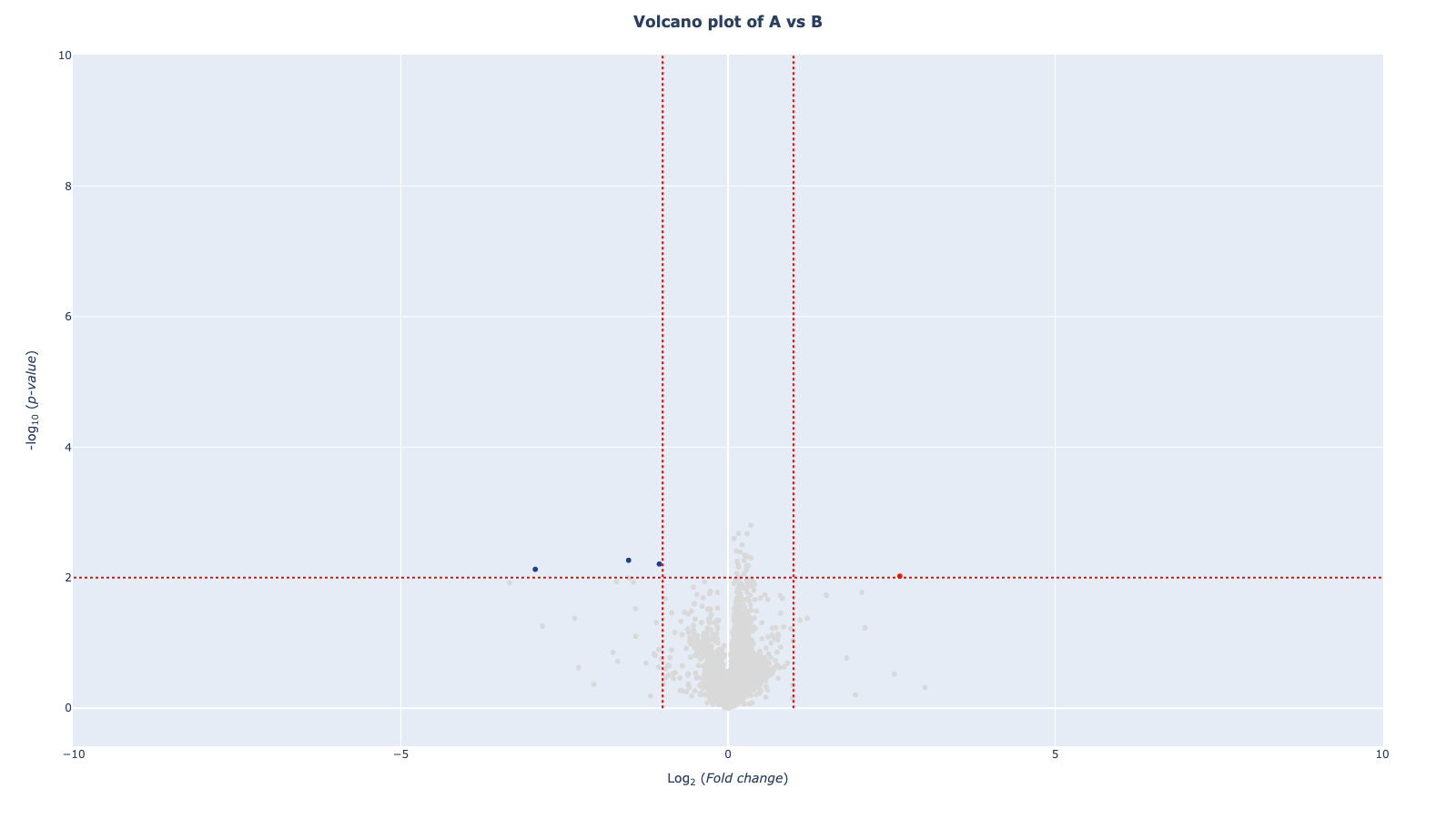
mod . resampling ( n_jobs = - 1 , n_boots = 100 , adj_method = 'fdr_bh' ) [Parallel(n_jobs=-1)]: Using backend LokyBackend with 8 concurrent workers.
[Parallel(n_jobs=-1)]: Done 6 tasks | elapsed: 3.7s
[Parallel(n_jobs=-1)]: Done 60 tasks | elapsed: 6.7s
[Parallel(n_jobs=-1)]: Done 150 tasks | elapsed: 11.2s
[Parallel(n_jobs=-1)]: Done 276 tasks | elapsed: 17.8s
...
[Parallel(n_jobs=-1)]: Done 6486 tasks | elapsed: 5.6min
[Parallel(n_jobs=-1)]: Done 7188 tasks | elapsed: 6.1min
[Parallel(n_jobs=-1)]: Done 7211 out of 7211 | elapsed: 6.1min finished
mod . resampling_df ()| Valeur p | Valeur PD PD | Coefficient bêta | STD Beta | VALUE P Moyenne (test F) | Valeur P STD (T-test) | Moyenne R-Square | STD R-Square | R2 | Réglage STD R-Square | Q_value |
|---|---|---|---|---|---|---|---|---|---|---|
| 0,60075 | 3.575454E-03 | 1.610523E-02 | 3.673194E + 06 | 502596.020205 | 0.434302 | 0.276809 | 0.138650 | 0,156244 | 0.030981 | 4.012856E-03 |
| 0,60125 | 2.327687E-04 | 6.418472E-04 | 4.208365E + 06 | 638734.119190 | Nan | Nan | 0.160225 | 0.175463 | 0,056503 | 3.531443E-04 |
| 0,60175 | 1.511846E-04 | 3.690541E-04 | 4.776924E + 06 | 582175.023885 | 0,272894 | 0,258094 | 0.250765 | 0.204542 | 0.157111 | 2.443829E-04 |
| 0,60225 | 2.724337E-04 | 7.138873E-04 | 4.450884E + 06 | 624407.676115 | 0.132108 | 0.188570 | 0,379931 | 0.198055 | 0.302422 | 4.037237E-04 |
| 0,60275 | 2.271675E-04 | 5.238926E-04 | 3.596622E + 06 | 643161.588649 | 0.030732 | 0,056968 | 0,558447 | 0.158948 | 0,503253 | 3.458106E-04 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 4.20375 | 2.542707E-09 | 1.077483E-08 | 2.231841E + 07 | 479783.299949 | Nan | Nan | 0,099255 | 0.130321 | -0.010838 | 4.472063E-08 |
| 4.20425 | 4.727199E-10 | 1.269310E-09 | 2.201865E + 07 | 631164.491894 | 0.420162 | 0.308196 | 0.163733 | 0.184153 | 0,059199 | 1.940690E-08 |
| 4.20475 | 1.710447E-09 | 4.659603E-09 | 2.285026E + 07 | 721568.566334 | Nan | Nan | 0.100927 | 0.138527 | -0.010207 | 3.595928E-08 |
| 4.20525 | 1.043658E-08 | 9.454456E-08 | 2.449345e + 07 | 287615.593479 | 0,310386 | 0.301403 | 0,263740 | 0.245996 | 0.171707 | 1.084412E-07 |
| 4.20575 | 1.606948E-08 | 1.123188E-07 | 2.621135e + 07 | 246414.620688 | 0,242344 | 0,257300 | 0.299881 | 0,244772 | 0.212366 | 1.457572E-07 |