Das Lingress -Projekt ist eine Initiative, die darauf abzielt, eine optimierte Pipeline für die Analyse der NMR -Datensätze (Nuclear Magnetresonanz) unter Verwendung eines univariaten linearen Regressionsmodells zu entwickeln. Dieses Paket umfasst die Ausführung der linearen Regressionsanalyse über die OLS -Methode (Minderster Quadrate) und liefert visuelle Interpretationen der resultierenden Daten. Insbesondere enthält es die P-Werte aller NMR-Peaks in seinem analytischen Bereich.
Funktionell bestrebt dieses Programm, durch die Anwendung der linearen Regression ein Modell von Stoffwechselprofilen zu erreichen. Das Design und seine Fähigkeiten bieten ein robustes Instrument für eingehende und differenzierte Datenanalysen im Bereich der Stoffwechselstudien.
pip install lingress #Example data
import numpy as np
from lingress import pickie_peak
import pandas as pd
df = pd . read_csv ( "https://raw.githubusercontent.com/aeiwz/example_data/main/dataset/Example_NMR_data.csv" )
spectra = df . iloc [:, 1 :]
ppm = spectra . columns . astype ( float ). to_list ()
#defind plot data and run UI
pickie_peak ( spectra = spectra , ppm = ppm ). run_ui ()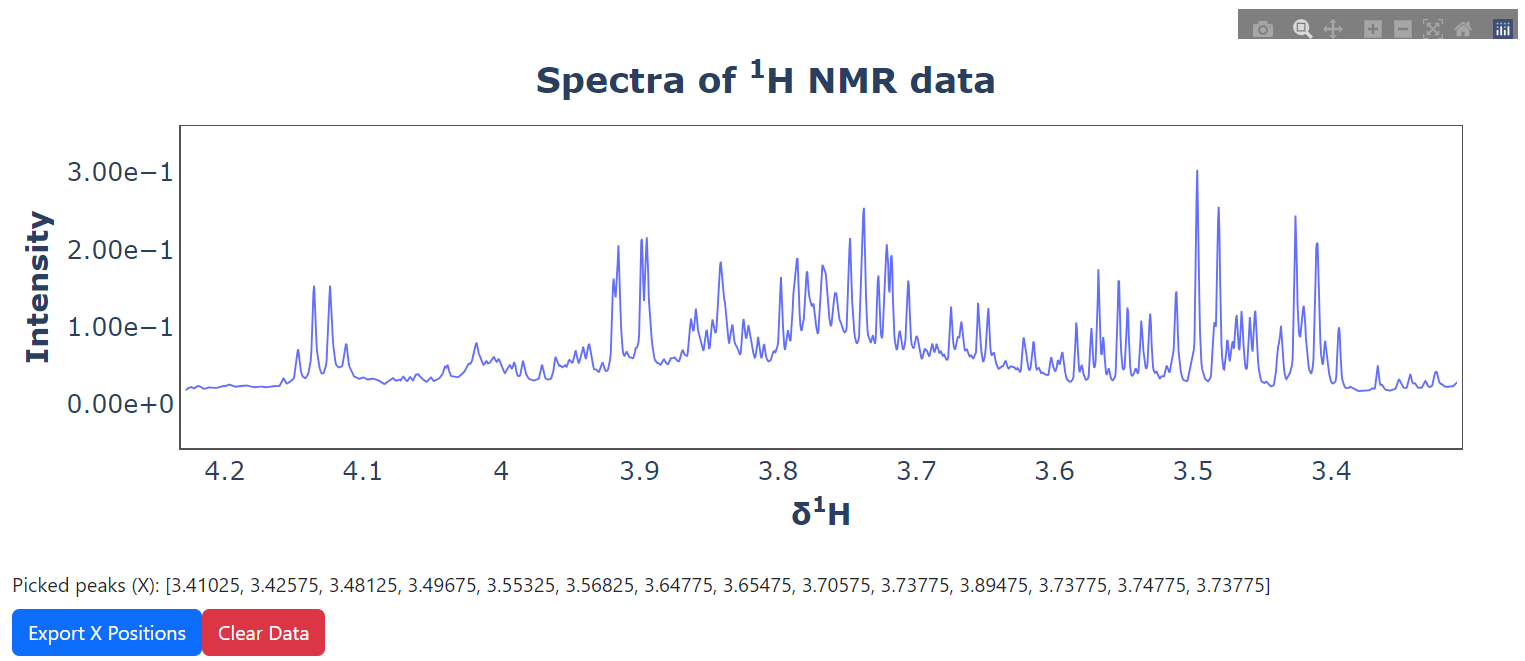
import pandas as pd
from lingress import lin_regression
df = pd . read_csv ( "https://raw.githubusercontent.com/aeiwz/example_data/main/dataset/Example_NMR_data.csv" )
X = df . iloc [:, 1 :]
ppm = spectra . columns . astype ( float ). to_list ()
y = df [ 'Group' ]
mod = lin_regression ( x = X , target = y , label = y , features_name = ppm , adj_method = 'fdr_bh' )
mod . create_dataset ()
mod . fit_model () mod . spec_uniplot ()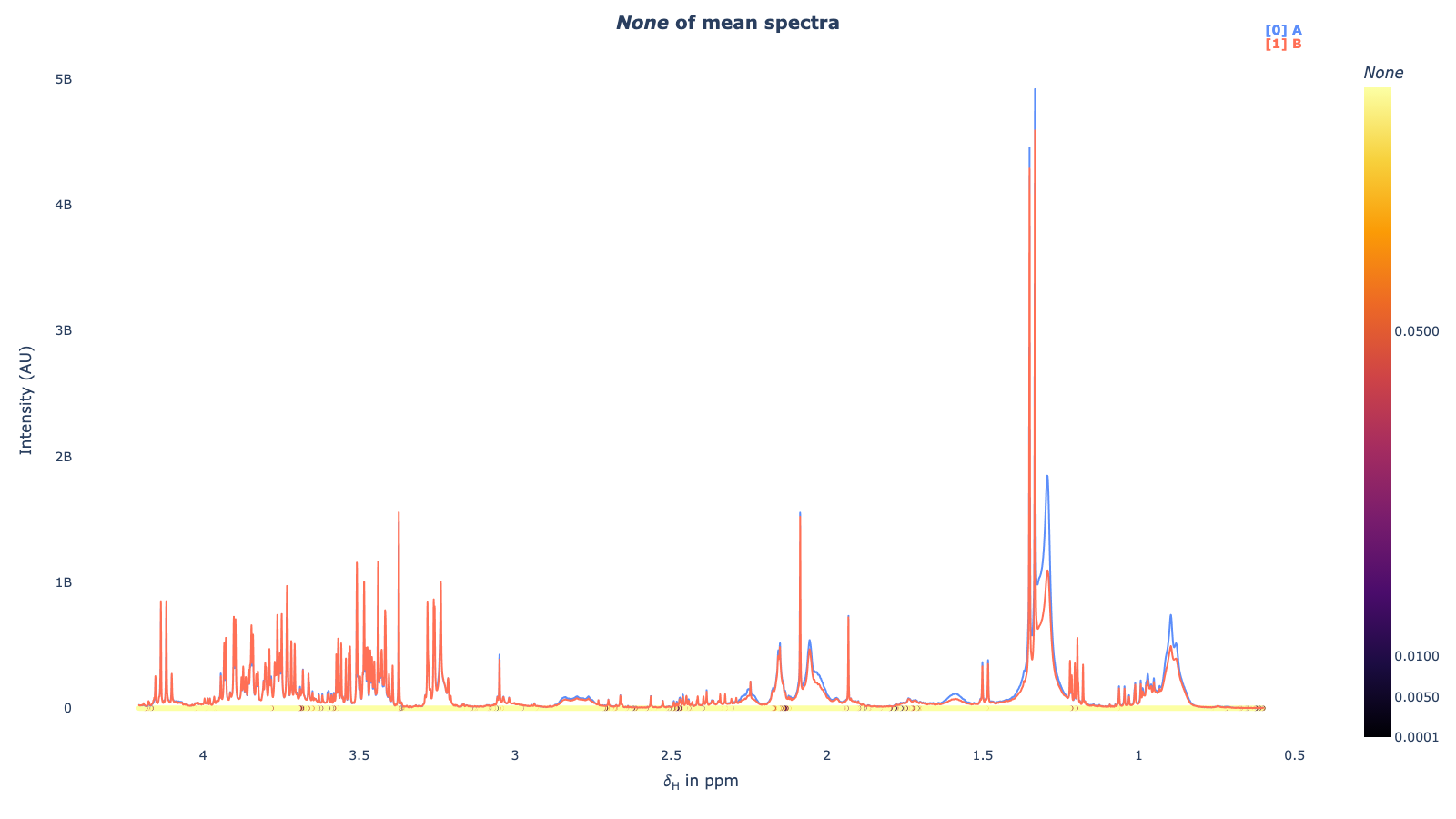
mod . volcano_plot ()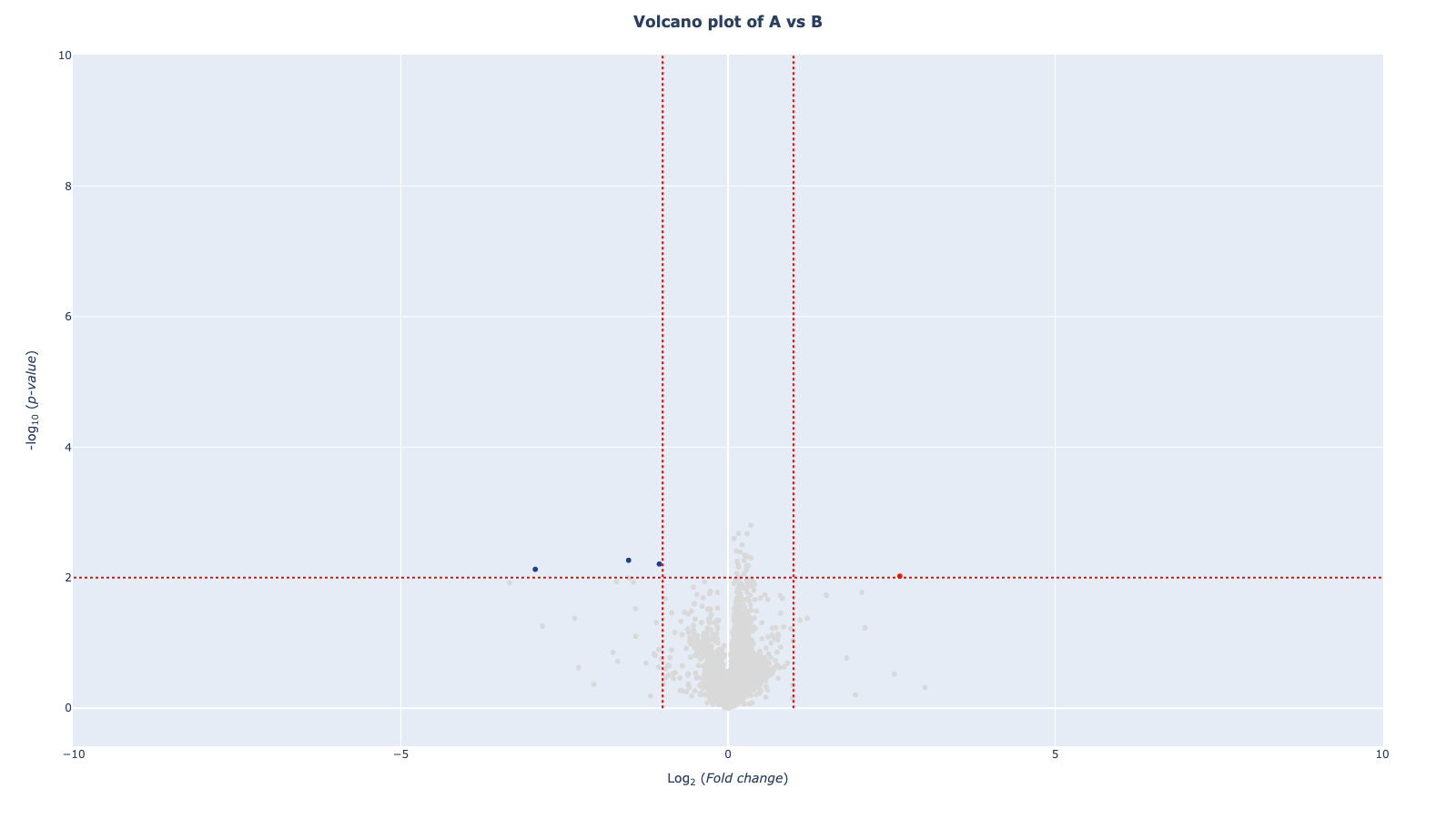
mod . resampling ( n_jobs = - 1 , n_boots = 100 , adj_method = 'fdr_bh' ) [Parallel(n_jobs=-1)]: Using backend LokyBackend with 8 concurrent workers.
[Parallel(n_jobs=-1)]: Done 6 tasks | elapsed: 3.7s
[Parallel(n_jobs=-1)]: Done 60 tasks | elapsed: 6.7s
[Parallel(n_jobs=-1)]: Done 150 tasks | elapsed: 11.2s
[Parallel(n_jobs=-1)]: Done 276 tasks | elapsed: 17.8s
...
[Parallel(n_jobs=-1)]: Done 6486 tasks | elapsed: 5.6min
[Parallel(n_jobs=-1)]: Done 7188 tasks | elapsed: 6.1min
[Parallel(n_jobs=-1)]: Done 7211 out of 7211 | elapsed: 6.1min finished
mod . resampling_df ()| P-Wert | std p-Wert | Beta -Koeffizient | std Beta | Mittlerer p-Wert (F-Test) | STD P-Wert (F-Test) | Mean R-Square | STD R-Quadrat | R2 | STD R-Square-Anpassung | q_value |
|---|---|---|---|---|---|---|---|---|---|---|
| 0,60075 | 3.575454e-03 | 1.610523E-02 | 3.673194e+06 | 502596.020205 | 0,434302 | 0,276809 | 0,138650 | 0,156244 | 0,030981 | 4.012856E-03 |
| 0,60125 | 2.327687E-04 | 6.418472E-04 | 4.208365e+06 | 638734.119190 | Nan | Nan | 0,160225 | 0,175463 | 0,056503 | 3.531443E-04 |
| 0,60175 | 1.511846e-04 | 3.690541E-04 | 4.776924e+06 | 582175.023885 | 0,272894 | 0,258094 | 0,250765 | 0,204542 | 0,157111 | 2.443829E-04 |
| 0,60225 | 2.724337E-04 | 7.138873e-04 | 4.450884e+06 | 624407.676115 | 0,132108 | 0,188570 | 0,379931 | 0,198055 | 0,302422 | 4.037237E-04 |
| 0,60275 | 2.271675e-04 | 5.238926e-04 | 3.596622E+06 | 643161.588649 | 0,030732 | 0,056968 | 0,558447 | 0,158948 | 0,503253 | 3.458106e-04 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 4.20375 | 2.542707E-09 | 1.077483e-08 | 2.231841e+07 | 479783.299949 | Nan | Nan | 0,099255 | 0,130321 | -0.010838 | 4.472063E-08 |
| 4.20425 | 4.727199e-10 | 1.269310e-09 | 2.201865e+07 | 631164.491894 | 0,420162 | 0,308196 | 0,163733 | 0,184153 | 0,059199 | 1.940690E-08 |
| 4.20475 | 1.710447e-09 | 4.659603E-09 | 2.285026e+07 | 721568.566334 | Nan | Nan | 0,100927 | 0,138527 | -0.010207 | 3.595928e-08 |
| 4.20525 | 1.043658e-08 | 9.454456e-08 | 2.449345e+07 | 287615.593479 | 0,310386 | 0,301403 | 0,263740 | 0,245996 | 0,171707 | 1.084412E-07 |
| 4.20575 | 1.606948e-08 | 1.123188e-07 | 2.621135e+07 | 246414.620688 | 0,242344 | 0,257300 | 0,299881 | 0,244772 | 0,212366 | 1.457572E-07 |