skpr
v1.7.0
SKPR ist ein Open -Source -Design der Experimenten -Suite zur Erzeugung und Bewertung optimaler Designs in R. Hier ist eine Stichprobe dessen, was SKPR anbietet:
# To install:
install.packages( " skpr " )
# To install the latest version from Github:
# install.packages("devtools")
devtools :: install_github( " tylermorganwall/skpr " )gen_design() generiert optimale Designs aus einem Kandidatensatz, bei dem ein Modell und die gewünschte Anzahl von Läufen.eval_design() bewertet die Stromversorgung parametrisch für lineare Modelle für normale und splitische Entwürfe.eval_design_mc() bewertet die Leistung mit einer Monte -Carlo -Simulation für lineare und verallgemeinerte lineare Modelle. Diese Funktion unterstützt auch die Berechnung der Leistung für Split-Plot-Designs unter Verwendung von REML.eval_design_survival_mc() bewertet die Leistung mit einer Monte -Carlo -Simulation, sodass der Benutzer einen Punkt angeben kann, an dem die Daten zensiert werden.eval_design_custom_mc() ermöglicht es dem Benutzer, seine eigenen Bibliotheken zu importieren und das von SKPR bereitgestellte Monte -Carlo -Framework zur Berechnung der Stromversorgung zu verwenden.calculate_power_curves() bietet eine Schnittstelle zur Automatisierung der Erzeugung und Bewertung von Designs zur Erstellung von Strom- und Stichprobengröße und Effektgrößenkurven.skprGUI() öffnet die GUI entweder in Rstudio oder in einem externen Browser.Wenn zusätzlich ist, bietet das Paket zwei Funktionen, um gemeinsame Handlungen im Zusammenhang mit Designs zu generieren:
plot_correlations() erzeugt eine Farbkarte mit Korrelationen zwischen Variablen.plot_fds() erzeugt den Bruchteil des Entwurfsraumdiagramms für ein bestimmtes Design.## SKPRGUI
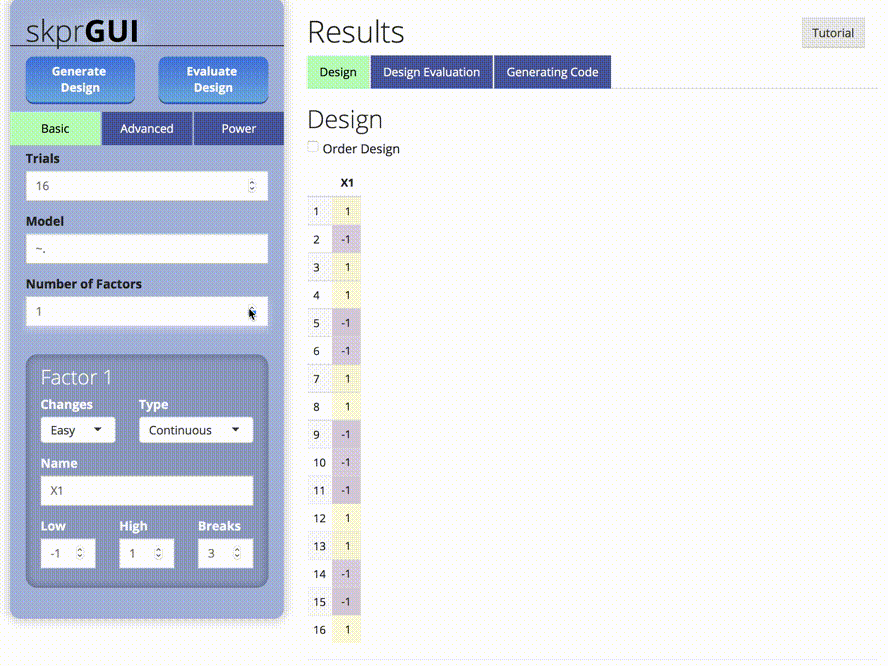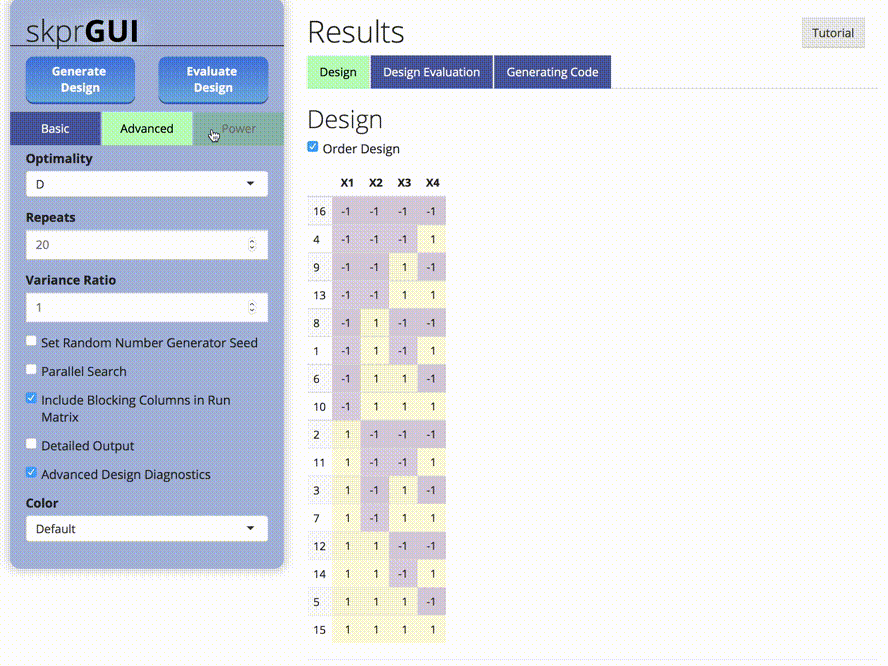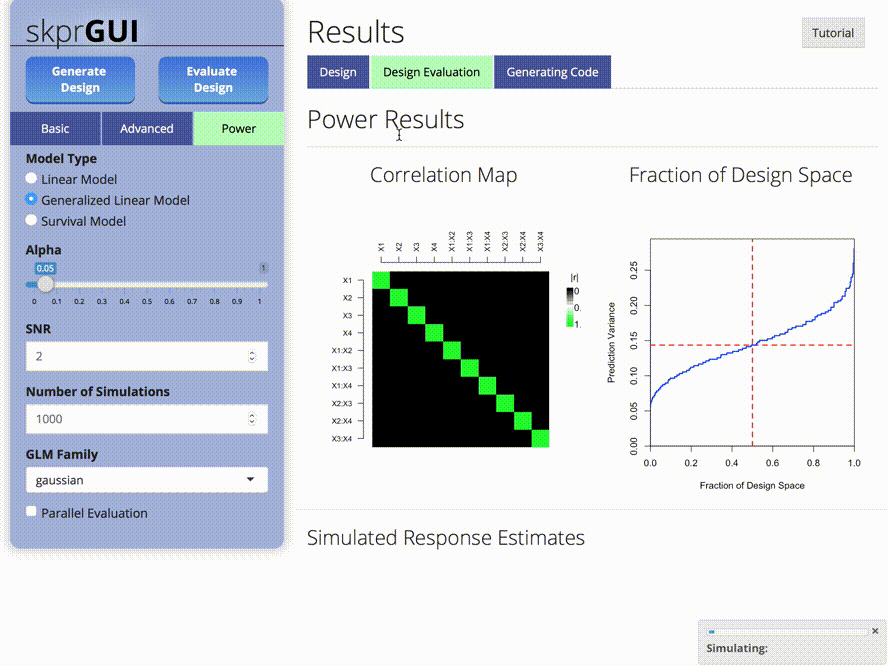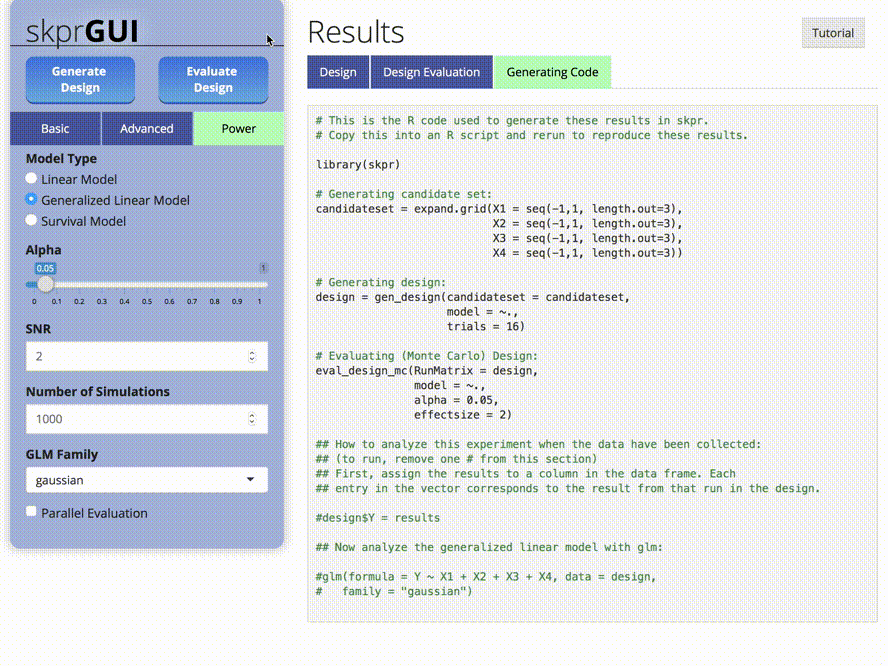
skprGUI() bietet eine grafische Benutzeroberfläche, um auf alle Hauptfunktionen von SKPR zugreifen zu können. Ein interaktives Tutorial wird bereitgestellt, um den Benutzer mit der verfügbaren Funktionalität vertraut zu machen. Geben Sie skprGUI() ein, um zu beginnen. Screenshots:
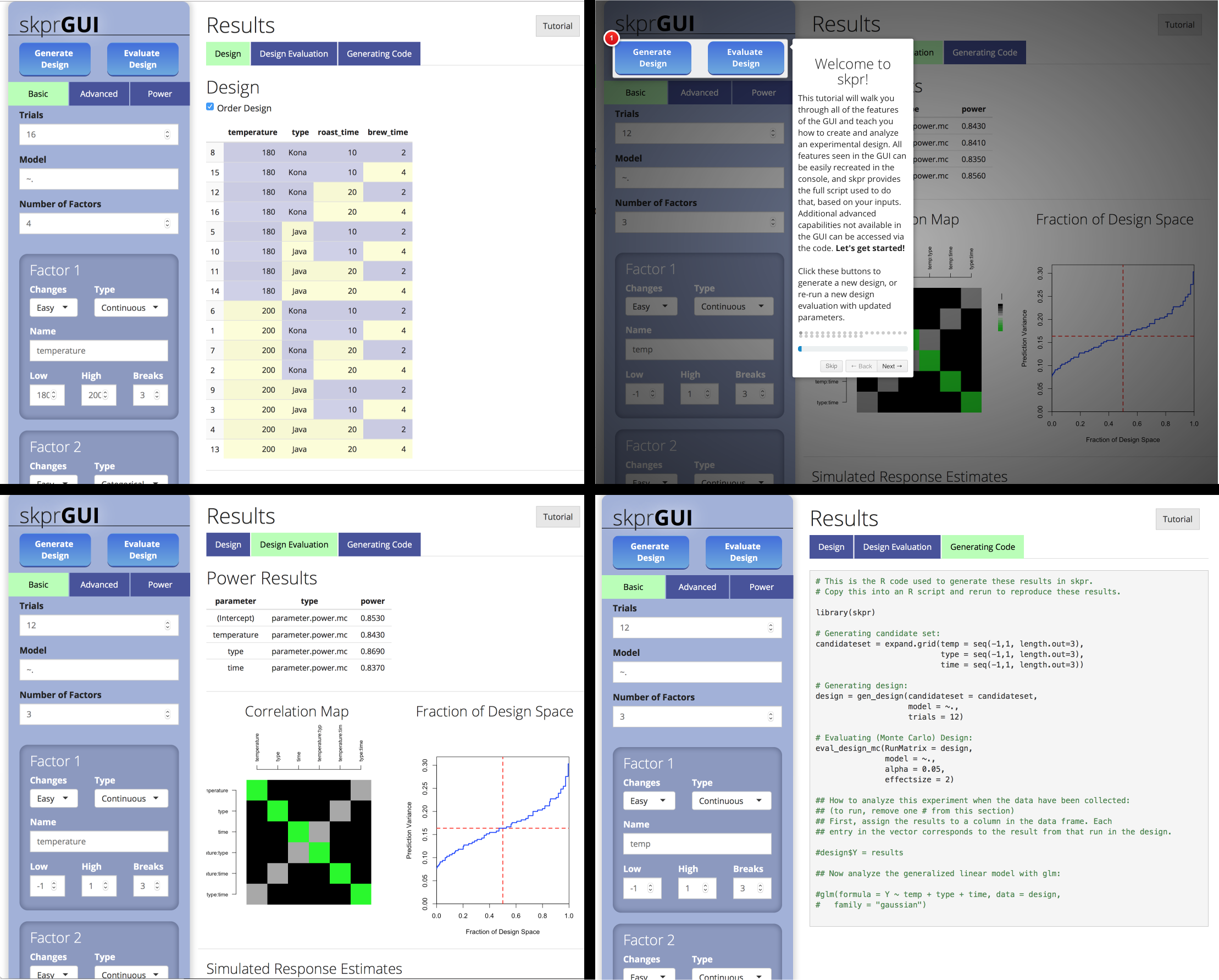
library( skpr )
# Generate a candidate set of all potential design points to be considered in the experiment
# The hypothetical experiment is determining what affects the caffeine content in coffee
candidate_set = expand.grid( temp = c( 80 , 90 , 100 ),
type = c( " Kona " , " Java " ),
beansize = c( " Large " , " Medium " , " Small " ))
candidate_set
# > temp type beansize
# > 1 80 Kona Large
# > 2 90 Kona Large
# > 3 100 Kona Large
# > 4 80 Java Large
# > 5 90 Java Large
# > 6 100 Java Large
# > 7 80 Kona Medium
# > 8 90 Kona Medium
# > 9 100 Kona Medium
# > 10 80 Java Medium
# > 11 90 Java Medium
# > 12 100 Java Medium
# > 13 80 Kona Small
# > 14 90 Kona Small
# > 15 100 Kona Small
# > 16 80 Java Small
# > 17 90 Java Small
# > 18 100 Java Small
# Generate the design (default D-optimal)
design = gen_design( candidateset = candidate_set ,
model = ~ temp + type + beansize ,
trials = 12 )
design
# > temp type beansize
# > 1 80 Java Medium
# > 2 100 Java Large
# > 3 100 Java Small
# > 4 80 Java Large
# > 5 80 Kona Medium
# > 6 80 Kona Small
# > 7 100 Kona Small
# > 8 100 Kona Medium
# > 9 80 Kona Large
# > 10 100 Java Medium
# > 11 100 Kona Large
# > 12 80 Java Small
# Evaluate power for the design with an allowable type-I error of 5% (default)
eval_design( design )
# > parameter type power
# > 1 (Intercept) effect.power 0.8424665
# > 2 temp effect.power 0.8424665
# > 3 type effect.power 0.8424665
# > 4 beansize effect.power 0.5165386
# > 5 (Intercept) parameter.power 0.8424665
# > 6 temp parameter.power 0.8424665
# > 7 type1 parameter.power 0.8424665
# > 8 beansize1 parameter.power 0.5593966
# > 9 beansize2 parameter.power 0.5593966
# > ============Evaluation Info============
# > * Alpha = 0.05 * Trials = 12 * Blocked = FALSE
# > * Evaluating Model = ~temp + type + beansize
# > * Anticipated Coefficients = c(1, 1, 1, 1, -1)
# > * Contrasts = `contr.sum`
# > * Parameter Analysis Method = `lm(...)`
# > * Effect Analysis Method = `car::Anova(fit, type = "III")`
# Evaluate power for the design using a Monte Carlo simulation.
# Here, we set the effect size (here, the signal-to-noise ratio) to 1.5.
eval_design_mc( design , effectsize = 1.5 )
# > parameter type power
# > 1 (Intercept) effect.power.mc 0.600
# > 2 temp effect.power.mc 0.612
# > 3 type effect.power.mc 0.610
# > 4 beansize effect.power.mc 0.316
# > 5 (Intercept) parameter.power.mc 0.600
# > 6 temp parameter.power.mc 0.612
# > 7 type1 parameter.power.mc 0.610
# > 8 beansize1 parameter.power.mc 0.359
# > 9 beansize2 parameter.power.mc 0.354
# > ===========Evaluation Info============
# > * Alpha = 0.05 * Trials = 12 * Blocked = FALSE
# > * Evaluating Model = ~temp + type + beansize
# > * Anticipated Coefficients = c(0.750, 0.750, 0.750, 0.750, -0.750)
# > * Contrasts = `contr.sum`
# > * Parameter Analysis Method = `lm(...)`
# > * Effect Analysis Method = `car::Anova(fit, type = "III")`
# Evaluate power for the design using a Monte Carlo simulation, for a non-normal response.
# Here, we also increase the number of simululations to improve the precision of the results.
eval_design_mc( design , nsim = 5000 , glmfamily = " poisson " , effectsize = c( 2 , 6 ))
# > parameter type power
# > 1 (Intercept) effect.power.mc 0.9968
# > 2 temp effect.power.mc 0.9826
# > 3 type effect.power.mc 0.9832
# > 4 beansize effect.power.mc 0.8502
# > 5 (Intercept) parameter.power.mc 0.9968
# > 6 temp parameter.power.mc 0.9826
# > 7 type1 parameter.power.mc 0.9832
# > 8 beansize1 parameter.power.mc 0.8842
# > 9 beansize2 parameter.power.mc 0.7052
# > ============Evaluation Info============
# > * Alpha = 0.05 * Trials = 12 * Blocked = FALSE
# > * Evaluating Model = ~temp + type + beansize
# > * Anticipated Coefficients = c(1.242, 0.549, 0.549, 0.549, -0.549)
# > * Contrasts = `contr.sum`
# > * Parameter Analysis Method = `glm(..., family = "poisson")`
# > * Effect Analysis Method = `car::Anova(fit, type = "III")`
# skpr was designed to operate with the pipe (|>) in mind.
# Here is an example of an entire design of experiments analysis in three lines:
expand.grid( temp = c( 80 , 90 , 100 ), type = c( " Kona " , " Java " ), beansize = c( " Large " , " Medium " , " Small " )) | >
gen_design( model = ~ temp + type + beansize + beansize : type + I( temp ^ 2 ), trials = 24 , optimality = " I " ) | >
eval_design_mc( detailedoutput = TRUE )
# > parameter type power anticoef alpha glmfamily trials
# > 1 (Intercept) effect.power.mc 0.912 NA 0.05 gaussian 24
# > 2 temp effect.power.mc 0.927 NA 0.05 gaussian 24
# > 3 type effect.power.mc 0.997 NA 0.05 gaussian 24
# > 4 beansize effect.power.mc 0.935 NA 0.05 gaussian 24
# > 5 I(temp^2) effect.power.mc 0.637 NA 0.05 gaussian 24
# > 6 type:beansize effect.power.mc 0.913 NA 0.05 gaussian 24
# > 7 (Intercept) parameter.power.mc 0.912 1 0.05 gaussian 24
# > 8 temp parameter.power.mc 0.927 1 0.05 gaussian 24
# > 9 type1 parameter.power.mc 0.997 1 0.05 gaussian 24
# > 10 beansize1 parameter.power.mc 0.917 1 0.05 gaussian 24
# > 11 beansize2 parameter.power.mc 0.913 -1 0.05 gaussian 24
# > 12 I(temp^2) parameter.power.mc 0.637 1 0.05 gaussian 24
# > 13 type1:beansize1 parameter.power.mc 0.899 1 0.05 gaussian 24
# > 14 type1:beansize2 parameter.power.mc 0.902 -1 0.05 gaussian 24
# > nsim blocking error_adjusted_alpha power_lcb power_ucb
# > 1 1000 FALSE 0.05 0.8927052 0.9288249
# > 2 1000 FALSE 0.05 0.9090858 0.9423464
# > 3 1000 FALSE 0.05 0.9912580 0.9993809
# > 4 1000 FALSE 0.05 0.9178989 0.9494797
# > 5 1000 FALSE 0.05 0.6063275 0.6668632
# > 6 1000 FALSE 0.05 0.8937921 0.9297315
# > 7 1000 FALSE 0.05 0.8927052 0.9288249
# > 8 1000 FALSE 0.05 0.9090858 0.9423464
# > 9 1000 FALSE 0.05 0.9912580 0.9993809
# > 10 1000 FALSE 0.05 0.8981467 0.9333511
# > 11 1000 FALSE 0.05 0.8937921 0.9297315
# > 12 1000 FALSE 0.05 0.6063275 0.6668632
# > 13 1000 FALSE 0.05 0.8786332 0.9169799
# > 14 1000 FALSE 0.05 0.8818715 0.9197225
# > =========================================================Evaluation Info==========================================================
# > * Alpha = 0.05 * Trials = 24 * Blocked = FALSE
# > * Evaluating Model = ~temp + type + beansize + type:beansize + I(temp^2)
# > * Anticipated Coefficients = c(1, 1, 1, 1, -1, 1, 1, -1)
# > * Contrasts = `contr.sum`
# > * Parameter Analysis Method = `lm(...)`
# > * Effect Analysis Method = `car::Anova(fit, type = "III")`
# > * MC Power CI Confidence = 95%