skpr
v1.7.0
SKPR est une conception open source de Suite Experiments pour générer et évaluer des conceptions optimales dans R. Voici un échantillon de ce que SKPR offre:
# To install:
install.packages( " skpr " )
# To install the latest version from Github:
# install.packages("devtools")
devtools :: install_github( " tylermorganwall/skpr " )gen_design() génère des conceptions optimales à partir d'un ensemble de candidats, étant donné un modèle et le nombre souhaité de cycles.eval_design() évalue la puissance paramétrique pour les modèles linéaires, pour les conceptions normales et divisées.eval_design_mc() évalue la puissance avec une simulation Monte Carlo, pour les modèles linéaires linéaires et généralisés. Cette fonction prend également en charge le calcul de la puissance pour les conceptions de places fendues à l'aide de REML.eval_design_survival_mc() Évalue la puissance avec une simulation Monte Carlo, permettant à l'utilisateur de spécifier un point auquel les données sont censurées.eval_design_custom_mc() permet à l'utilisateur d'importer ses propres bibliothèques et d'utiliser le cadre Monte Carlo fourni par SKPR pour calculer la puissance.calculate_power_curves() fournit une interface pour automatiser la génération et l'évaluation des conceptions pour créer des courbes de taille et de taille de la taille de l'échantillon et de l'échantillon.skprGUI() ouvre l'interface graphique dans rstudio ou un navigateur externe.Si en plus, le package propose deux fonctions pour générer des tracés communs liés aux conceptions:
plot_correlations() génère une carte couleur des corrélations entre les variables.plot_fds() génère la fraction du tracé de l'espace de conception pour une conception donnée.## skprgui
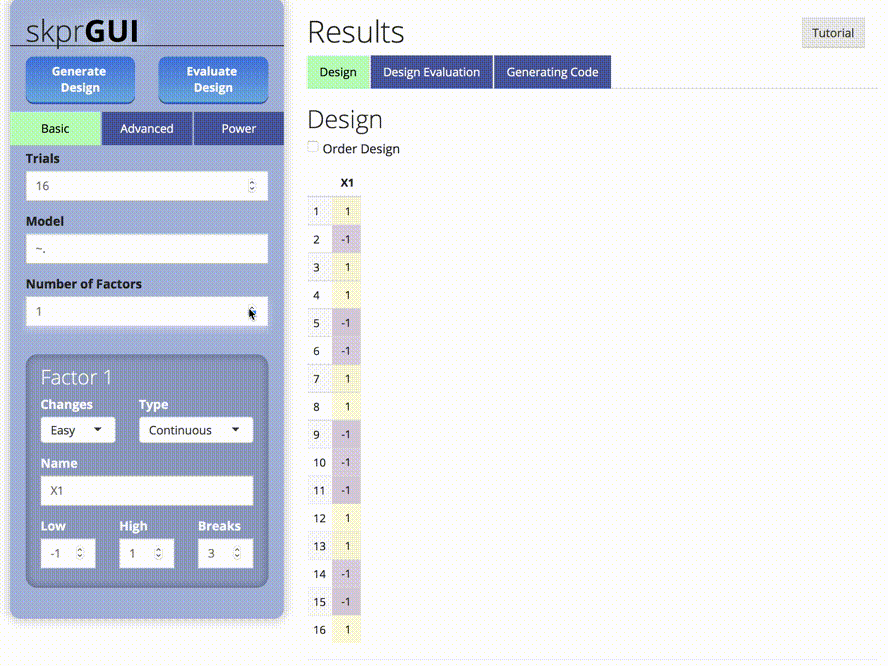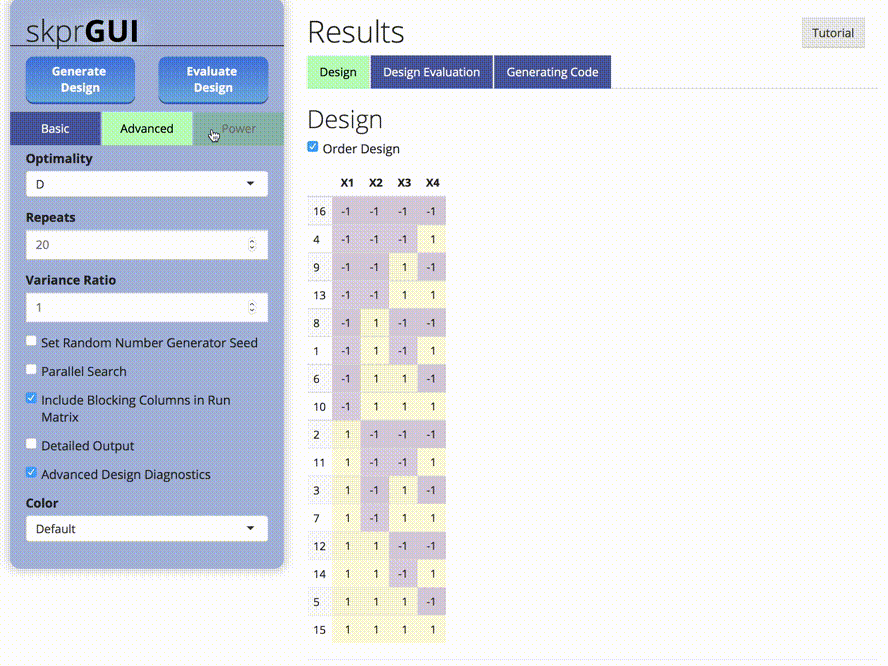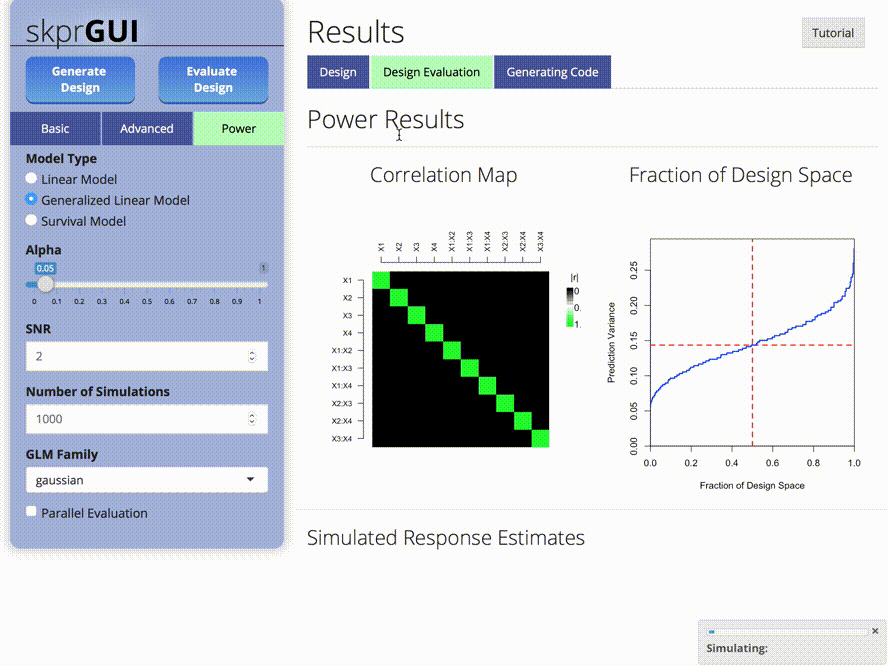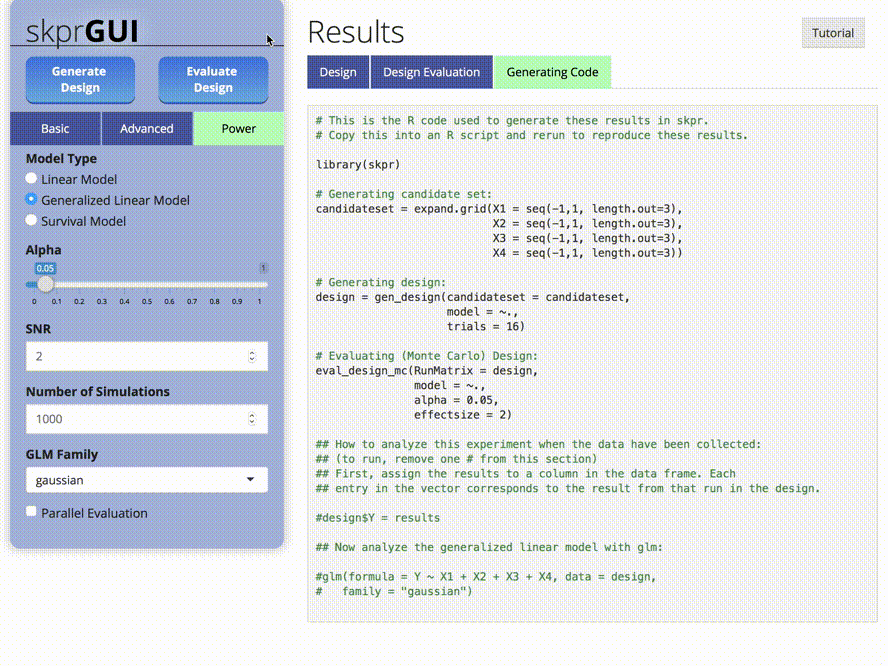
skprGUI() fournit une interface utilisateur graphique pour accéder à toutes les principales fonctionnalités de SKPR. Un tutoriel interactif est fourni pour familiariser l'utilisateur avec les fonctionnalités disponibles. Tapez skprGUI() pour commencer. Captures d'écran:
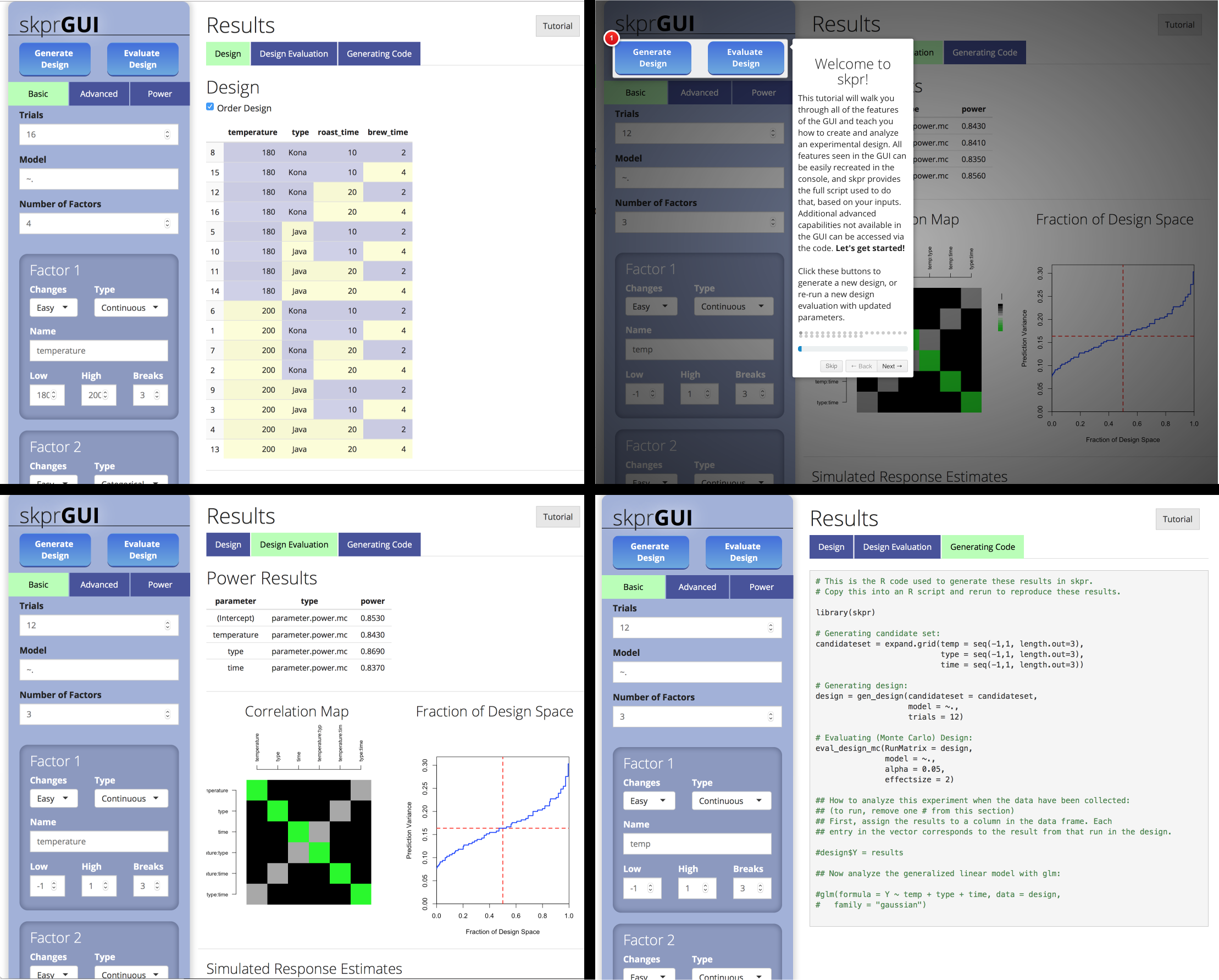
library( skpr )
# Generate a candidate set of all potential design points to be considered in the experiment
# The hypothetical experiment is determining what affects the caffeine content in coffee
candidate_set = expand.grid( temp = c( 80 , 90 , 100 ),
type = c( " Kona " , " Java " ),
beansize = c( " Large " , " Medium " , " Small " ))
candidate_set
# > temp type beansize
# > 1 80 Kona Large
# > 2 90 Kona Large
# > 3 100 Kona Large
# > 4 80 Java Large
# > 5 90 Java Large
# > 6 100 Java Large
# > 7 80 Kona Medium
# > 8 90 Kona Medium
# > 9 100 Kona Medium
# > 10 80 Java Medium
# > 11 90 Java Medium
# > 12 100 Java Medium
# > 13 80 Kona Small
# > 14 90 Kona Small
# > 15 100 Kona Small
# > 16 80 Java Small
# > 17 90 Java Small
# > 18 100 Java Small
# Generate the design (default D-optimal)
design = gen_design( candidateset = candidate_set ,
model = ~ temp + type + beansize ,
trials = 12 )
design
# > temp type beansize
# > 1 80 Java Medium
# > 2 100 Java Large
# > 3 100 Java Small
# > 4 80 Java Large
# > 5 80 Kona Medium
# > 6 80 Kona Small
# > 7 100 Kona Small
# > 8 100 Kona Medium
# > 9 80 Kona Large
# > 10 100 Java Medium
# > 11 100 Kona Large
# > 12 80 Java Small
# Evaluate power for the design with an allowable type-I error of 5% (default)
eval_design( design )
# > parameter type power
# > 1 (Intercept) effect.power 0.8424665
# > 2 temp effect.power 0.8424665
# > 3 type effect.power 0.8424665
# > 4 beansize effect.power 0.5165386
# > 5 (Intercept) parameter.power 0.8424665
# > 6 temp parameter.power 0.8424665
# > 7 type1 parameter.power 0.8424665
# > 8 beansize1 parameter.power 0.5593966
# > 9 beansize2 parameter.power 0.5593966
# > ============Evaluation Info============
# > * Alpha = 0.05 * Trials = 12 * Blocked = FALSE
# > * Evaluating Model = ~temp + type + beansize
# > * Anticipated Coefficients = c(1, 1, 1, 1, -1)
# > * Contrasts = `contr.sum`
# > * Parameter Analysis Method = `lm(...)`
# > * Effect Analysis Method = `car::Anova(fit, type = "III")`
# Evaluate power for the design using a Monte Carlo simulation.
# Here, we set the effect size (here, the signal-to-noise ratio) to 1.5.
eval_design_mc( design , effectsize = 1.5 )
# > parameter type power
# > 1 (Intercept) effect.power.mc 0.600
# > 2 temp effect.power.mc 0.612
# > 3 type effect.power.mc 0.610
# > 4 beansize effect.power.mc 0.316
# > 5 (Intercept) parameter.power.mc 0.600
# > 6 temp parameter.power.mc 0.612
# > 7 type1 parameter.power.mc 0.610
# > 8 beansize1 parameter.power.mc 0.359
# > 9 beansize2 parameter.power.mc 0.354
# > ===========Evaluation Info============
# > * Alpha = 0.05 * Trials = 12 * Blocked = FALSE
# > * Evaluating Model = ~temp + type + beansize
# > * Anticipated Coefficients = c(0.750, 0.750, 0.750, 0.750, -0.750)
# > * Contrasts = `contr.sum`
# > * Parameter Analysis Method = `lm(...)`
# > * Effect Analysis Method = `car::Anova(fit, type = "III")`
# Evaluate power for the design using a Monte Carlo simulation, for a non-normal response.
# Here, we also increase the number of simululations to improve the precision of the results.
eval_design_mc( design , nsim = 5000 , glmfamily = " poisson " , effectsize = c( 2 , 6 ))
# > parameter type power
# > 1 (Intercept) effect.power.mc 0.9968
# > 2 temp effect.power.mc 0.9826
# > 3 type effect.power.mc 0.9832
# > 4 beansize effect.power.mc 0.8502
# > 5 (Intercept) parameter.power.mc 0.9968
# > 6 temp parameter.power.mc 0.9826
# > 7 type1 parameter.power.mc 0.9832
# > 8 beansize1 parameter.power.mc 0.8842
# > 9 beansize2 parameter.power.mc 0.7052
# > ============Evaluation Info============
# > * Alpha = 0.05 * Trials = 12 * Blocked = FALSE
# > * Evaluating Model = ~temp + type + beansize
# > * Anticipated Coefficients = c(1.242, 0.549, 0.549, 0.549, -0.549)
# > * Contrasts = `contr.sum`
# > * Parameter Analysis Method = `glm(..., family = "poisson")`
# > * Effect Analysis Method = `car::Anova(fit, type = "III")`
# skpr was designed to operate with the pipe (|>) in mind.
# Here is an example of an entire design of experiments analysis in three lines:
expand.grid( temp = c( 80 , 90 , 100 ), type = c( " Kona " , " Java " ), beansize = c( " Large " , " Medium " , " Small " )) | >
gen_design( model = ~ temp + type + beansize + beansize : type + I( temp ^ 2 ), trials = 24 , optimality = " I " ) | >
eval_design_mc( detailedoutput = TRUE )
# > parameter type power anticoef alpha glmfamily trials
# > 1 (Intercept) effect.power.mc 0.912 NA 0.05 gaussian 24
# > 2 temp effect.power.mc 0.927 NA 0.05 gaussian 24
# > 3 type effect.power.mc 0.997 NA 0.05 gaussian 24
# > 4 beansize effect.power.mc 0.935 NA 0.05 gaussian 24
# > 5 I(temp^2) effect.power.mc 0.637 NA 0.05 gaussian 24
# > 6 type:beansize effect.power.mc 0.913 NA 0.05 gaussian 24
# > 7 (Intercept) parameter.power.mc 0.912 1 0.05 gaussian 24
# > 8 temp parameter.power.mc 0.927 1 0.05 gaussian 24
# > 9 type1 parameter.power.mc 0.997 1 0.05 gaussian 24
# > 10 beansize1 parameter.power.mc 0.917 1 0.05 gaussian 24
# > 11 beansize2 parameter.power.mc 0.913 -1 0.05 gaussian 24
# > 12 I(temp^2) parameter.power.mc 0.637 1 0.05 gaussian 24
# > 13 type1:beansize1 parameter.power.mc 0.899 1 0.05 gaussian 24
# > 14 type1:beansize2 parameter.power.mc 0.902 -1 0.05 gaussian 24
# > nsim blocking error_adjusted_alpha power_lcb power_ucb
# > 1 1000 FALSE 0.05 0.8927052 0.9288249
# > 2 1000 FALSE 0.05 0.9090858 0.9423464
# > 3 1000 FALSE 0.05 0.9912580 0.9993809
# > 4 1000 FALSE 0.05 0.9178989 0.9494797
# > 5 1000 FALSE 0.05 0.6063275 0.6668632
# > 6 1000 FALSE 0.05 0.8937921 0.9297315
# > 7 1000 FALSE 0.05 0.8927052 0.9288249
# > 8 1000 FALSE 0.05 0.9090858 0.9423464
# > 9 1000 FALSE 0.05 0.9912580 0.9993809
# > 10 1000 FALSE 0.05 0.8981467 0.9333511
# > 11 1000 FALSE 0.05 0.8937921 0.9297315
# > 12 1000 FALSE 0.05 0.6063275 0.6668632
# > 13 1000 FALSE 0.05 0.8786332 0.9169799
# > 14 1000 FALSE 0.05 0.8818715 0.9197225
# > =========================================================Evaluation Info==========================================================
# > * Alpha = 0.05 * Trials = 24 * Blocked = FALSE
# > * Evaluating Model = ~temp + type + beansize + type:beansize + I(temp^2)
# > * Anticipated Coefficients = c(1, 1, 1, 1, -1, 1, 1, -1)
# > * Contrasts = `contr.sum`
# > * Parameter Analysis Method = `lm(...)`
# > * Effect Analysis Method = `car::Anova(fit, type = "III")`
# > * MC Power CI Confidence = 95%