Код Pytorch для статьи «Медицинский трансформатор: закрытое осевое сопровождение для сегментации медицинской изображения», Miccai 2021
Бумага | Плакат
: Проверьте нашу последнюю работу Unext, более быструю и более эффективную архитектуру сегментации, которая также легко тренировать и реализовать! Код доступен здесь.
Это репо размещает код для следующих сетей:
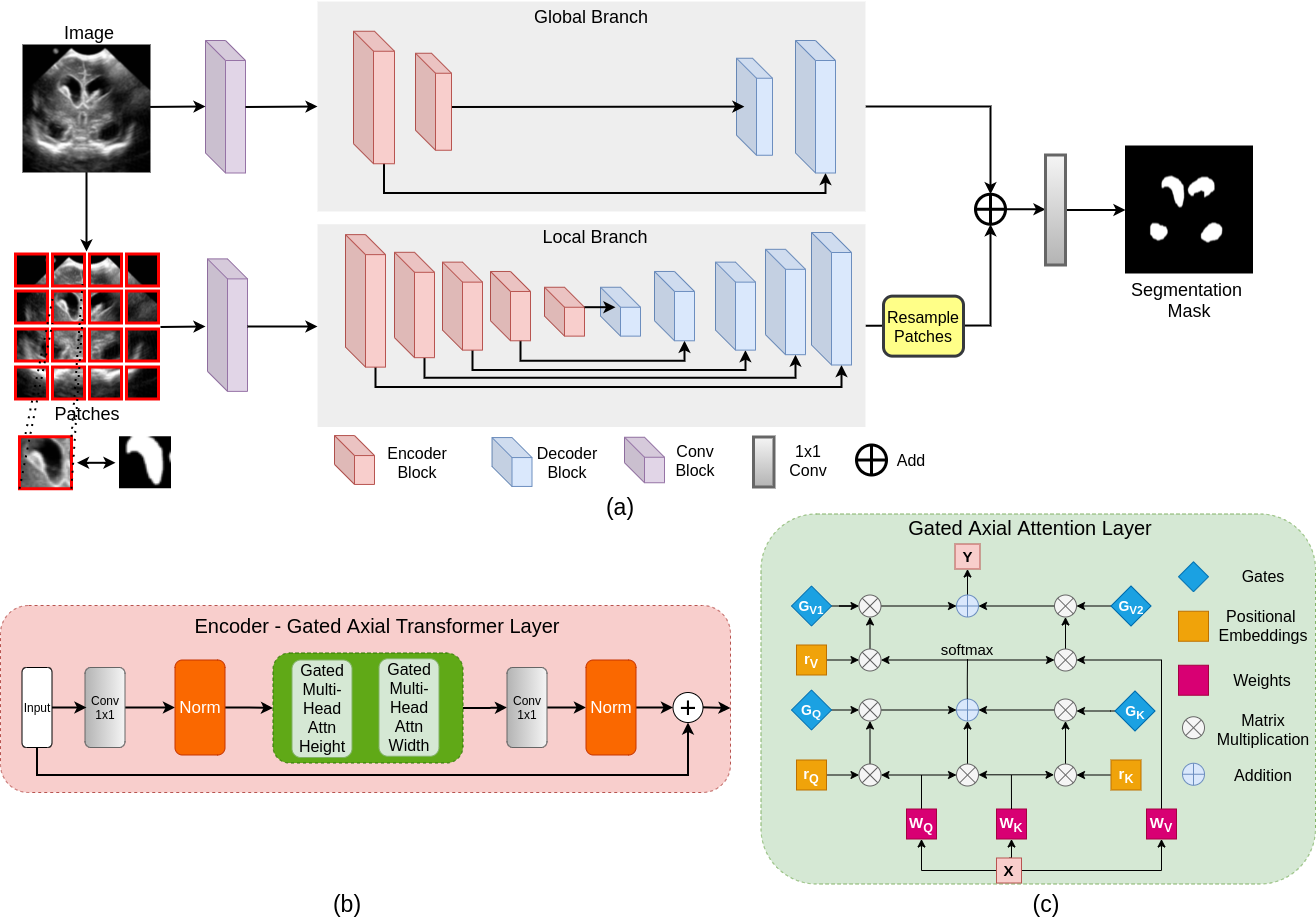
Большинство существующих сетевых архитектур на основе трансформамеров, предложенных для приложений для зрения, требуют крупномасштабных наборов данных для правильной тренировки. Однако по сравнению с наборами данных для применений зрения, для медицинской визуализации количество образцов данных относительно низкое, что затрудняет эффективное обучение трансформаторов для медицинских применений. С этой целью мы предлагаем закрытую модель осевого внимания, которая расширяет существующую архитектуру, внедряя дополнительный механизм управления в модуле самопричастования. Кроме того, чтобы эффективно обучить модель медицинским изображениям, мы предлагаем локальную глобальную учебную страту (логотип), которая еще больше улучшает производительность. В частности, мы указываем на все изображение и патчи, чтобы изучать глобальные и локальные функции, соответственно. Предлагаемый медицинский трансформатор (MERT) использует стратегию обучения логотипа по закрытому осевому вниманию U-Net.
git clone https://github.com/jeya-maria-jose/Medical-Transformer
cd Medical-TransformerКод стабилен с использованием Python 3.6.10, Pytorch 1.4.0
Чтобы установить все зависимости, используя Conda:
conda env create -f environment.yml
conda activate medtЧтобы установить все зависимости, используя PIP:
pip install -r requirements.txtПодготовьте набор данных в следующем формате для легкого использования кода. Поезда и тестовые папки должны содержать по двум подпапкам каждый: IMG и метка. Убедитесь, что изображения их соответствующие маски сегментации расположены под этими папками и имеют одинаковое имя для удобной корреспонденции. Пожалуйста, измените погрузчики данных на вашу потребность, если вы предпочитаете не подготовить набор данных в этом формате.
Train Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
Validation Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
Test Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
python train.py --train_dataset " enter train directory " --val_dataset " enter validation directory " --direc ' path for results to be saved ' --batch_size 4 --epoch 400 --save_freq 10 --modelname " gatedaxialunet " --learning_rate 0.001 --imgsize 128 --gray " no " Change modelname to MedT or logo to train thempython test.py --loaddirec " ./saved_model_path/model_name.pth " --val_dataset " test dataset directory " --direc ' path for results to be saved ' --batch_size 1 --modelname " gatedaxialunet " --imgsize 128 --gray " no "Результаты, включая прогнозируемые карты сегментации, будут размещены в папке результатов вместе с весами модели. Запустите код показателей производительности в MATLAB для расчета оценки F1 и MIO.
1) Обратите внимание, что эти эксперименты проводились в Nvidia Quadro 8000 с памятью 48 ГБ. 2) Код Google Colab является неофициальной реализацией для быстрого поезда/теста. Пожалуйста, следуйте оригинальному коду для правильного обучения.
Код DataLoader вдохновлен от Pytorch-Unet. Код осевого внимания разработан из осевой диапазоны.
@InProceedings{jose2021medical,
author= " Valanarasu, Jeya Maria Jose
and Oza, Poojan
and Hacihaliloglu, Ilker
and Patel, Vishal M. " ,
title= " Medical Transformer: Gated Axial-Attention for Medical Image Segmentation " ,
booktitle= " Medical Image Computing and Computer Assisted Intervention -- MICCAI 2021 " ,
year= " 2021 " ,
publisher= " Springer International Publishing " ,
address= " Cham " ,
pages= " 36--46 " ,
isbn= " 978-3-030-87193-2 "
}
Откройте проблему или напишите мне напрямую в случае любых запросов или предложений.