Código de Pytorch para el documento "Transformador médico: asistencia axial cerrada para la segmentación de imágenes médicas", Miccai 2021
Papel | Póster
: Consulte nuestro último trabajo sin EEXT, una arquitectura de segmentación más rápida y eficiente que también es fácil de entrenar e implementar. El código está disponible aquí.
Este repositorio aloja el código para las siguientes redes:
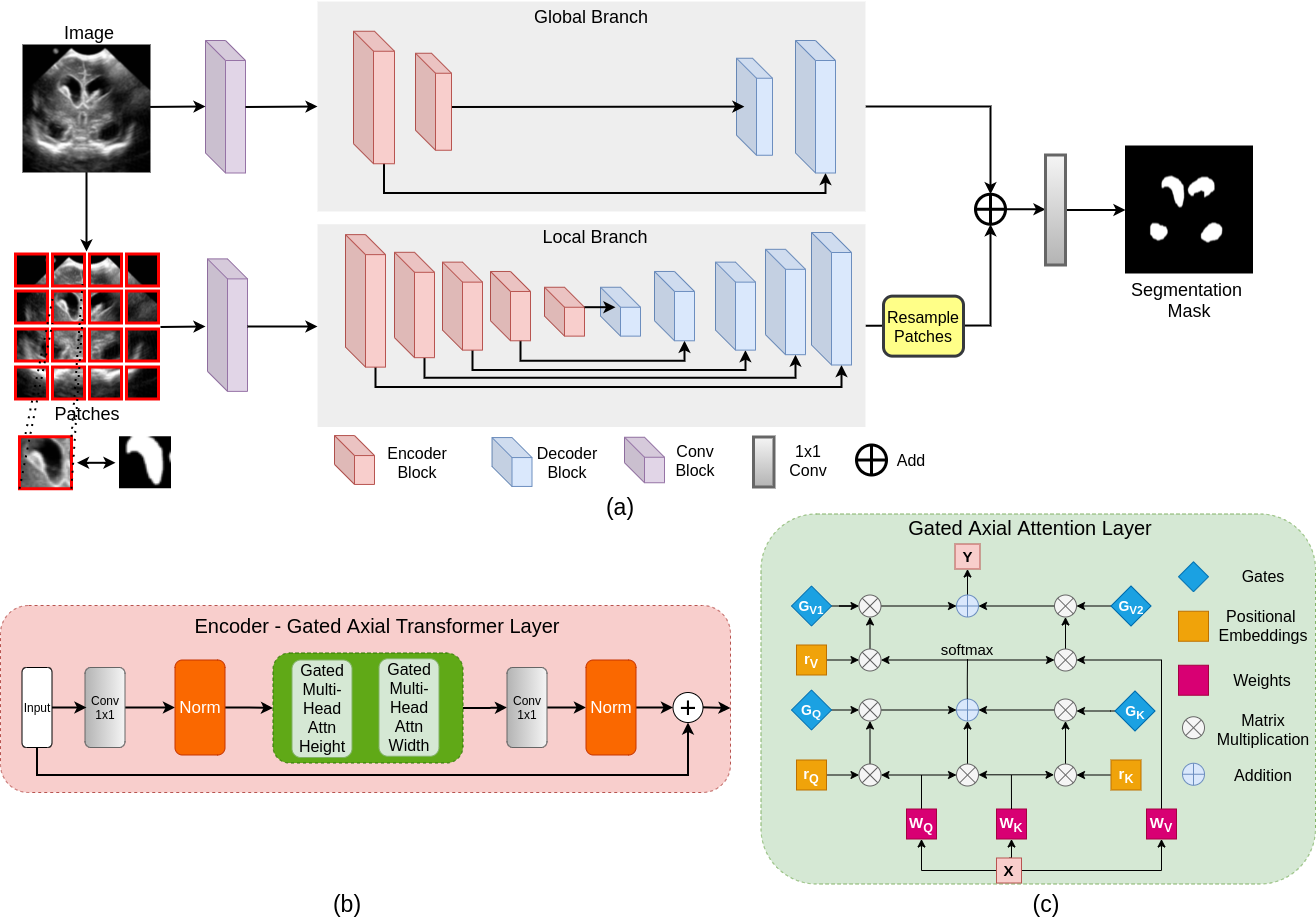
La mayoría de las arquitecturas de red basadas en transformadores existentes propuestas para aplicaciones de visión requieren conjuntos de datos a gran escala para entrenar correctamente. Sin embargo, en comparación con los conjuntos de datos para aplicaciones de visión, para imágenes médicas, el número de muestras de datos es relativamente bajo, lo que dificulta entrenar de manera eficiente los transformadores para aplicaciones de medicina. Con este fin, proponemos un modelo de atención axial cerrada que extiende las arquitecturas existentes mediante la introducción de un mecanismo de control adicional en el módulo de autoatención. Además, para entrenar el modelo de manera efectiva en imágenes médicas, proponemos una estrategia de entrenamiento global local (logotipo) que mejora aún más el rendimiento. Específicamente, optimizamos en toda la imagen y parches para aprender características globales y locales, respectivamente. El transformador médico propuesto (MEDT) utiliza la estrategia de entrenamiento de logotipos en la red de atención a axial cerrada.
git clone https://github.com/jeya-maria-jose/Medical-Transformer
cd Medical-TransformerEl código es estable usando Python 3.6.10, Pytorch 1.4.0
Para instalar todas las dependencias usando conda:
conda env create -f environment.yml
conda activate medtPara instalar todas las dependencias utilizando PIP:
pip install -r requirements.txtPrepare el conjunto de datos en el siguiente formato para un fácil uso del código. El tren y las carpetas de prueba deben contener dos subcarpetas cada una: IMG y etiqueta. Asegúrese de que las imágenes sus máscaras de segmentación correspondientes estén colocadas en estas carpetas y tengan el mismo nombre para facilitar la correspondencia. Cambie los cargadores de datos a su necesidad si prefiere no preparar el conjunto de datos en este formato.
Train Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
Validation Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
Test Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
python train.py --train_dataset " enter train directory " --val_dataset " enter validation directory " --direc ' path for results to be saved ' --batch_size 4 --epoch 400 --save_freq 10 --modelname " gatedaxialunet " --learning_rate 0.001 --imgsize 128 --gray " no " Change modelname to MedT or logo to train thempython test.py --loaddirec " ./saved_model_path/model_name.pth " --val_dataset " test dataset directory " --direc ' path for results to be saved ' --batch_size 1 --modelname " gatedaxialunet " --imgsize 128 --gray " no "Los resultados, incluidos los mapas de segmentación predichos, se colocarán en la carpeta de resultados junto con los pesos del modelo. Ejecute el código de métricas de rendimiento en MATLAB para calcular la puntuación F1 y miu.
1) Tenga en cuenta que estos experimentos se realizaron en Nvidia Quadro 8000 con memoria de 48 GB. 2) El código de Google Colab es una implementación no oficial para el tren/prueba rápida. Siga el código original para una capacitación adecuada.
El código DataLoader está inspirado en Pytorch-Unet. El código de atención axial se desarrolla a partir de Axial-DePlab.
@InProceedings{jose2021medical,
author= " Valanarasu, Jeya Maria Jose
and Oza, Poojan
and Hacihaliloglu, Ilker
and Patel, Vishal M. " ,
title= " Medical Transformer: Gated Axial-Attention for Medical Image Segmentation " ,
booktitle= " Medical Image Computing and Computer Assisted Intervention -- MICCAI 2021 " ,
year= " 2021 " ,
publisher= " Springer International Publishing " ,
address= " Cham " ,
pages= " 36--46 " ,
isbn= " 978-3-030-87193-2 "
}
Abra un problema o envíeme un correo electrónico directamente en caso de cualquier consulta o sugerencia.