Code Pytorch pour le document "Transformateur médical: l'attention axiale fermée pour la segmentation de l'image médicale", Miccai 2021
Papier | Affiche
: Découvrez nos derniers travaux, une architecture de segmentation plus rapide et plus efficace qui est également facile à former et à mettre en œuvre! Le code est disponible ici.
Ce repo héberge le code des réseaux suivants:
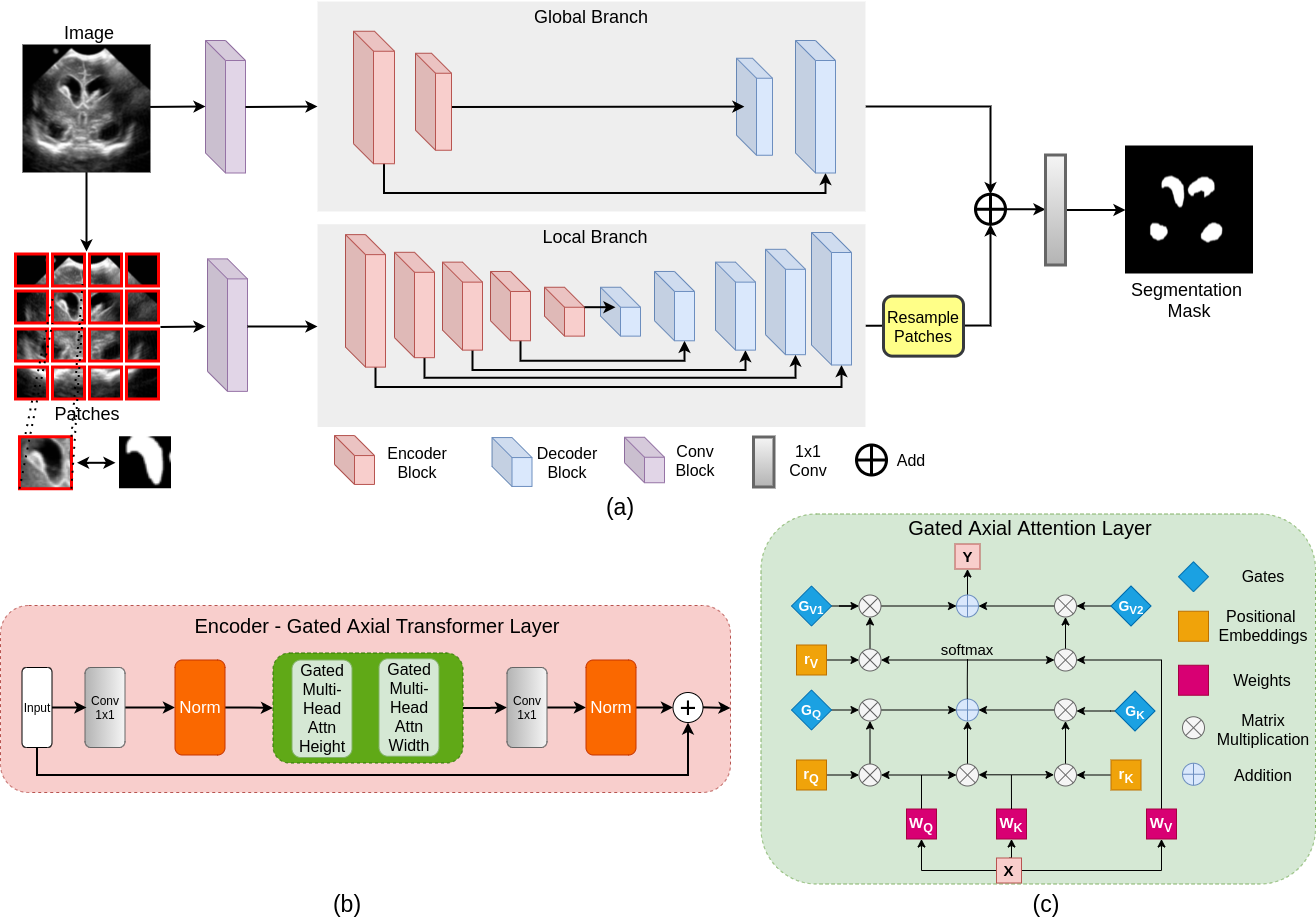
La majorité des architectures de réseau basées sur les transformateurs existantes proposées pour les applications de vision nécessitent des ensembles de données à grande échelle pour s'entraîner correctement. Cependant, par rapport aux ensembles de données pour les applications de vision, pour l'imagerie médicale, le nombre d'échantillons de données est relativement faible, ce qui rend difficile de former efficacement les transformateurs pour les applications médicales. À cette fin, nous proposons un modèle d'attention axiale fermé qui étend les architectures existantes en introduisant un mécanisme de contrôle supplémentaire dans le module d'auto-atténuation. De plus, pour former efficacement le modèle sur les images médicales, nous proposons une stratégie de formation locale-globale (logo) qui améliore encore la performance. Plus précisément, nous opposons à l'ensemble de l'image et des correctifs pour apprendre les fonctionnalités globales et locales, respectivement. Le transformateur médical proposé (MEDT) utilise la stratégie de formation du logo sur l'attention axiale fermée U-NET.
git clone https://github.com/jeya-maria-jose/Medical-Transformer
cd Medical-TransformerLe code est stable à l'aide de Python 3.6.10, Pytorch 1.4.0
Pour installer toutes les dépendances à l'aide de conda:
conda env create -f environment.yml
conda activate medtPour installer toutes les dépendances à l'aide de PIP:
pip install -r requirements.txtPréparez l'ensemble de données dans le format suivant pour une utilisation facile du code. Les dossiers de train et de test doivent contenir deux sous-dossiers chacun: IMG et étiquette. Assurez-vous que les images de leurs masques de segmentation correspondantes sont placées sous ces dossiers et avoir le même nom pour une correspondance facile. Veuillez modifier les chargeurs de données par votre besoin si vous préférez ne pas préparer l'ensemble de données dans ce format.
Train Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
Validation Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
Test Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
python train.py --train_dataset " enter train directory " --val_dataset " enter validation directory " --direc ' path for results to be saved ' --batch_size 4 --epoch 400 --save_freq 10 --modelname " gatedaxialunet " --learning_rate 0.001 --imgsize 128 --gray " no " Change modelname to MedT or logo to train thempython test.py --loaddirec " ./saved_model_path/model_name.pth " --val_dataset " test dataset directory " --direc ' path for results to be saved ' --batch_size 1 --modelname " gatedaxialunet " --imgsize 128 --gray " no "Les résultats, y compris les cartes de segmentations prévus, seront placés dans le dossier des résultats ainsi que les poids du modèle. Exécutez le code de métriques de performances dans MATLAB pour calculer le score F1 et MIOU.
1) Notez que ces expériences ont été menées dans NVIDIA Quadro 8000 avec une mémoire de 48 Go. 2) Le code Google Colab est une implémentation non officielle pour le train / test rapide. Veuillez suivre le code d'origine pour une formation appropriée.
Le code DatalOader est inspiré de Pytorch-Unet. Le code d'attention axial est développé à partir de la profondeur axiale.
@InProceedings{jose2021medical,
author= " Valanarasu, Jeya Maria Jose
and Oza, Poojan
and Hacihaliloglu, Ilker
and Patel, Vishal M. " ,
title= " Medical Transformer: Gated Axial-Attention for Medical Image Segmentation " ,
booktitle= " Medical Image Computing and Computer Assisted Intervention -- MICCAI 2021 " ,
year= " 2021 " ,
publisher= " Springer International Publishing " ,
address= " Cham " ,
pages= " 36--46 " ,
isbn= " 978-3-030-87193-2 "
}
Ouvrez un problème ou envoyez-moi un courrier directement en cas de requêtes ou de suggestions.