Código de Pytorch para o artigo "Transformador médico: atendimento axial portátil para segmentação de imagem médica", Miccai 2021
Papel | Poster
: Confira nosso trabalho mais recente, uma arquitetura de segmentação mais rápida e eficiente, que também é fácil de treinar e implementar! O código está disponível aqui.
Este repo hospeda o código para as seguintes redes:
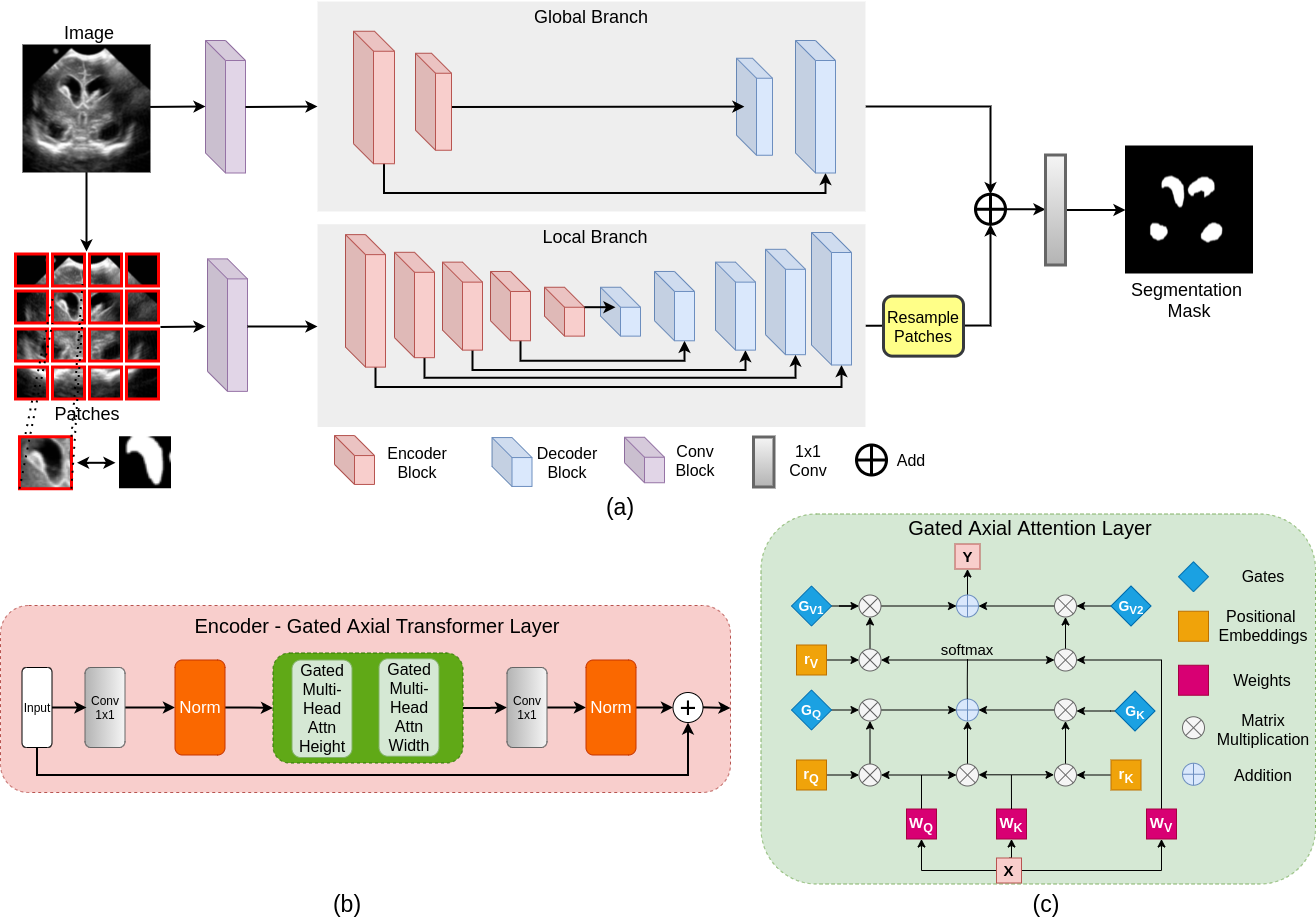
A maioria das arquiteturas de rede baseadas em transformador existentes propostas para aplicações de visão exigem conjuntos de dados em larga escala para treinar corretamente. No entanto, em comparação com os conjuntos de dados para aplicações de visão, para imagens médicas, o número de amostras de dados é relativamente baixo, dificultando o treino com eficiência transformadores para aplicações médicas. Para esse fim, propomos um modelo de atendimento axial fechado que estende as arquiteturas existentes, introduzindo um mecanismo de controle adicional no módulo de auto-atenção. Além disso, para treinar o modelo de maneira eficaz em imagens médicas, propomos uma estratégia de treinamento local-global (logotipo), o que melhora ainda mais o desempenho. Especificamente, optamos por toda a imagem e patches para aprender recursos globais e locais, respectivamente. O Transformador Médico Proposto (MEDT) usa a estratégia de treinamento de logotipo na atenção axial fechada.
git clone https://github.com/jeya-maria-jose/Medical-Transformer
cd Medical-TransformerO código é estável usando o Python 3.6.10, Pytorch 1.4.0
Para instalar todas as dependências usando o CONDA:
conda env create -f environment.yml
conda activate medtPara instalar todas as dependências usando PIP:
pip install -r requirements.txtPrepare o conjunto de dados no formato a seguir para facilitar o uso do código. As pastas de trem e teste devem conter duas subpastas cada: img e etiqueta. Verifique se as imagens suas máscaras de segmentação correspondentes são colocadas nessas pastas e têm o mesmo nome para facilitar a correspondência. Altere os carregadores de dados para sua necessidade, se você preferir não preparar o conjunto de dados neste formato.
Train Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
Validation Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
Test Folder-----
img----
0001.png
0002.png
.......
labelcol---
0001.png
0002.png
.......
python train.py --train_dataset " enter train directory " --val_dataset " enter validation directory " --direc ' path for results to be saved ' --batch_size 4 --epoch 400 --save_freq 10 --modelname " gatedaxialunet " --learning_rate 0.001 --imgsize 128 --gray " no " Change modelname to MedT or logo to train thempython test.py --loaddirec " ./saved_model_path/model_name.pth " --val_dataset " test dataset directory " --direc ' path for results to be saved ' --batch_size 1 --modelname " gatedaxialunet " --imgsize 128 --gray " no "Os resultados, incluindo mapas de segmentações previstos, serão colocados na pasta de resultados junto com os pesos do modelo. Execute o código de métricas de desempenho no MATLAB para calcular a pontuação F1 e o MIOU.
1) Observe que essas experiências foram realizadas no Nvidia Quadro 8000 com 48 GB de memória. 2) O código do Google Colab é uma implementação não oficial para trem/teste rápido. Siga o código original para o treinamento adequado.
O código Dataloader é inspirado no Pytorch-Unet. O código de atenção axial é desenvolvido a partir de Axial-Deeplab.
@InProceedings{jose2021medical,
author= " Valanarasu, Jeya Maria Jose
and Oza, Poojan
and Hacihaliloglu, Ilker
and Patel, Vishal M. " ,
title= " Medical Transformer: Gated Axial-Attention for Medical Image Segmentation " ,
booktitle= " Medical Image Computing and Computer Assisted Intervention -- MICCAI 2021 " ,
year= " 2021 " ,
publisher= " Springer International Publishing " ,
address= " Cham " ,
pages= " 36--46 " ,
isbn= " 978-3-030-87193-2 "
}
Abra um problema ou envie -me diretamente em caso de dúvidas ou sugestões.