? Leia o papel completo
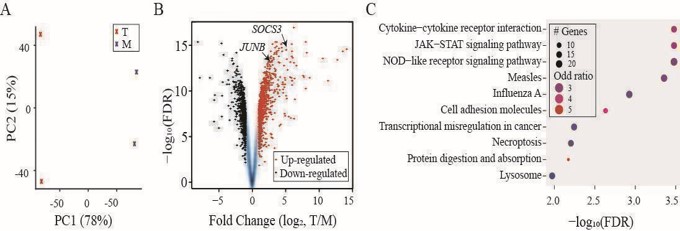
(A) Análise de PCA mostrando dois grupos de amostras: "T" em vermelho e "M" em azul. (B) Um gráfico de vulcão para FCs e FDRs a partir de análise genética diferencialmente expressa com genes regulados para cima em genes vermelhos e regulados para baixo em preto. SOCS3 e JUNB indicados por Arrowheads. (C) O gráfico de pontos resumindo os dez principais hits da análise de enriquecimento da via de KEGG aplicada aos genes regulados para cima.
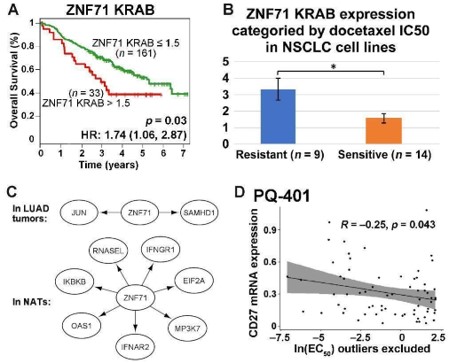
(A) Gráfico de sobrevivência de Kaplan -Meier para pacientes com NSCLC (GSE81089) classificados pela expressão de isoforma KRAB ZNF71. Vermelho: pacientes com uma expressão mais alta de ZNF71 KRAB (TPM> 1,5); Verde: outros; (B) Um gráfico de barras para a expressão da isoforma Krab Znf71 entre as linhas celulares NSCLC classificadas pela sensibilidade à docetaxel. (C) Rede de associação genética de genes Znf71 e IIIR em tumores Luad e Nats. (D) Gráfico de dispersão para o nível de expressão do mRNA de CD27 e resposta a medicamentos ao PQ-401 (EC50) entre as linhas celulares NSCLS.
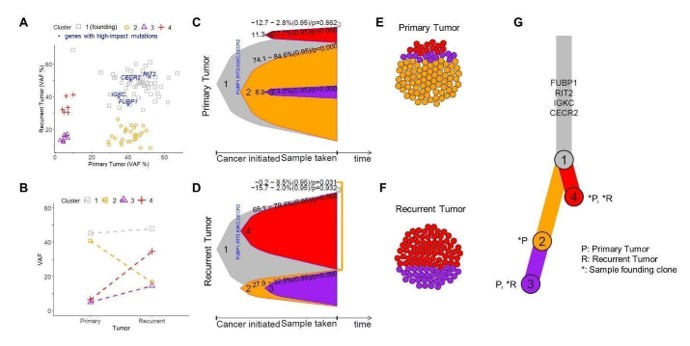
(A) Gráfico de dispersão para Tumor recorrente contrastante de VAF (eixo y) ao tumor primário (eixo x). Cada ponto é um gene e genes com impacto significativo são indicados. Tumores primários e recorrentes provenientes do mesmo paciente. (B) Gráfico de pontos conectado ilustrando as alterações da VAF média de cada cluster do tumor primário para o recorrente. (C) Gráfico de sino ilustrando a prevalência relativa de cada clone (sub) em função da progressão do câncer para o tumor primário e (d) tumor recorrente. (E) Gráficos de empacotamento de círculo ilustrando a composição celular do tumor primário e (f) tumor recorrente, examinado com um total de 100 células. (G) Uma árvore baseada em ramificação que ilustra os padrões relacionais e de semeadura dos clones (sub) entre as amostras primárias e recorrentes. Indicados são genes que facilitam o crescimento do clone fundador.
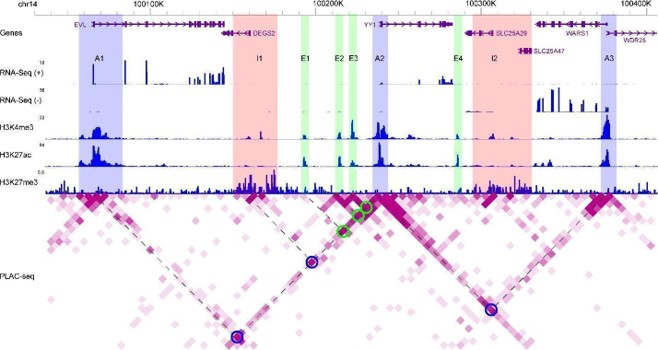
Tracks do navegador epigenoma de Washu ilustrando distribuições de leituras de RNA-seq específicas da fita, leituras de chip-seq para H3K4me3, H3K27AC e H3K27me3, bem como as ladas de interações cromatinas-cromatinas. Retângulo azul (A1 a A3): regiões promotoras ativas; Retângulos rosa (i1 e i2): regiões promotoras inativas; Retângulos verdes (E1 a E4): Regiões de intensificadores ativos; Círculos azuis: regiões promotoras (A1, i1 e A3) interagindo com YY1; Círculos verdes: regiões de intensificadores (E1 a E3) interagindo com YY1. Linhas tracejadas compartilhadas com um círculo no mapa de calor: duas regiões genômicas que interagem. Grau de pupforma no mapa de calor indicando força de interação.

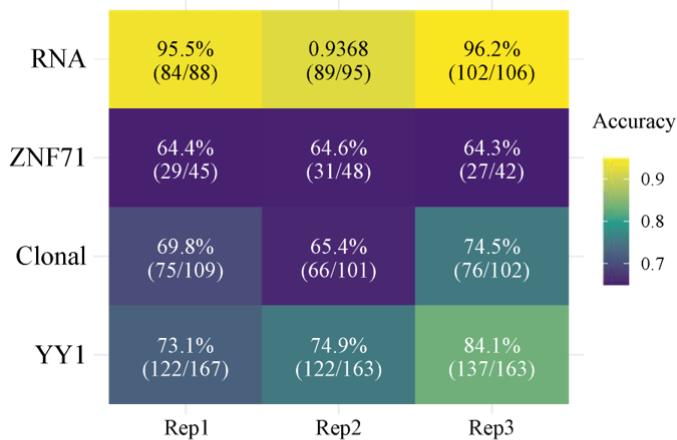
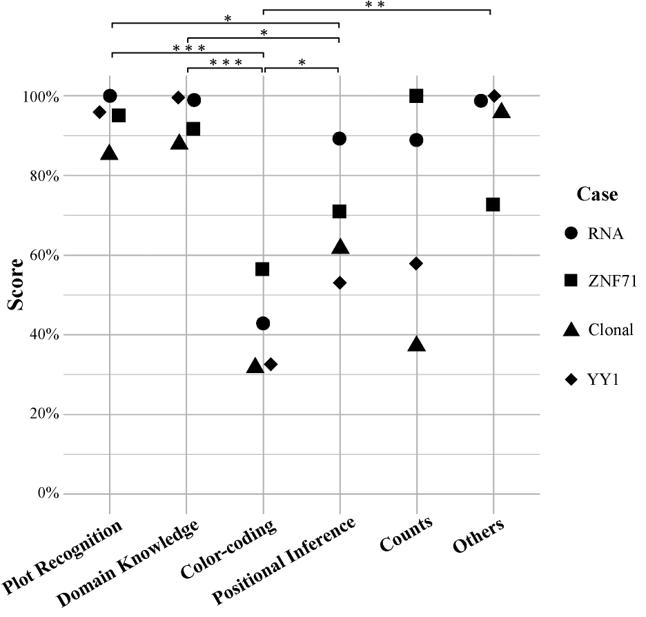
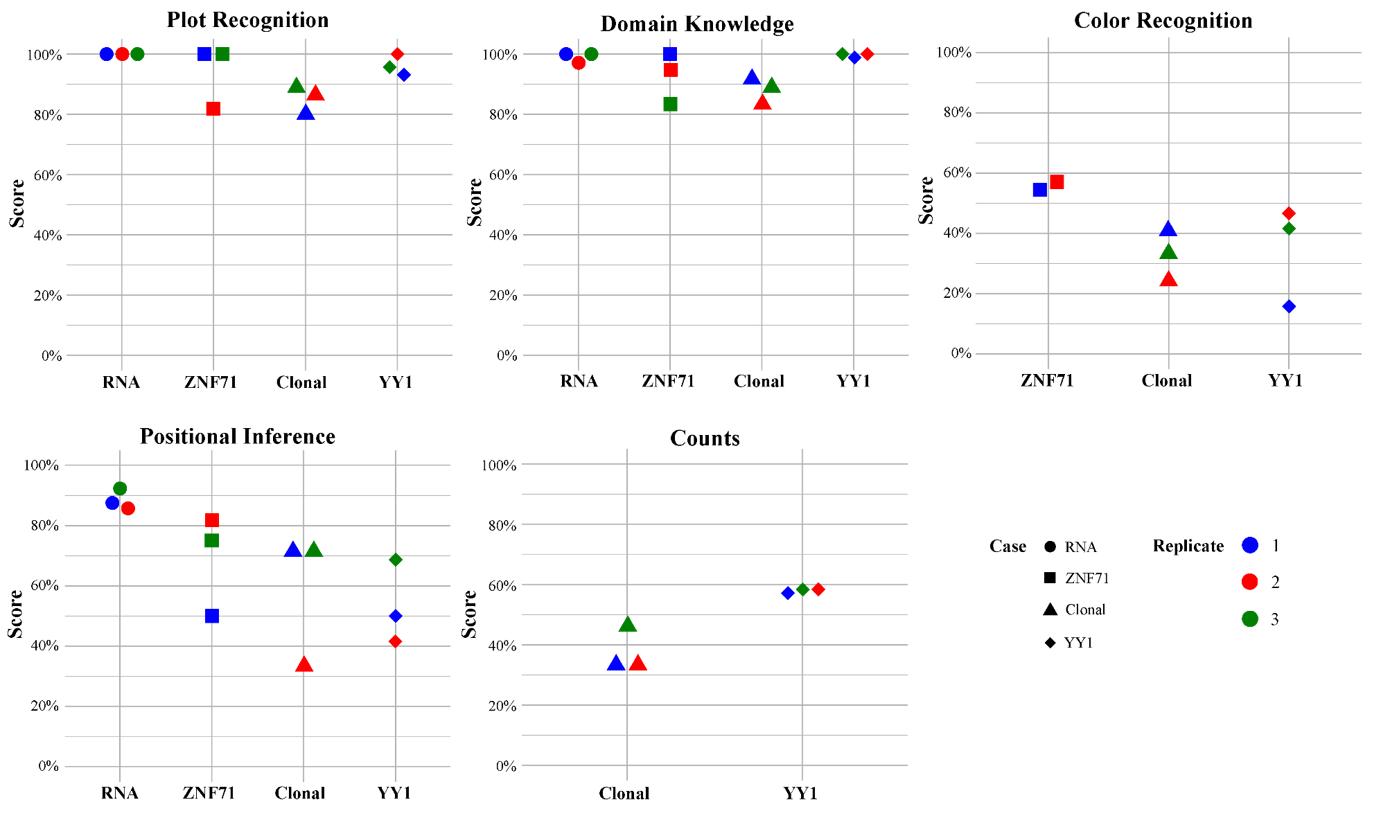

@Article {Wang2024Scientific, title = {figuras científicas interpretadas por chatgpt: forças no reconhecimento da plotagem e limites na percepção de cores}, autor = {wang, jinge e ye, qing e liU, li e guo, nancy lan e hu, gangqing}, journal = {npj nmemonnem onem,}} {npjumnuming on), na {{npj), na {{npj), na {n). páginas = {84}, ano = {2024}, editor = {Nature Publishing Group UK London}}