? Lea el papel completo
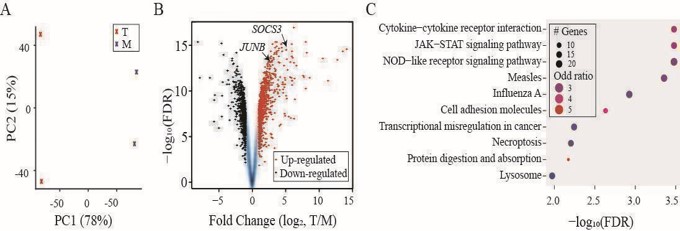
(A) Análisis de PCA que muestra dos grupos de muestras: "T" en rojo y "M" en azul. (B) Una gráfica de volcán para FCS y FDRS del análisis de genes expresados diferencialmente con genes regulados por ascenso en genes rojos y regulados negativos en negro. SOCS3 y Junb indicados por puntas de flecha. (C) Parcela de puntos que resume los diez mejores éxitos del análisis de enriquecimiento de la vía KEGG aplicada a los genes regulados hacia arriba.
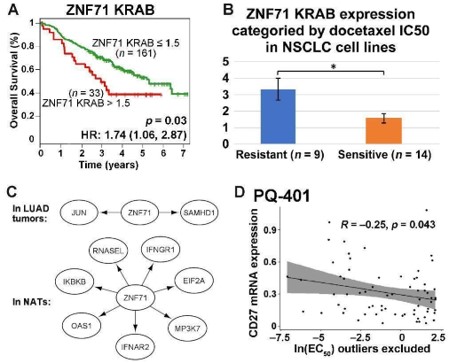
(A) Gráfico de supervivencia de Kaplan -Meier para pacientes con CPNC (GSE81089) ordenado por la expresión de la isoforma KRAB ZNF71. Rojo: pacientes con una expresión de KRAB ZNF71 más alta (TPM> 1.5); Verde: otros; (B) Un gráfico de barras para la expresión de la isoforma KRAB ZNF71 entre las líneas celulares NSCLC ordenadas por la sensibilidad de Docetaxel. (C) Red de asociación de genes de genes ZNF71 y IIIR en tumores LUAD y NATS. (D) Gráfico de dispersión para el nivel de expresión de ARNm de CD27 y la respuesta al fármaco a PQ-401 (EC50) entre las líneas celulares de NSCLS.
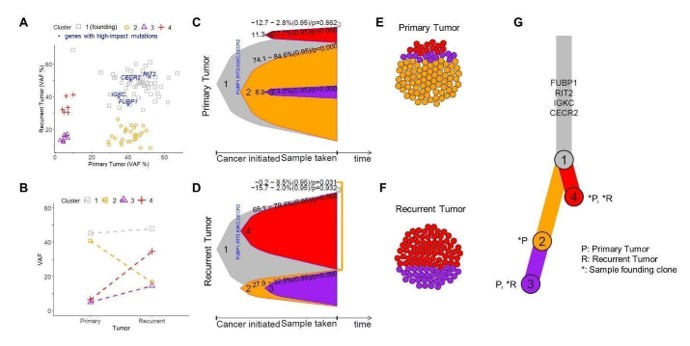
(A) Gráfico de dispersión para Vaf% contrastante tumor recurrente (eje Y) al tumor primario (eje x). Cada punto es un gen y se indican genes con un impacto significativo. Tumores primarios y recurrentes procedentes del mismo paciente. (B) TRÁNTO DE DOT conectado que ilustra los cambios de VAF medio de cada grupo desde el tumor primario hasta el recurrente. (C) Parcela de campana que ilustra la prevalencia relativa de cada clon (sub) en función de la progresión del cáncer para el tumor primario y (d) tumor recurrente. (E) Gráficos de empaque del círculo que ilustran la composición celular del tumor primario y (f) tumor recurrente, examinado con un total de 100 células. (G) Un árbol a base de ramas que ilustra los patrones relacionales y de siembra de los (sub) clones entre las muestras primarias y recurrentes. Se indican genes que facilitan el crecimiento del clon fundador.
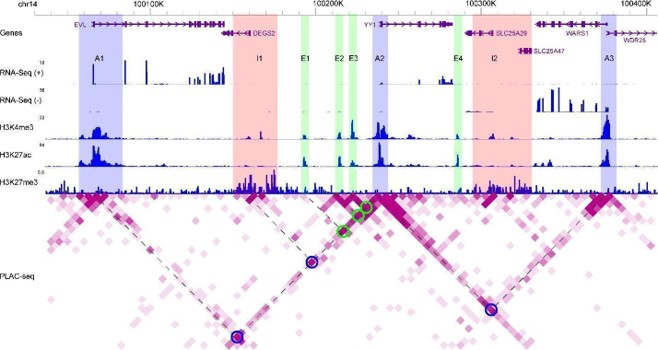
Lecturas de BROWSER EPIGENOME WASHU que ilustran las distribuciones de lecturas de ARN-seq específicas de Strand, ChIP-Seq dice para H3K4ME3, H3K27AC y H3K27ME3, así como lecturas de Plac-Seq para las interacciones con cromatina-cromatina. Rectángulos azules (A1 a A3): regiones promotoras activas; Rectángulos rosados (I1 e I2): regiones promotoras inactivas; Rectángulos verdes (E1 a E4): regiones potenciantes activas; Círculos azules: regiones promotoras (A1, I1 y A3) que interactúan con YY1; Círculos verdes: regiones potenciantes (E1 a E3) que interactúan con YY1. Líneas discontinuas compartidas con un círculo en el mapa de calor: dos regiones del genoma interactuante. Grado de púrpura en el mapa de calor que acusa a la resistencia a la interacción.

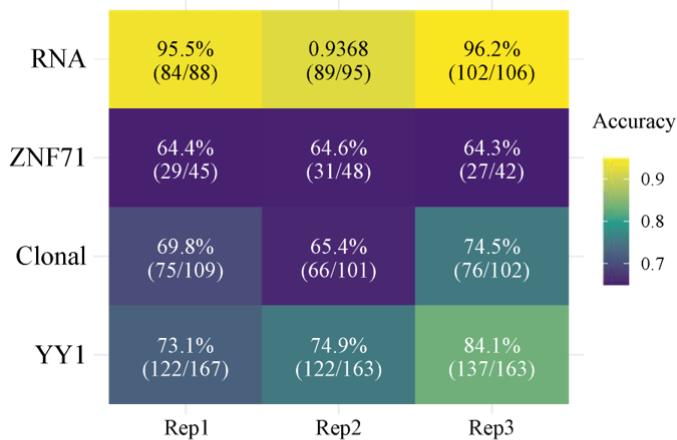
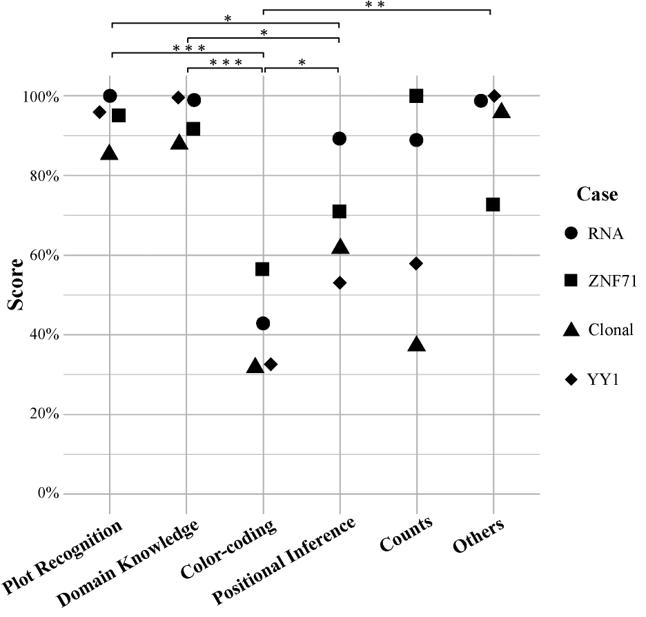
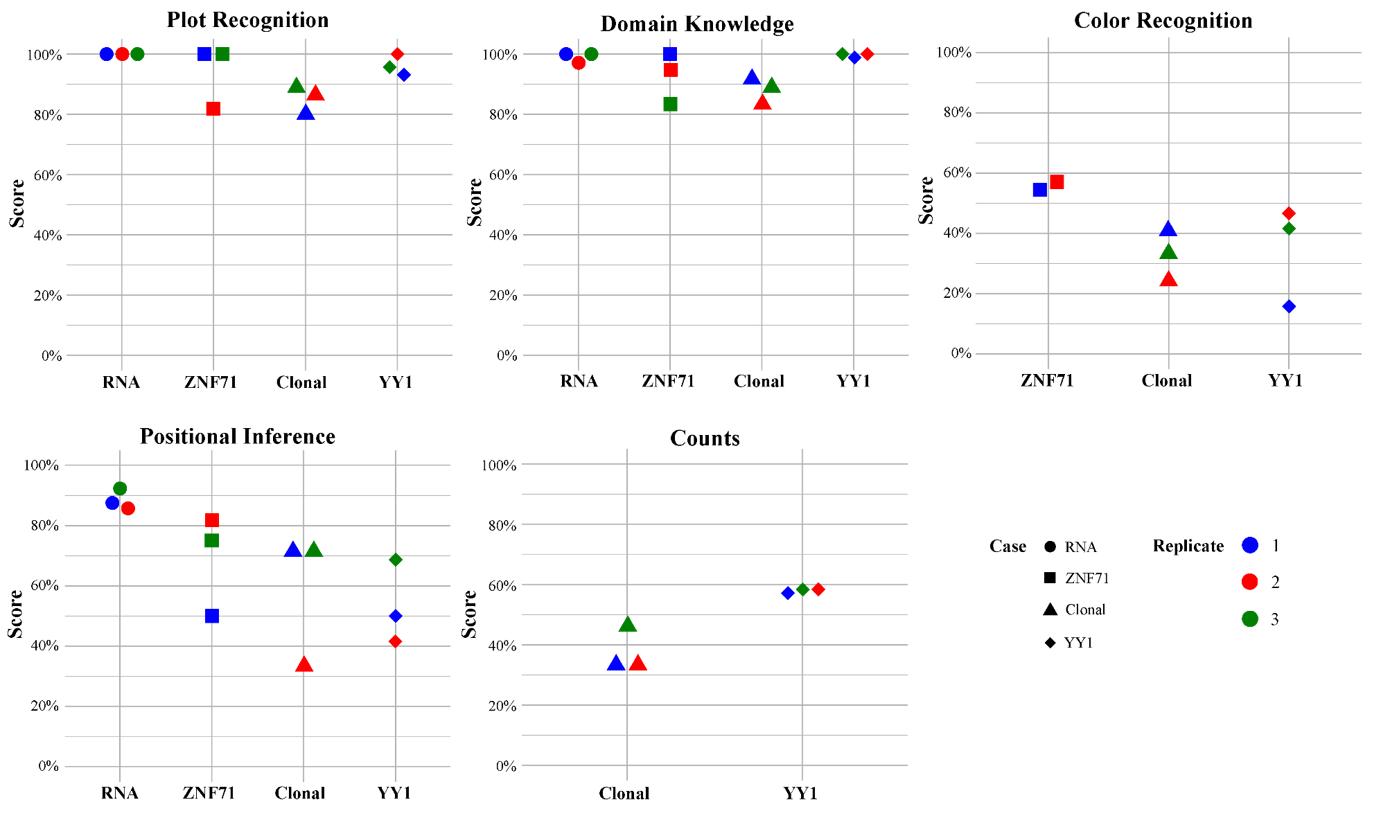

@article {wang2024scientific, title = {figuras científicas interpretadas por chatgpt: fortalezas en el reconocimiento de la trama y límites en la percepción del color}, autor = {wang, jinge y ye, qing y liu, li y guo, nancy lan y hu, gangqing}, Journal = {npj precision oncology}, volumen = {8}, número =}, n. Páginas = {84}, año = {2024}, Publisher = {Nature Publishing Group UK London}}