? Lesen Sie das vollständige Papier
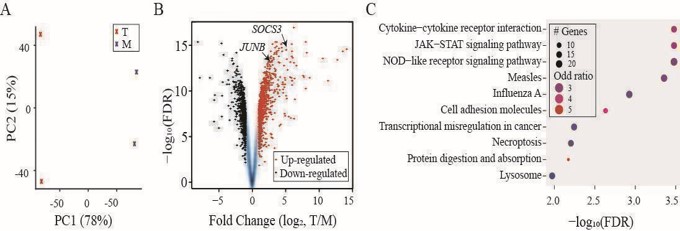
(A) PCA -Analyse mit zwei Probengruppen: „T“ in Rot und „M“ in Blau. (B) ein Vulkandiagramm für FCS und FDRs aus differentiell exprimierter Genanalyse mit hochregulierten Genen in roten und herunterregulierten Genen in Schwarz. SOCS3 und JUNB durch Pfeilspitzen angezeigt. (C) DOT-Diagramm, das die zehn Top-Treffer der Analyse der Kegg-Pfadanreicherung zusammenfasst, die auf die hochregulierten Gene angewendet wird.
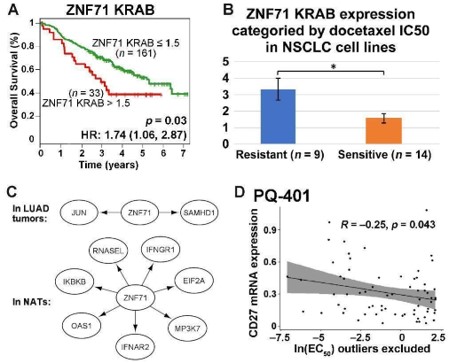
(A) Kaplan -Meier -Überlebensdiagramm für NSCLC -Patienten (GSE81089) sortiert durch die Expression von Znf71 KrAB -Isoform. Rot: Patienten mit einer höheren znf71 krab -Expression (TPM> 1,5); Grün: andere; (B) Ein Balkendiagramm für die Expression von Znf71 -Krab -Isoform zwischen NSCLC -Zelllinien, sortiert durch Docetaxel -Empfindlichkeit. (C) Genassoziationsnetzwerk von ZnF71- und IIIR -Genen in Luad -Tumoren und NATs. (D) Streudiagramm für das CD27-mRNA-Expressionsniveau und die Arzneimittelreaktion auf PQ-401 (EC50) zwischen NSCLS-Zelllinien.
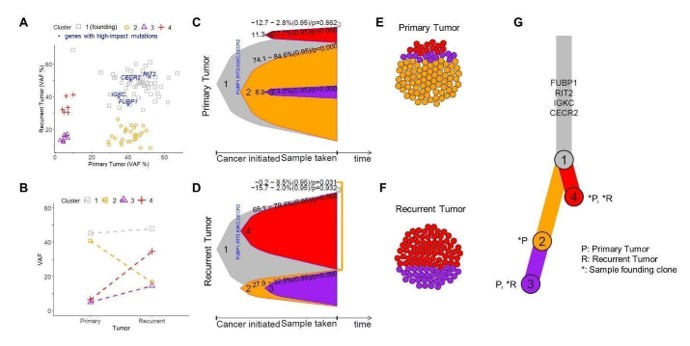
(A) Streudiagramm für VAF% kontrastierende wiederkehrende Tumor (y-Achse) zu Primärtumor (x-Achse). Jeder Punkt ist ein Gen und Gene mit signifikanten Auswirkungen sind angezeigt. Primäre und wiederkehrende Tumoren, die aus demselben Patienten stammen. (B) Verbundenes DOT -Diagramm, das die Veränderungen des mittleren VAF jedes Clusters vom Primärtumor zum Wiederkehrenden veranschaulicht. (C) Glockendiagramm, das die relative Prävalenz jedes (Sub-) Klons als Funktion des Krebsprogressions für Primärtumor und (d) wiederkehrender Tumor veranschaulicht. (E) Kreisverpackungsplotten, die die zelluläre Zusammensetzung des Primärtumors und (f) wiederkehrender Tumor veranschaulichen, untersucht mit insgesamt 100 Zellen. (G) Ein verzweigter Baum, der die relationalen und saatlichen Muster der (Sub-) Klone zwischen den primären und wiederkehrenden Proben veranschaulicht. Angegeben werden Gene, die das Wachstum des Gründungsklones erleichtern.
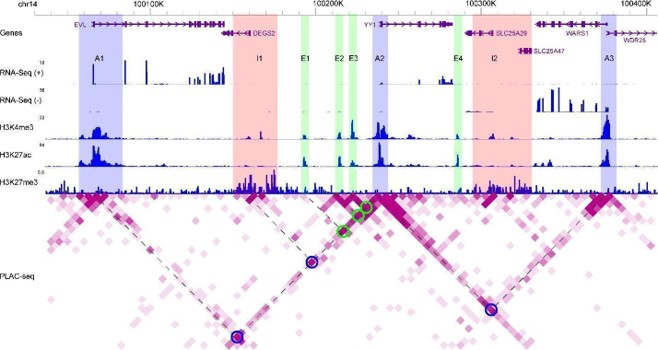
Washu-Epigenom-Browser-Tracks, die Verteilungen von Strangspezifischen RNA-Seq-Lesevorgängen, Chip-seq-Reads für H3K4Me3, H3K27AC und H3K27me3, sowie Plac-Seq-Lesevorgänge für Chromatin-Chromatin-Interaktionen veranschaulichen. Blaue Rechtecke (A1 bis A3): aktive Promotorregionen; Pink Rechtecke (I1 und I2): Inaktive Promoterregionen; Grüne Rechtecke (E1 bis E4): aktive Enhancer -Regionen; Blaue Kreise: Promotorregionen (A1, I1 und A3), die mit YY1 interagieren; Grüne Kreise: Enhancer -Regionen (E1 bis E3), die mit YY1 interagieren. Gestrichelte Linien, die mit einem Kreis auf der Heatmap geteilt werden: zwei interagierende Genomregionen. Grad der Purpie auf der Wärmemap, die die Wechselwirkungsstärke anzeigt.

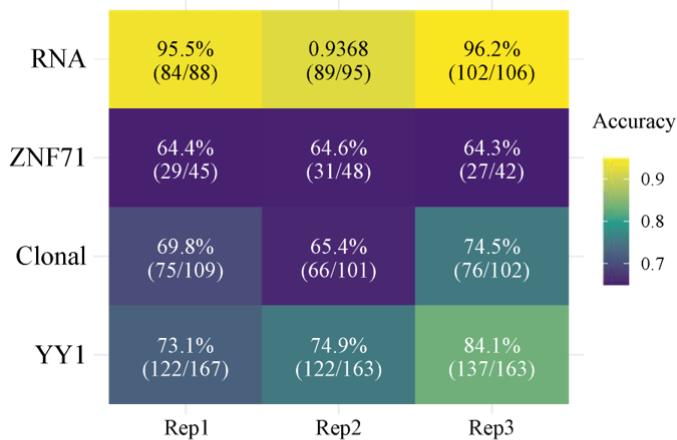
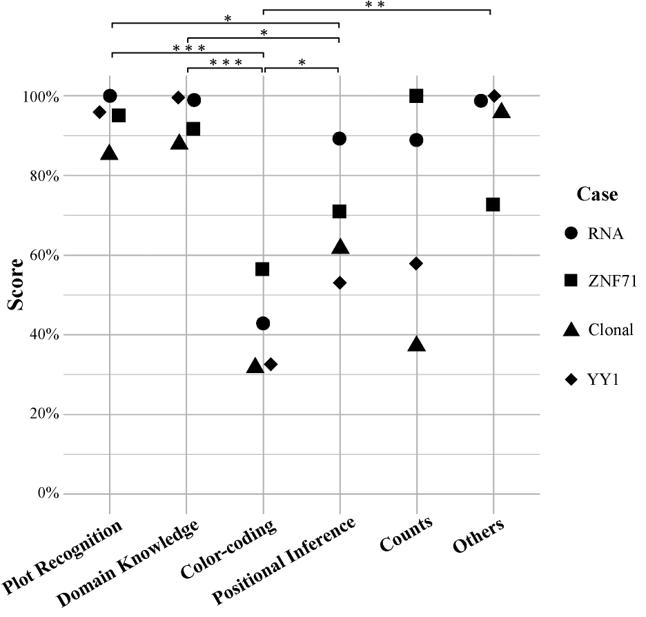
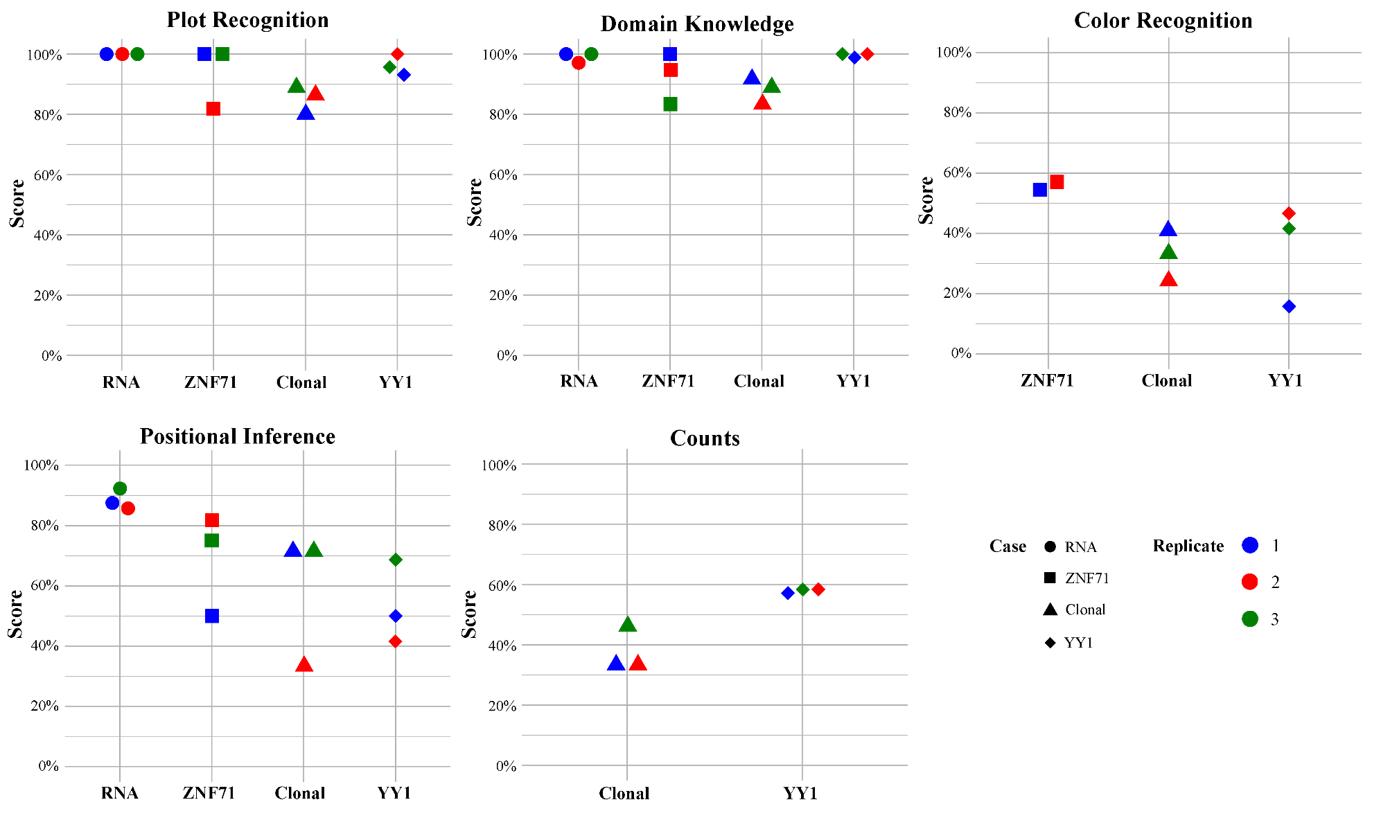

@article{wang2024scientific, title={Scientific figures interpreted by ChatGPT: strengths in plot recognition and limits in color perception}, author={Wang, Jinge and Ye, Qing and Liu, Li and Guo, Nancy Lan and Hu, Gangqing}, journal={NPJ Precision Oncology}, volume={8}, number={1}, Seiten = {84}, Jahr = {2024}, Publisher = {Nature Publishing Group UK London}}