pre_post_synthesis
1.0.0
Dans SPIE Medical Imaging 2024.
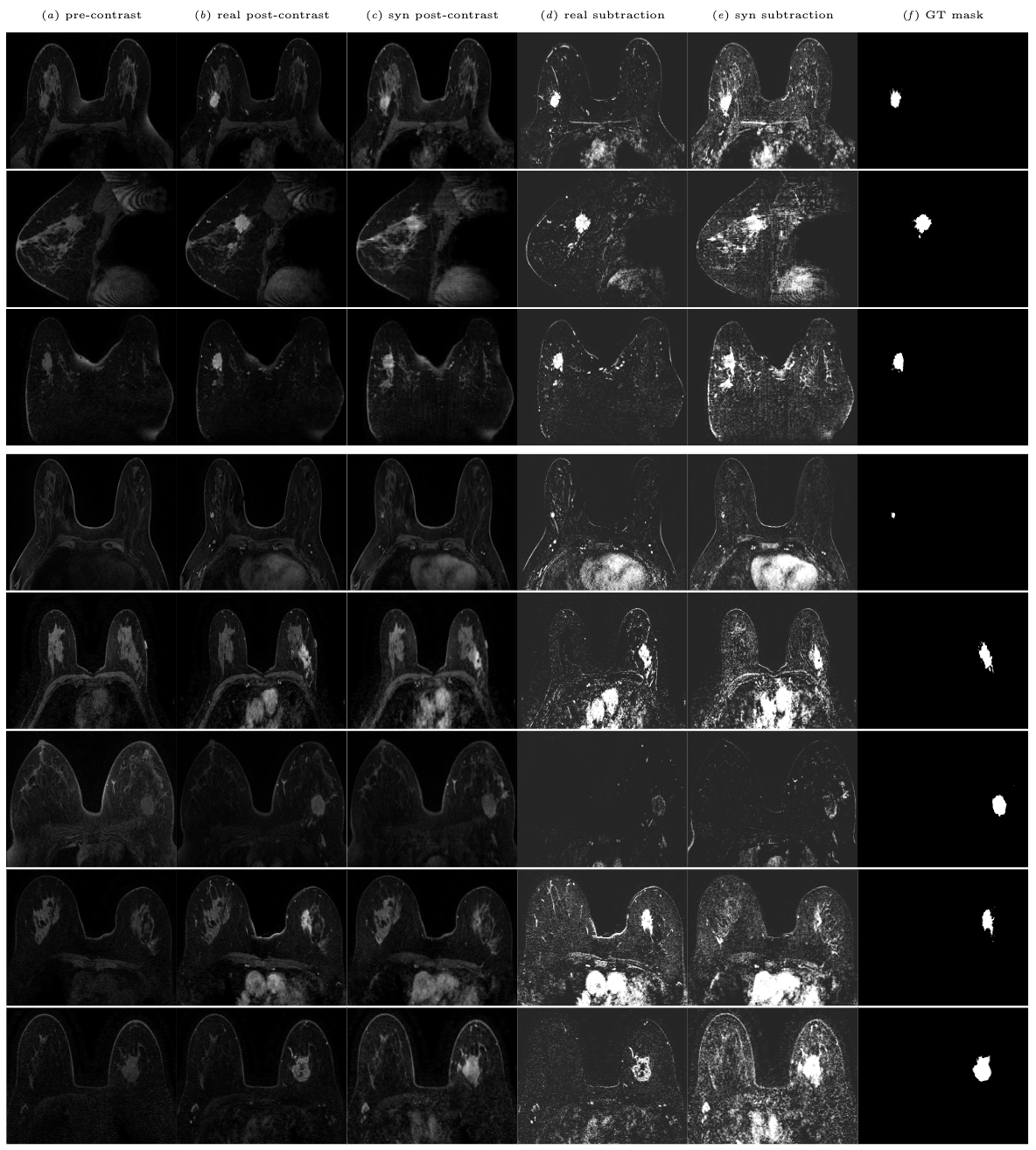
L'ensemble de données Duke utilisé dans cette étude est disponible sur l'archive d'imagerie du cancer (TCIA).
Vous pouvez trouver quelques exemples de fichiers nifti synthétiques en synthèse / exemples.
Les poids du modèle sont stockés sur Zenodo et mis à disposition via la bibliothèque Medigan.
Pour créer vos propres données post-contraste, exécutez simplement:
pip install medigan # import medigan and initialize Generators
from medigan import Generators
generators = Generators ()
# generate 10 samples with model 23 (00023_PIX2PIXHD_BREAST_DCEMRI).
# Also, auto-install required model dependencies.
generators . generate ( model_id = '00023_PIX2PIXHD_BREAST_DCEMRI' , num_samples = 10 , install_dependencies = True )Veuillez envisager de citer notre travail si vous l'avez trouvé utile pour vos recherches:
@article { osuala2023pre ,
title = { {Pre-to Post-Contrast Breast MRI Synthesis for Enhanced Tumour Segmentation} } ,
author = { Osuala, Richard and Joshi, Smriti and Tsirikoglou, Apostolia and Garrucho, Lidia and Pinaya, Walter HL and Diaz, Oliver and Lekadir, Karim } ,
journal = { arXiv preprint arXiv:2311.10879 } ,
year = { 2023 }
}Ce référentiel emprunte le code du PIX2PIXHD et des référentiels NNunet. Les 254 masques de segmentation tumorale utilisés dans cette étude ont été fournis par Caballo et al.