pre_post_synthesis
1.0.0
In Spie Medical Imaging 2024.
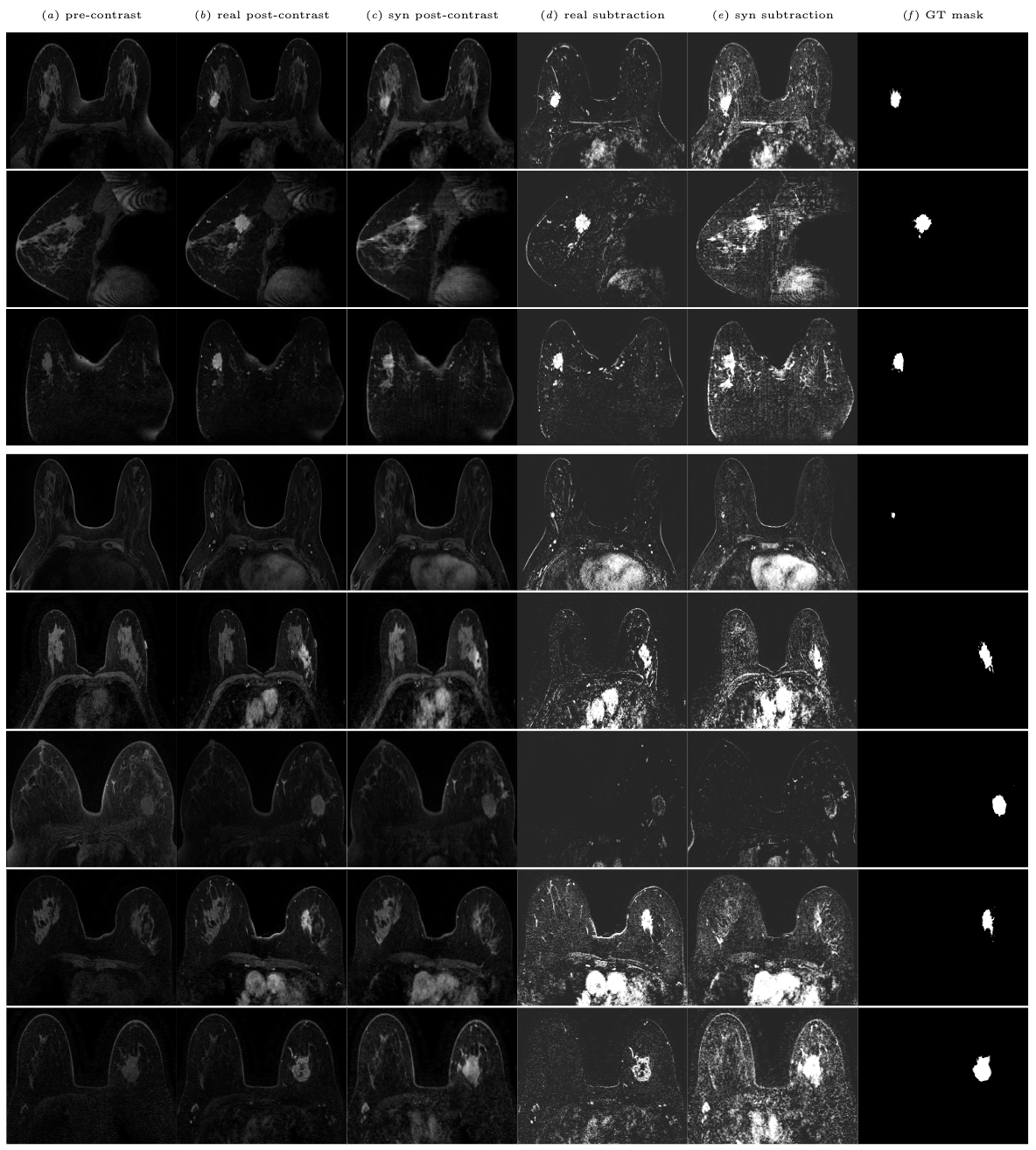
Der in dieser Studie verwendete Duke -Datensatz ist im Cancer Imaging Archive (TCIA) verfügbar.
Sie können einige Beispiele für synthetische NIFTI -Dateien in der Synthese/Beispiele finden.
Modellgewichte werden auf Zenodo aufbewahrt und über die Medigan -Bibliothek zur Verfügung gestellt.
Um Ihre eigenen Daten nach der Kontrast zu erstellen, leiten Sie einfach aus:
pip install medigan # import medigan and initialize Generators
from medigan import Generators
generators = Generators ()
# generate 10 samples with model 23 (00023_PIX2PIXHD_BREAST_DCEMRI).
# Also, auto-install required model dependencies.
generators . generate ( model_id = '00023_PIX2PIXHD_BREAST_DCEMRI' , num_samples = 10 , install_dependencies = True )Bitte erwägen Sie, unsere Arbeit zu zitieren, wenn Sie sie für Ihre Recherche als nützlich empfanden:
@article { osuala2023pre ,
title = { {Pre-to Post-Contrast Breast MRI Synthesis for Enhanced Tumour Segmentation} } ,
author = { Osuala, Richard and Joshi, Smriti and Tsirikoglou, Apostolia and Garrucho, Lidia and Pinaya, Walter HL and Diaz, Oliver and Lekadir, Karim } ,
journal = { arXiv preprint arXiv:2311.10879 } ,
year = { 2023 }
}Dieser Repository -Code aus dem PIX2PixHD und den Nnunet -Repositories. Die in dieser Studie verwendeten 254 Tumorsegmentierungsmasken wurden von Caballo et al.