Implementación de diferentes tipos de modelos de unlo para la segmentación de imágenes
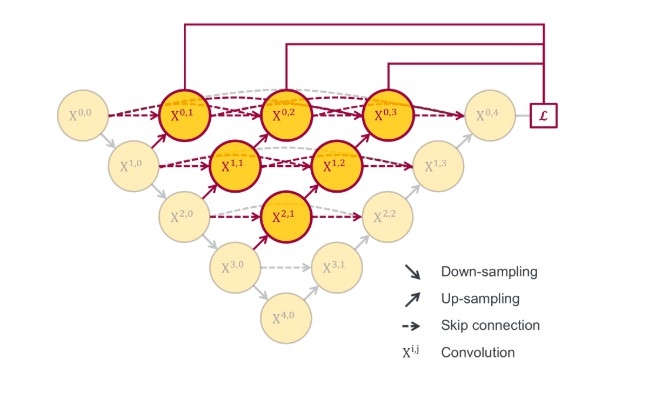
Unet - U -Net: redes convolucionales para la segmentación de imágenes biomédicas https://arxiv.org/abs/1505.04597
RCNN-Unet -Red neural convolucional residual recurrente basada en U-Net (R2U-NET) para la segmentación de imágenes médicas https://arxiv.org/abs/1802.06955
Atención Unet - Atención U -Net: Aprendiendo dónde buscar el páncreas https://arxiv.org/abs/1804.03999
RCNN-ATENCIÓN UNET -ATENCIÓN R2U-NET: Just Integración de dos obras avanzadas recientes (R2U-NET + Atención U-Net)
Con visualización de capa
Clon el repositorio:
git clone https://github.com/bigmb/Unet-Segmentation-Pytorch-Nest-of-Unets.git python>=3.6
torch>=0.4.0
torchvision
torchsummary
tensorboardx
natsort
numpy
pillow
scipy
scikit-image
sklearn
Instale todas las bibliotecas dependientes:
pip install -r requirements.txtAgregue todas sus carpetas a esta línea 106-113
t_data = '' # Input data
l_data = '' #Input Label
test_image = '' #Image to be predicted while training
test_label = '' #Label of the prediction Image
test_folderP = '' #Test folder Image
test_folderL = '' #Test folder Label for calculating the Dice score
Unena 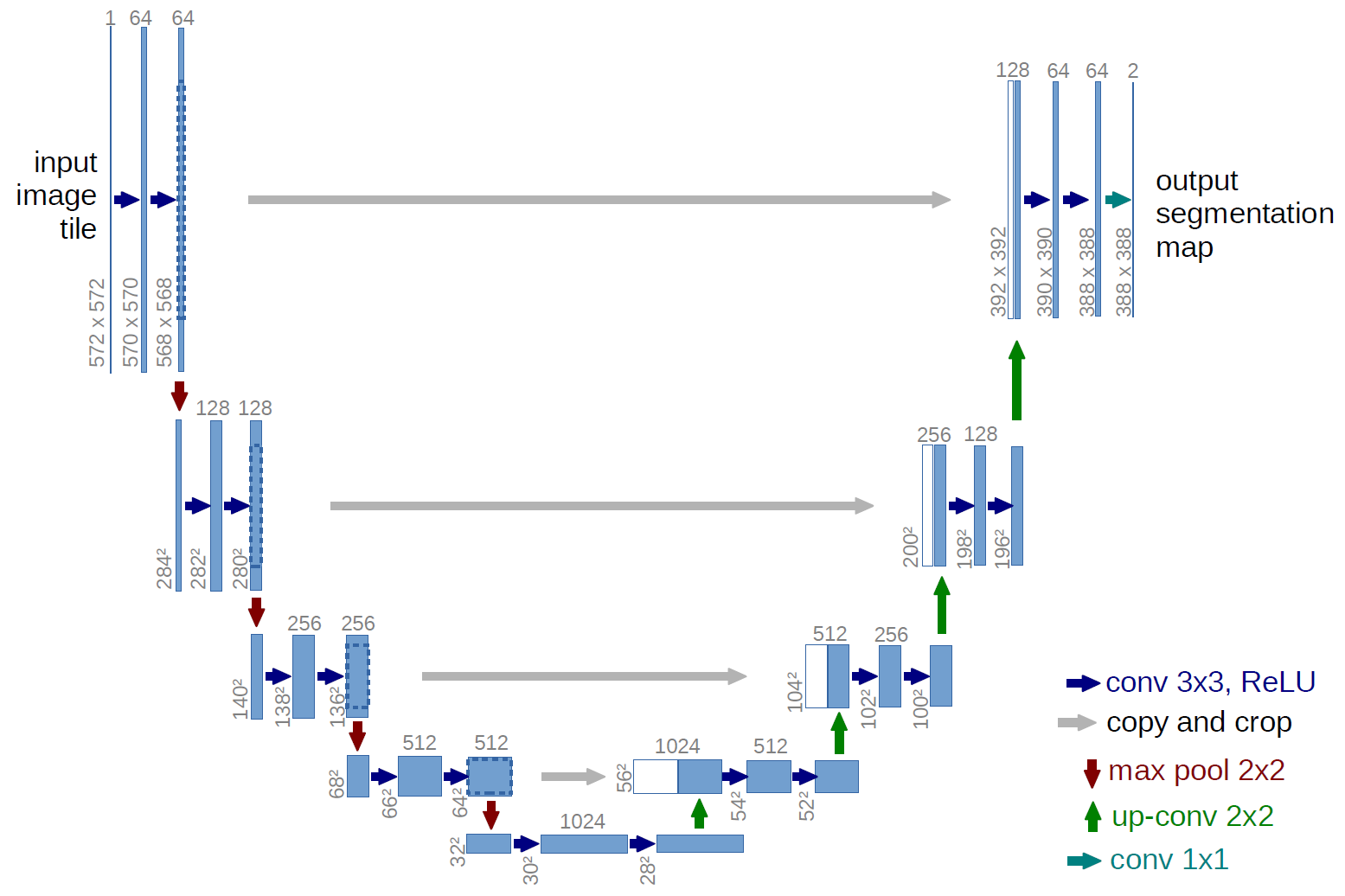
Rcnn unet 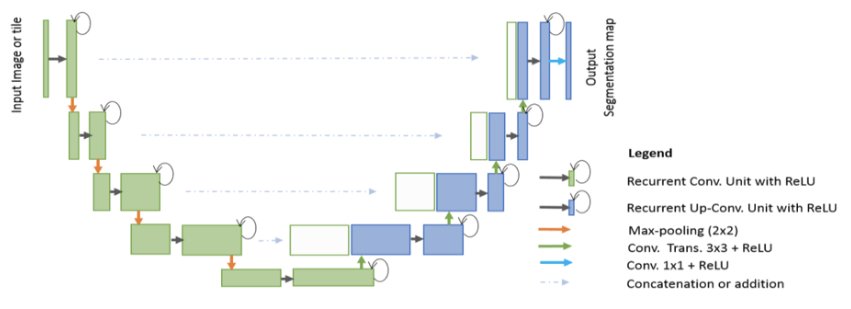
Atención un metro 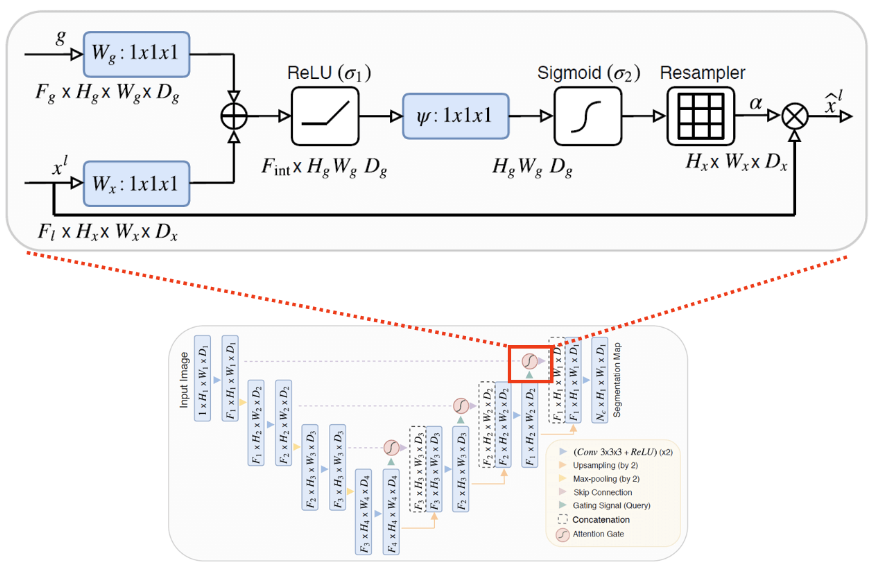
Atención-rcnn unt 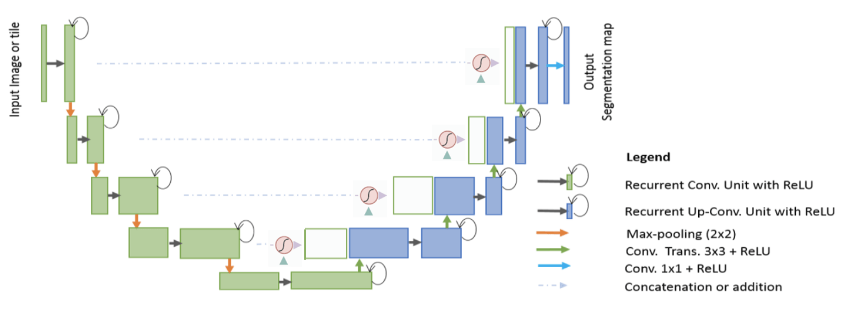
Unes
Para trazar la pérdida, se requeriría Visdom. El código ya está escrito, solo incomment la parte requerida. El flujo de gradiente también se puede usar. Tomado de (https://discuss.pytorch.org/t/check-cradient-flow-in-network/15063/10)
Se crea una carpeta de modelo y se almacenan todos los datos dentro de eso. La última capa se guardará en la carpeta del modelo. Si se requiere una capa particular, mencione en la línea 361.
Visulación de capas
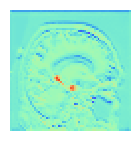
Visulación de filtro
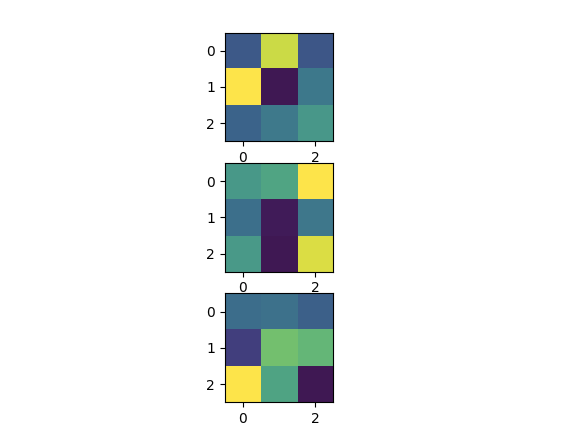
TensorBoardX todavía tiene que ajustar algunos parámetros para obtener visualización. Han confundido esto tratando de hacer que Pytorch 1.1.0 funcione directamente con TensorBoard (y luego llegué a saber que actualmente no admite nada aparte de los gráficos lineales) 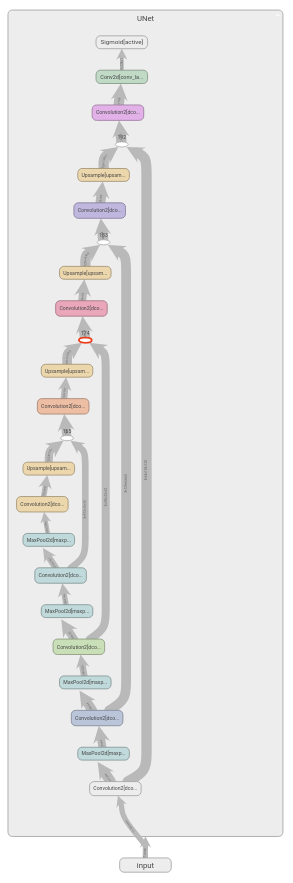
Visulización de la imagen de entrada para verificar
a) Imagen original
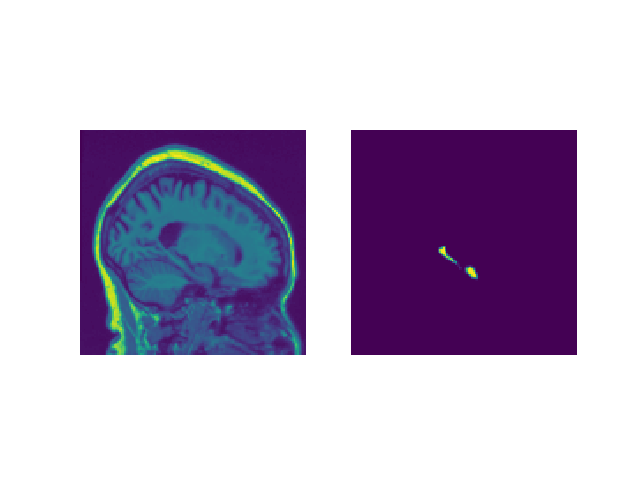
b) Imagen de CenterCrop

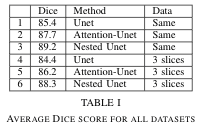
Puntuación de dados para la segmentación del hipocampo Adni-Loni DataSet
Si lo encuentra útil para su trabajo.
@article{DBLP:journals/corr/abs-1906-07160,
author = {Malav Bateriwala and
Pierrick Bourgeat},
title = {Enforcing temporal consistency in Deep Learning segmentation of brain
{MR} images},
journal = {CoRR},
volume = {abs/1906.07160},
year = {2019},
url = {http://arxiv.org/abs/1906.07160},
archivePrefix = {arXiv},
eprint = {1906.07160},
timestamp = {Mon, 24 Jun 2019 17:28:45 +0200},
biburl = {https://dblp.org/rec/bib/journals/corr/abs-1906-07160},
bibsource = {dblp computer science bibliography, https://dblp.org}
}
In progress