Implementierung verschiedener Arten von UNET -Modellen für die Bildsegmentierung
UNET - U -NET: Faltungsnetzwerke für biomedizinische Bildsegmentierung https://arxiv.org/abs/1505.04597
RCNN-UNET -RESAUTIERUNG VON RESAULE FALLE NEURALNELNELD BATTE AUF UNTER (R2U-NET) für die medizinische Bildsegmentierung https://arxiv.org/abs/1802.06955
Achtung Unet - Achtung U -NET: Lernen Sie, wo Sie nach der Bauchspeicheldrüse suchen können https://arxiv.org/abs/1804.03999
Rcnn-attierung unet -Achtung R2U-NET: Nur Integration von zwei aktuellen fortschrittlichen Werken (R2U-NET + Achtung U-NET)
Mit Schichtvisualisierung
Klonen Sie das Repo:
git clone https://github.com/bigmb/Unet-Segmentation-Pytorch-Nest-of-Unets.git python>=3.6
torch>=0.4.0
torchvision
torchsummary
tensorboardx
natsort
numpy
pillow
scipy
scikit-image
sklearn
Installieren Sie alle abhängigen Bibliotheken:
pip install -r requirements.txtFügen Sie alle Ihre Ordner dieser Zeile 106-113 hinzu
t_data = '' # Input data
l_data = '' #Input Label
test_image = '' #Image to be predicted while training
test_label = '' #Label of the prediction Image
test_folderP = '' #Test folder Image
test_folderL = '' #Test folder Label for calculating the Dice score
Unet 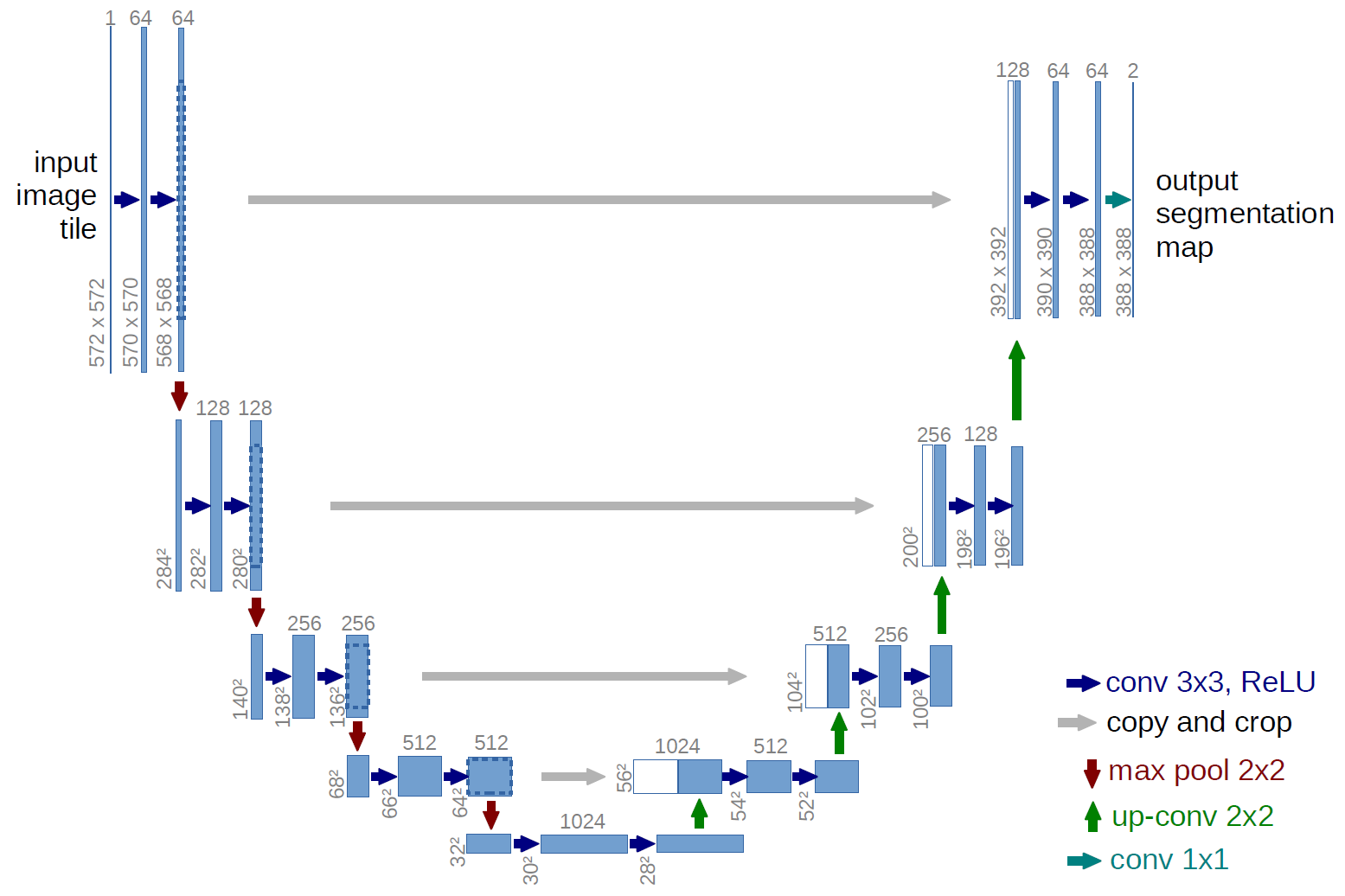
Rcnn unet 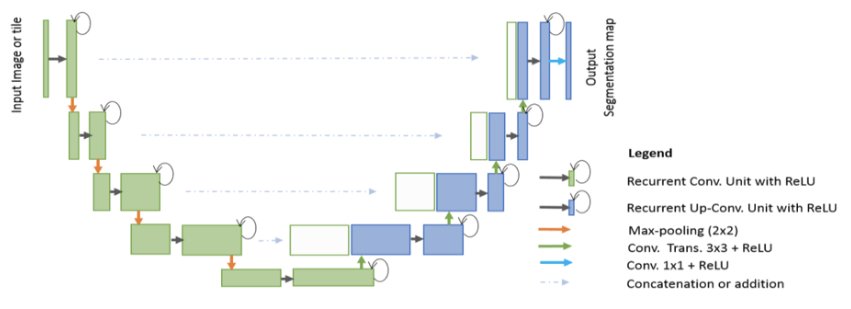
Aufmerksamkeit unet 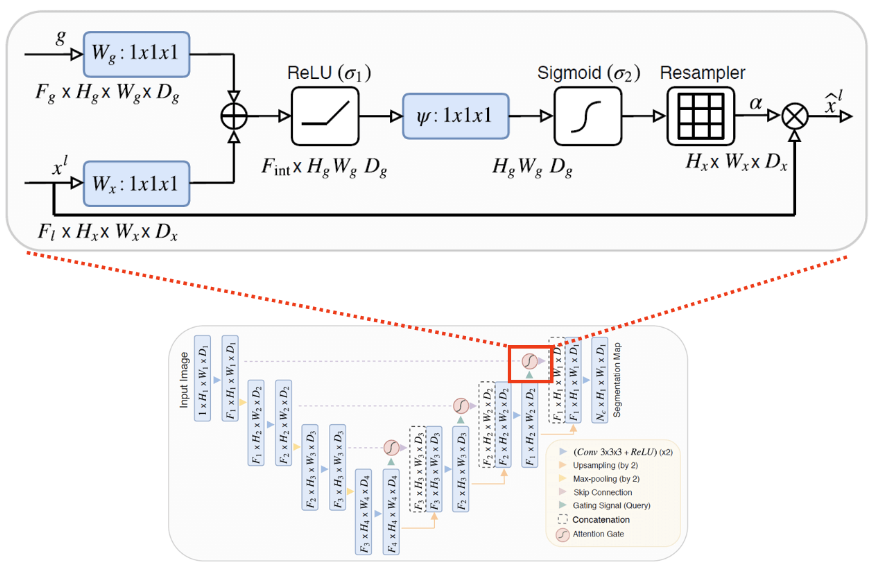
Achtung 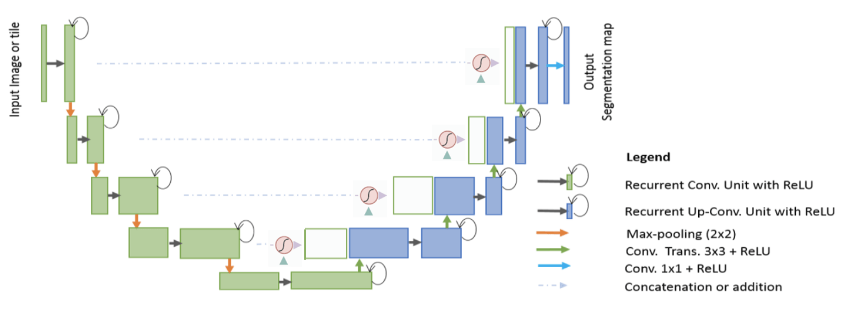
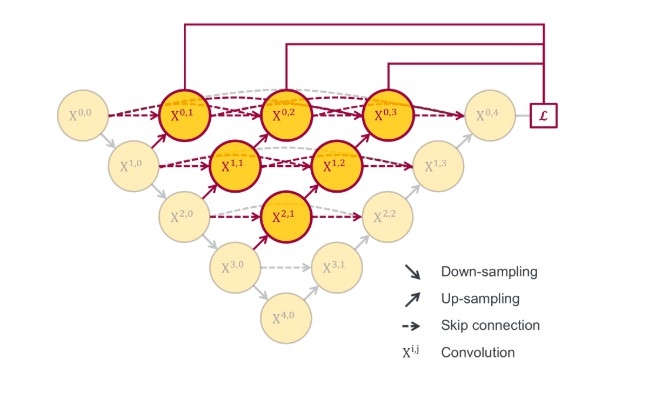
Verschachtelte unet
Um den Verlust zu zeichnen, wäre Visom erforderlich. Der Code ist bereits geschrieben, nur den erforderlichen Teil überzutragen. Gradientenfluss kann auch verwendet werden. Entnommen von (https://discuss.pytorch.org/t/check-gradient-flow-in-network/15063/10)
Es wird ein Modellordner erstellt und alle Daten werden darin gespeichert. Die letzte Ebene wird im Modellordner gespeichert. Wenn eine bestimmte Schicht erforderlich ist, erwähnen Sie sie in der Zeile 361.
Schichtvisulisierung
Filtervisulisierung
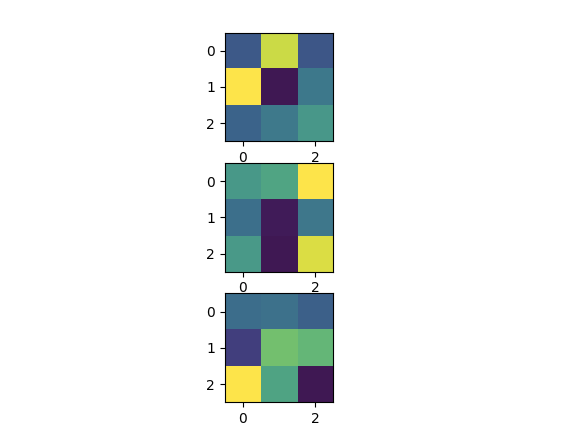
Tensorboardx muss noch einige Parameter optimieren, um eine Visualisierung zu erhalten. Ich habe diesen Versuch durcheinander gebracht, Pytorch 1.1.0 direkt mit Tensorboard zu machen (und dann zu erkennen, dass es derzeit nichts anderes unterstützt, außer lineare Graphen). 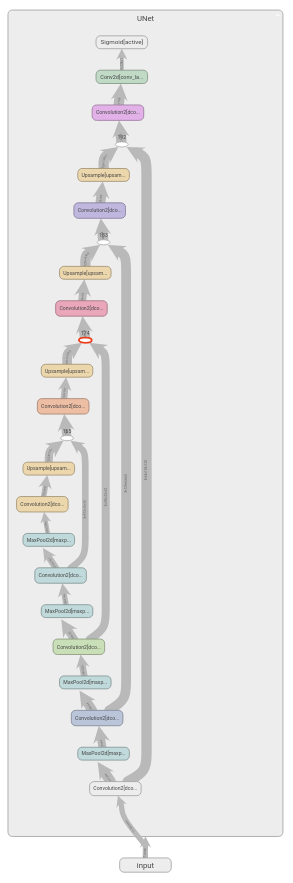
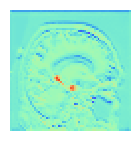
Eingabebildvisulisierung zur Überprüfung
a) Originalbild
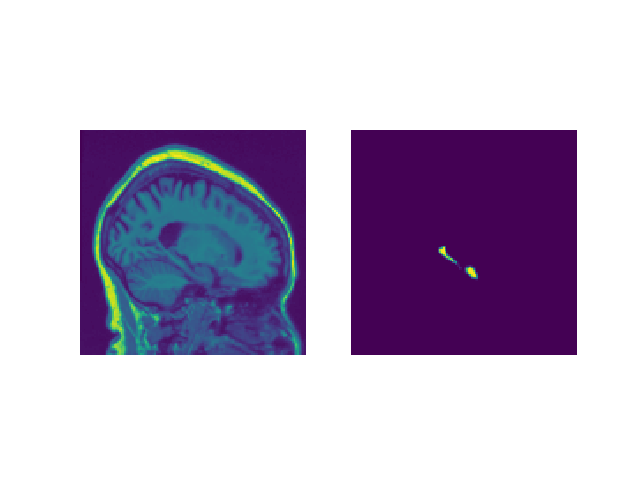
b) Centercrop -Bild

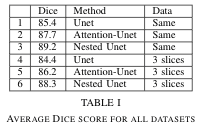
Würfelbewertung für Hippocampus-Segmentierung ADNI-LONI-Datensatz
Wenn Sie es für Ihre Arbeit nützlich finden.
@article{DBLP:journals/corr/abs-1906-07160,
author = {Malav Bateriwala and
Pierrick Bourgeat},
title = {Enforcing temporal consistency in Deep Learning segmentation of brain
{MR} images},
journal = {CoRR},
volume = {abs/1906.07160},
year = {2019},
url = {http://arxiv.org/abs/1906.07160},
archivePrefix = {arXiv},
eprint = {1906.07160},
timestamp = {Mon, 24 Jun 2019 17:28:45 +0200},
biburl = {https://dblp.org/rec/bib/journals/corr/abs-1906-07160},
bibsource = {dblp computer science bibliography, https://dblp.org}
}
In progress