
แพ็คเกจ Python ที่ใช้ในการจำลอง Neural Networks (SNNS) บนซีพียูหรือ GPU โดยใช้ฟังก์ชันการทำงานของ Pytorch Tensor
BindsNet เป็นห้องสมุดการจำลองเครือข่ายประสาทแบบ Spiking ซึ่งมุ่งเน้นไปที่การพัฒนาอัลกอริทึมที่ได้รับแรงบันดาลใจทางชีวภาพสำหรับการเรียนรู้ของเครื่อง
แพ็คเกจนี้ใช้เป็นส่วนหนึ่งของการวิจัยอย่างต่อเนื่องเกี่ยวกับการใช้ SNNs, การเรียนรู้ของเครื่อง (ML) และปัญหาการเรียนรู้การเสริมแรง (RL) ในห้องปฏิบัติการระบบประสาทและพลวัตที่ได้รับแรงบันดาลใจทางชีวภาพ
ตรวจสอบตัวอย่าง BindSnet สำหรับการรวบรวมการทดลองฟังก์ชั่นสำหรับการวิเคราะห์ผลลัพธ์แผนการทดลองผลการทดลองและอื่น ๆ เอกสารสำหรับแพ็คเกจสามารถดูได้ที่นี่

เพื่อติดตั้งรีลีสที่เสถียรล่าสุดจากที่เก็บ GitHub
pip install git+https://github.com/BindsNET/bindsnet.git
หรือเพื่อสร้างแพ็คเกจ bindsnet จากแหล่งที่มาโคลนที่เก็บ GitHub เปลี่ยนไดเรกทอรีเป็นระดับสูงสุดของโครงการนี้และออก
pip install .
หรือในการติดตั้งในโหมดแก้ไขได้ (อนุญาตให้มีการปรับเปลี่ยนแพ็คเกจโดยไม่ต้องติดตั้งใหม่):
pip install -e .
ในการติดตั้งแพ็คเกจที่จำเป็นในการเชื่อมต่อกับ OpenAI Gym Environment Library ให้ทำตามคำแนะนำสำหรับการติดตั้งแพ็คเกจที่จำเป็นในการเรียกใช้ตัวจำลองสภาพแวดล้อม RL (บน Linux / MacOS)
ลิงก์ไปยังที่เก็บ Docker
นอกจากนี้เรายังให้บริการ DockerFile ที่ BindSnet และการพึ่งพาทั้งหมดติดตั้งในปัญหา
docker build .
ที่ไดเรกทอรีระดับบนสุดของโครงการนี้เพื่อสร้างภาพนักเทียบท่า
หากต้องการเปลี่ยนชื่อของภาพที่สร้างขึ้นใหม่ปัญหา
docker tag <IMAGE_ID> <NEW_IMAGE_ID>
ในการเรียกใช้คอนเทนเนอร์และรับเทอร์มินัลทุบตีภายในออกให้ออก
docker run -it <NEW_IMAGE_ID> bash
ในการเรียกใช้การจำลองแบบใกล้กับบทความนี้ปัญหา
cd examples/mnist
python eth_mnist.py
มีอาร์กิวเมนต์บรรทัดคำสั่งทางเลือกจำนวนมากซึ่งสามารถส่งผ่านได้รวมถึง --plot (แสดงตัวเลขการตรวจสอบที่มีประโยชน์), --n_neurons [int] (จำนวน excitatory, เซลล์ประสาทยับยั้งจำลอง), --mode ['train' | 'test'] (ตั้งค่าการดำเนินงานเครือข่ายเป็นขั้นตอนการฝึกอบรมหรือการทดสอบ) และอื่น ๆ เรียกใช้สคริปต์ด้วย --help หรือ -h Flag สำหรับข้อมูลเพิ่มเติม
มีตัวอย่างอื่น ๆ อีกมากมายในไดเรกทอรี examples ที่มีไว้เพื่อแสดงฟังก์ชั่นของ BindSnet ลองดูและแจ้งให้เราทราบว่าคุณคิดอย่างไร!
ออกต่อไปนี้เพื่อเรียกใช้การทดสอบ:
python -m pytest test/
การทดสอบบางอย่างจะล้มเหลวหากไม่ได้ติดตั้ง AI gym บนเครื่องของคุณ
การจำลองการเปลี่ยนแปลงของเซลล์ประสาท spiking ที่เป็นไปได้ทางชีวภาพอาจเป็นสิ่งที่ท้าทาย โดยทั่วไปแล้วจะทำโดยการแก้สมการเชิงอนุพันธ์สามัญ (ODEs) ซึ่งอธิบายถึงการเปลี่ยนแปลงดังกล่าว Pytorch ไม่สนับสนุนการแก้ปัญหาของสมการเชิงอนุพันธ์อย่างชัดเจน (ตรงข้ามกับ brian2 เป็นต้น) แต่เราสามารถแปลง ODEs ที่กำหนดพลวัตเป็นสมการที่แตกต่างและแก้ปัญหาได้ในช่วงเวลาสั้น ๆ ( dt ตามลำดับ 1 มิลลิวินาที) เป็นการประมาณ แน่นอนภายใต้ประทุนแพ็คเกจอย่าง brian2 กำลังทำสิ่งเดียวกัน การทำสิ่งนี้ใน PyTorch นั้นน่าตื่นเต้นด้วยเหตุผลบางประการ:
เราสามารถใช้คบเพลิงที่ทรงพลังและยืดหยุ่นวัตถุ torch.Tensor ซึ่งเป็น wrapper รอบ ๆ numpy.ndarray ซึ่งสามารถถ่ายโอนไปยังและจากอุปกรณ์ GPU
เราสามารถหลีกเลี่ยง "การสร้างวงล้อใหม่" โดยการเปลี่ยนฟังก์ชั่นจาก torch.nn.functional Pytorch Submodule ในสถาปัตยกรรม SNN ของเรา เช่นฟังก์ชั่น convolution หรือ pooling
แนวคิดที่ว่าการสั่งซื้อของเซลล์ประสาทสไปค์และการเข้ารหัสเวลาที่สัมพันธ์กันของพวกเขาเป็นธีมหลักในด้านประสาทวิทยาศาสตร์ Markram และคณะ (1997) เสนอว่า synapses ระหว่างเซลล์ประสาทควรเสริมสร้างหรือลดลงตามเวลาที่สัมพันธ์กันนี้และก่อนหน้านั้น Donald Hebb เสนอทฤษฎีการเรียนรู้ของเฮบเบียมักจะระบุว่า "เซลล์ประสาทที่ยิงเข้าด้วยกัน การขยายทฤษฎี Hebbian ของ Markram et al. เป็นที่รู้จักกันในชื่อพลาสติกพลาสติกที่ขึ้นอยู่กับสไปค์ (STDP)
เราสนใจที่จะใช้ SNNs กับปัญหา ML และ RL เราใช้ STDP เพื่อแก้ไขน้ำหนักของ synapses ที่เชื่อมต่อคู่หรือประชากรของเซลล์ประสาทใน SNNs ในบริบทของ ML เราต้องการเรียนรู้การตั้งค่าน้ำหนัก synapse ซึ่งจะสร้างกิจกรรม spiking ที่ขึ้นกับข้อมูลใน SNNs กิจกรรมนี้จะช่วยให้เราสามารถทำภารกิจ ML ที่น่าสนใจได้ในภายหลัง เช่นการแยกแยะหรือการจัดกลุ่มข้อมูลอินพุต ในบริบทของ RL เราอาจนึกถึงเครือข่ายประสาท spiking ในฐานะตัวแทน RL ซึ่งกิจกรรม spiking อาจถูกแปลงเป็นการกระทำในพื้นที่การกระทำของสภาพแวดล้อม
เราได้จัดเตรียมสคริปต์เริ่มต้นอย่างง่าย ๆ สำหรับการเรียนรู้ที่ไม่ได้รับการดูแล (เรียนรู้การเป็นตัวแทนที่เชื่อมต่ออย่างเต็มที่หรือ convolutional ผ่าน STDP) การเรียนรู้ภายใต้การดูแล
เราจำลองเครือข่ายที่มีประชากรของเซลล์ประสาทอินพุต N Poisson ด้วยอัตราการยิง (ใน Hertz) แบบสุ่มจาก U (0, 100) เชื่อมต่อกับทุกคนกับจำนวนประชากรที่มีขนาดเท่ากันของเซลล์ประสาทรวมและไฟ) ที่มีน้ำหนักการเชื่อมต่อตัวอย่างจาก N (0,1) เราเปลี่ยนแปลง N อย่างเป็นระบบจาก 250 ถึง 10,000 ในขั้นตอนที่ 250 และดำเนินการจำลองแต่ละครั้งกับทุกไลบรารีสำหรับ 1,000ms ด้วยความละเอียดเวลา DT = 1.0 เราทดสอบ bindsNet (ด้วยการคำนวณ CPU และ GPU), Brian2, Pynest (อินเทอร์เฟซ Python ไปยังอินเตอร์เฟส Nest SLI ที่เรียกใช้ C ++ Nest Core Simulator), Annarchy (ด้วยการคำนวณ CPU และ GPU) และ Brian2Genn
แพ็คเกจหลายอย่างรวมถึง Brian และ Pynest อนุญาตให้มีการตั้งค่าการตั้งค่าทั่วโลกบางอย่าง เช่นจำนวนเธรด CPU จำนวนกระบวนการ OpenMP ฯลฯ เราเลือกการตั้งค่าเหล่านี้สำหรับการศึกษาเกณฑ์มาตรฐานของเราในความพยายามที่จะเพิ่มความเร็วของห้องสมุดให้สูงสุด แต่โปรดทราบว่า BindSNET ไม่จำเป็นต้องมีการตั้งค่าตัวเลือกดังกล่าว วิธีการของเราที่สืบทอดรูปแบบการคำนวณของ Pytorch ดูเหมือนจะใช้ประโยชน์จากฮาร์ดแวร์ที่มีอยู่ให้ดีที่สุดและทำให้ง่ายสำหรับผู้ปฏิบัติงานที่จะได้รับประสิทธิภาพที่ดีที่สุดจากระบบของพวกเขาด้วยความพยายามน้อยที่สุด
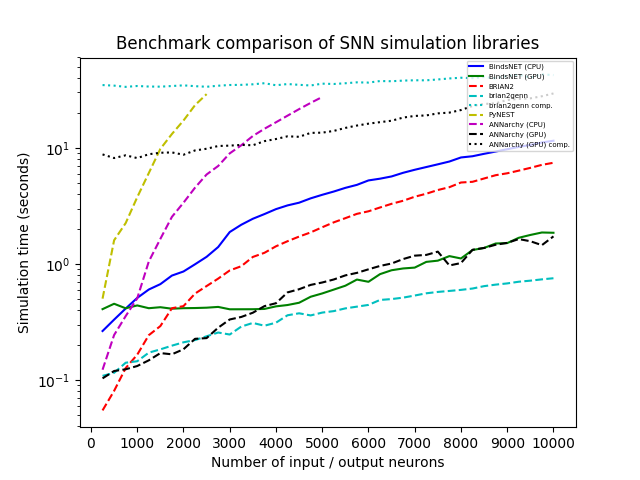
การจำลองทั้งหมดทำงานบน Ubuntu 16.04 LTS กับ Intel (R) Xeon (R) CPU E5-2687W V3 @ 3.10GHz, 128GB RAM @ 2133MHz และ GEFEFERCE GTX TITAN X (GM200) GPU Python 3.6 ใช้ในทุกกรณี เวลานาฬิกาถูกบันทึกสำหรับการจำลองการจำลองแต่ละครั้ง
หากคุณใช้ bindsNet ในการวิจัยของคุณโปรดอ้างอิงบทความต่อไปนี้:
@ARTICLE{10.3389/fninf.2018.00089,
AUTHOR={Hazan, Hananel and Saunders, Daniel J. and Khan, Hassaan and Patel, Devdhar and Sanghavi, Darpan T. and Siegelmann, Hava T. and Kozma, Robert},
TITLE={BindsNET: A Machine Learning-Oriented Spiking Neural Networks Library in Python},
JOURNAL={Frontiers in Neuroinformatics},
VOLUME={12},
PAGES={89},
YEAR={2018},
URL={https://www.frontiersin.org/article/10.3389/fninf.2018.00089},
DOI={10.3389/fninf.2018.00089},
ISSN={1662-5196},
}
ทำด้วย contrib.rocks
GNU Affero ทั่วไปใบอนุญาตสาธารณะ v3.0