
Un package Python utilisé pour simuler des réseaux de neurones à pointe (SNN) sur des CPU ou des GPU en utilisant la fonctionnalité Tensor Pytorch.
Bindsnet est une bibliothèque de simulation de réseau neuronal à pointe axée sur le développement d'algorithmes d'inspiration biologique pour l'apprentissage automatique.
Ce package est utilisé dans le cadre de la recherche en cours sur l'application des problèmes SNN, d'apprentissage automatique (ML) et d'apprentissage par renforcement (RL) dans le laboratoire des systèmes neuronaux et dynamiques) inspirés biologiquement (BINDS) et le Allen Discovery Center de l'Université Tufts.
Consultez les exemples de Bindsnet pour une collection d'expériences, des fonctions pour l'analyse des résultats, des graphiques de résultats d'expérience, etc. La documentation du package est disponible ici.

Pour installer la version stable la plus récente du référentiel GitHub
pip install git+https://github.com/BindsNET/bindsnet.git
Ou, pour construire le package bindsnet à partir de la source, clonez le référentiel GitHub, modifiez le répertoire au niveau supérieur de ce projet et émettez
pip install .
Ou, pour installer en mode modifiable (permet la modification du package sans réinstaller):
pip install -e .
Pour installer les packages nécessaires pour s'interfacer avec la bibliothèque Openai Gym RL Environments, suivez leurs instructions pour l'installation des packages nécessaires pour exécuter le simulateur des environnements RL (sur Linux / MacOS).
Lien vers le référentiel docker.
Nous fournissons également un dockerfile dans lequel BindsNet et toutes ses dépendances sont installés.
docker build .
au répertoire supérieur de ce projet pour créer une image Docker.
Pour changer le nom de l'image nouvellement construite, problème
docker tag <IMAGE_ID> <NEW_IMAGE_ID>
Pour exécuter un conteneur et obtenir un terminal bash à l'intérieur, problème
docker run -it <NEW_IMAGE_ID> bash
Pour exécuter une quasi-réplication du SNN de ce document,
cd examples/mnist
python eth_mnist.py
Il existe un certain nombre d'arguments de ligne de commande facultatifs qui peuvent être transmis, y compris --plot (affiche des chiffres de surveillance utiles), --n_neurons [int] (nombre de neurones inhibiteurs excitateurs simulés), --mode ['train' | 'test'] (définit le fonctionnement du réseau à la phase de formation ou de test), et plus encore. Exécutez le script avec l'indicateur --help ou -h pour plus d'informations.
Un certain nombre d'autres exemples sont disponibles dans le répertoire examples qui sont destinés à présenter la fonctionnalité de BindsNet. Jetez un œil et dites-nous ce que vous en pensez!
Insérer ce qui suit pour exécuter les tests:
python -m pytest test/
Certains tests échoueront si gym AI ouvert n'est pas installé sur votre machine.
La simulation de la dynamique des neurones à dopage biologiquement plausible peut être difficile. Cela se fait généralement en résolvant des équations différentielles ordinaires (ODE) qui décrivent ladite dynamique. Pytorch ne supporte pas explicitement la solution d'équations différentielles (par opposition à brian2 , par exemple), mais nous pouvons convertir les ODE définissant la dynamique en équations de différence et les résoudre à des intervalles courts réguliers (un dt à l'ordre de 1 milliseconde) comme approximation. Bien sûr, sous le capot, des forfaits comme brian2 font la même chose. Faire cela à PyTorch est passionnant pour plusieurs raisons:
Nous pouvons utiliser l'objet torch.Tensor puissant et flexible, un wrapper autour du numpy.ndarray qui peut être transféré vers et depuis les appareils GPU.
Nous pouvons éviter de «réinventer la roue» en réutilisant les fonctions du sous-module pytorch torch.nn.functional . Par exemple, les fonctions de convolution ou de mise en commun.
Le concept selon lequel l'ordre des pointes des neurones et leur renseignement de synchronisation relative sont un thème central dans les neurosciences. Markram et al. (1997) ont proposé que les synapses entre les neurones se renforcent ou se dégradent en fonction de ce timing relatif, et avant cela, Donald Hebb a proposé la théorie de l'apprentissage hébbien, souvent simplement énoncées comme "des neurones qui tirent ensemble, câlinent ensemble". L'extension de Markram et al. De la théorie hébbienne est connue sous le nom de plasticité dépendante du temps de pointes (STDP).
Nous souhaitons appliquer des SNN aux problèmes ML et RL. Nous utilisons STDP pour modifier les poids des synapses reliant les paires ou les populations de neurones dans les SNN. Dans le contexte de ML, nous voulons apprendre un cadre de poids de synapse qui générera une activité de dopage dépendante des données dans SNNS. Cette activité nous permettra d'effectuer par la suite une tâche d'intérêt ML; par exemple, discriminant ou clustering les données d'entrée. Dans le contexte de la RL, nous pouvons considérer le réseau de neurones à pointe comme un agent de RL, dont l'activité de dopage peut être convertie en actions dans l'espace d'action d'un environnement.
Nous avons fourni des scripts de démarrage simples pour faire de l'apprentissage non supervisé (apprentissage d'une représentation entièrement connectée ou convolutionnelle via STDP), un apprentissage supervisé (serrage des neurones de sortie vers le comportement de dopage souhaité en fonction des labels de données) et un apprentissage de renforcement (convertissant les observations de l'espace de jeu Atari Invaders en entrée dans un SNN et convertissant l'activité du réseau en actions dans le jeu).
Nous avons simulé un réseau avec une population de n neurones d'entrée de Poisson avec des taux de tir (dans Hertz) tirés au hasard de U (0, 100), connecté tout à tout à tous avec une population de neurones d'intégration et de tir (LIF) de taille égale, avec des poids de connexion échantillonnés à partir de N (0,1). Nous avons varié N systématiquement de 250 à 10 000 par étapes de 250 et avons effectué chaque simulation avec chaque bibliothèque pendant 1 000 ms avec une résolution de temps dt = 1,0. Nous avons testé BindsNet (avec CPU et Calcul GPU), Brian2, Pynest (l'interface Python avec l'interface NEST SLI qui exécute le simulateur de noyau de nid C ++), Anchyy (avec CPU et calcul GPU) et Brian2Genn (la frontale Brian2 au Simulator GENN).
Plusieurs packages, dont Brian et Pynest, permettent le réglage de certaines préférences mondiales; Par exemple, le nombre de threads CPU, le nombre de processus OpenMP, etc. Nous avons choisi ces paramètres pour notre étude de référence pour tenter de maximiser la vitesse de chaque bibliothèque, mais notez que BindsNet ne nécessite aucun réglage de ces options. Notre approche, héritant du modèle de calcul de Pytorch, semble faire le meilleur usage du matériel disponible, et permet donc aux praticiens d'obtenir les meilleures performances de leur système avec le moindre effort.
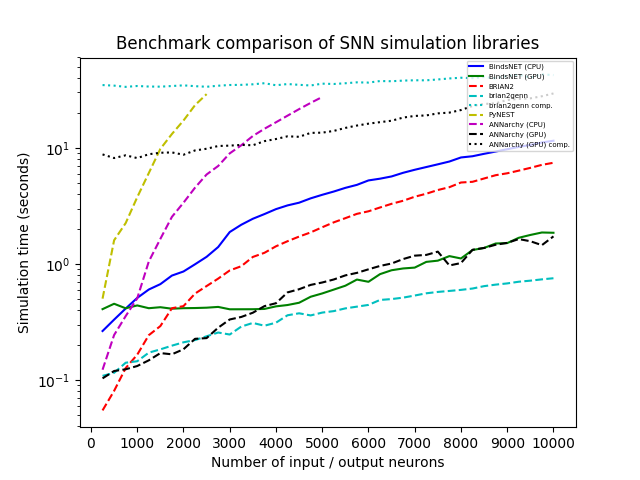
Toutes les simulations fonctionnent sur Ubuntu 16.04 LTS avec Intel (R) Xeon (R) CPU E5-2687W V3 @ 3,10 GHz, 128 Go RAM @ 2133MHz et deux GPU Geforce GTX Titan X (GM200). Python 3.6 est utilisé dans tous les cas. Le temps d'horloge a été enregistré pour chaque exécution de simulation.
Si vous utilisez Bindsnet dans vos recherches, veuillez citer l'article suivant:
@ARTICLE{10.3389/fninf.2018.00089,
AUTHOR={Hazan, Hananel and Saunders, Daniel J. and Khan, Hassaan and Patel, Devdhar and Sanghavi, Darpan T. and Siegelmann, Hava T. and Kozma, Robert},
TITLE={BindsNET: A Machine Learning-Oriented Spiking Neural Networks Library in Python},
JOURNAL={Frontiers in Neuroinformatics},
VOLUME={12},
PAGES={89},
YEAR={2018},
URL={https://www.frontiersin.org/article/10.3389/fninf.2018.00089},
DOI={10.3389/fninf.2018.00089},
ISSN={1662-5196},
}
Fait avec contribution.
Licence publique générale de GNU Affero v3.0