
Ein Python -Paket, das zum Simulieren von Spike -Nervennetzwerken (SNNs) auf CPUs oder GPUs mithilfe von Pytorch Tensor -Funktionalität verwendet wird.
Bindsnet ist eine Spike -Simulationsbibliothek für neuronale Netzwerke, die auf die Entwicklung biologisch inspirierter Algorithmen für maschinelles Lernen ausgerichtet ist.
Dieses Paket wird im Rahmen der laufenden Forschung zur Anwendung von SNNs, maschinellem Lernen (ML) und Verstärkungslernen (RL) in dem biologisch inspirierten Labor für neuronale und dynamische Systeme (Binds) und im Allen Discovery Center der Tufts University verwendet.
Schauen Sie sich die BindsNet -Beispiele für eine Sammlung von Experimenten, Funktionen zur Analyse der Ergebnisse, Diagramme der Experimentergebnisse und vielem mehr an. Die Dokumentation für das Paket finden Sie hier.

So installieren Sie die neueste stabile Version aus dem Github -Repository
pip install git+https://github.com/BindsNET/bindsnet.git
Oder um das bindsnet -Paket aus Quelle zu erstellen, das Github -Repository klonen, das Verzeichnis auf die oberste Ebene dieses Projekts ändern und ausgeben
pip install .
Oder um im bearbeitbaren Modus zu installieren (ermöglicht eine Änderung des Pakets ohne Neuinstallation):
pip install -e .
Um die Pakete zu installieren, die für die Schnittstelle mit der OpenAI Gym RL Environments Library erforderlich sind, befolgen Sie ihre Anweisungen zum Installieren der zum Ausführen des RL -Umgebungssimulators benötigten Pakete (unter Linux / macOS).
Link zum Docker -Repository.
Wir bieten auch eine Dockerfile, in der BindsNet und alle seine Abhängigkeiten installiert sind. Problem
docker build .
Auf der obersten Ebene dieses Projekts zum Erstellen eines Docker -Images.
Um den Namen des neu erstellten Bildes zu ändern, Problem
docker tag <IMAGE_ID> <NEW_IMAGE_ID>
Um einen Container auszuführen und ein Bash -Terminal darin zu erhalten
docker run -it <NEW_IMAGE_ID> bash
Aus diesem Papier eine nahe Replikation des SNN aus diesem Papier zu führen
cd examples/mnist
python eth_mnist.py
Es gibt eine Reihe optionaler Befehlszeilenargumente, die übergeben werden können, einschließlich --plot (Zeigt nützliche Überwachungszahlen an), --n_neurons [int] (Anzahl der exzitatorischen, inhibitorischen Neuronen simuliert), --mode ['train' | 'test'] (setzt den Netzwerkbetrieb in die Schulungs- oder Testphase) und mehr. Führen Sie das Skript mit der Flag --help oder -h aus, um weitere Informationen zu erhalten.
Eine Reihe anderer Beispiele sind im examples verfügbar, die die Funktionalität von BindsNet vorstellen sollen. Schauen Sie sich an und lassen Sie uns wissen, was Sie denken!
Geben Sie Folgendes aus, um die Tests durchzuführen:
python -m pytest test/
Einige Tests scheitern, wenn das offene AI gym nicht auf Ihrem Computer installiert ist.
Die Simulation der biologisch plausiblen Spike -Neuronendynamik kann eine Herausforderung sein. Es wird typischerweise durch Lösen gewöhnlicher Differentialgleichungen (ODES) durchgeführt, die diese Dynamik beschreiben. Pytorch unterstützt die Lösung von Differentialgleichungen (z. B. im Gegensatz zu brian2 ) nicht ausdrücklich, aber wir können die Oden, die die Dynamik in Differenzgleichungen definieren, umwandeln und sie in regelmäßigen, kurzen Intervallen ( dt in der Größenordnung von 1 Millisekunden) als Annäherung lösen. Natürlich machen Pakete wie brian2 unter der Motorhaube das Gleiche. Dies in PyTorch ist aus einigen Gründen aufregend:
Wir können das leistungsstarke und flexible torch.Tensor -Objekt verwenden, eine Wrapper um die numpy.ndarray , die auf und von GPU -Geräten übertragen werden kann.
Wir können es vermeiden, das Rad neu zu erfinden, indem wir Funktionen aus der torch.nn.functional neu auszahlen. zB, Faltung oder Pooling -Funktionen.
Das Konzept, das der Neuronspike -Order und ihre relativen Timing -Informationen codieren, ist ein zentrales Thema in der Neurowissenschaften. Markram et al. (1997) schlugen vor, dass Synapsen zwischen Neuronen aufgrund dieses relativen Timings stärken oder abgebaut werden sollten, und zuvor schlug Donald Hebb die Theorie des hebbischen Lernens vor, oft einfach als "Neuronen, die zusammen abfeuern, zusammen verdrahten". Die Erweiterung der Hebbischen Theorie von Markram et al. Ist als Spike-Timing-abhängige Plastizität (STDP) bekannt.
Wir sind daran interessiert, SNNs auf ML- und RL -Probleme anzuwenden. Wir verwenden STDP, um die Gewichte von Synapsen zu ändern, die Paare oder Populationen von Neuronen in SNNs verbinden. Im Kontext von ML möchten wir eine Einstellung von Synapsengewichten lernen, die datenabhängige Spike-Aktivität in SNNs erzeugt. Diese Aktivität ermöglicht es uns, anschließend einige ML -Aufgaben von Interesse auszuführen. ZB, diskriminieren oder clustering Eingabedaten. Im Kontext von RL können wir uns das spikörische neuronale Netzwerk als RL -Agent vorstellen, dessen Spikeaktivität in Aktionen im Aktionsraum einer Umgebung umgewandelt werden kann.
Wir haben einige einfache Starter-Skripte für unbeaufsichtigtes Lernen (Lernen einer vollständig vernetzten oder fließenden Repräsentation über STDP), beaufsichtigtes Lernen (Einklemmen der Ausgabe-Neuronen auf das gewünschte Spikeverhalten abhängig von Datenbezeichnungen) und Verstärkungslernen (Konvertieren von Beobachtungen von den ATARI-Spielen-Invaders zu einer SNN-Konvertierung und Konvertieren von Netzwerkaktivitäten in Aktionen) bereitgestellt.
Wir simulierten ein Netzwerk mit einer Population von N-Poisson-Input-Neuronen mit Brennraten (in Hertz), die zufällig aus u (0, 100) gezogen wurden, die All-zu-All-zu-All-zu-All-Größenpopulation von Neuronen (LIVE-und-Fire) mit einer gleichgroßen Population von N (0,1) angeschlossen haben. Wir haben n systematisch von 250 bis 10.000 in Schritten von 250 variiert und jede Simulation mit jeder Bibliothek für 1.000 ms mit einer zeitlichen Auflösung DT = 1,0 ausgeführt. Wir haben BindsNet (mit CPU- und GPU-Berechnung), Brian2, Pynest (die Python-Schnittstelle zur Nest-SLI-Schnittstelle, die den C ++ Nest-Kernsimulator ausführt), Annarchy (mit CPU- und GPU-Berechnung) und BRIAN2GENN (BRIAN2-Front-End-Simulator).
Mehrere Pakete, einschließlich Brian und Pynest, ermöglichen die Einstellung bestimmter globaler Präferenzen. z. B. die Anzahl der CPU -Threads, die Anzahl der OpenMP -Prozesse usw. Wir haben diese Einstellungen für unsere Benchmark -Studie ausgewählt, um die Geschwindigkeit jeder Bibliothek zu maximieren. Beachten Sie jedoch, dass BindsNet keine Einstellung solcher Optionen erfordert. Unser Ansatz, der das Rechenmodell von Pytorch erbt, scheint die verfügbare Hardware am besten zu nutzen, und macht es den Praktikern daher einfach, die beste Leistung ihres Systems mit dem geringsten Aufwand zu erzielen.
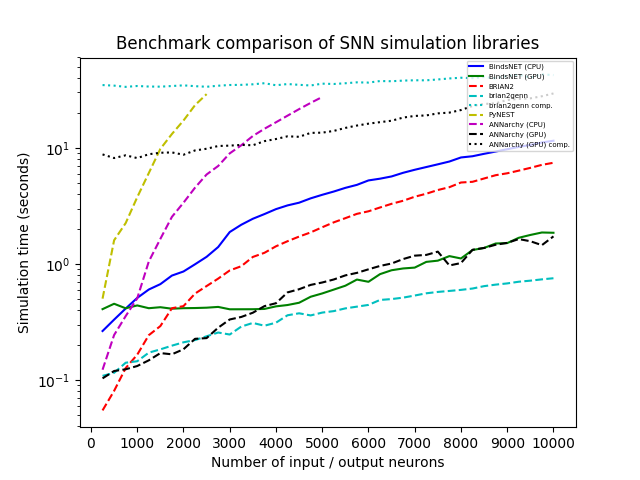
Alle Simulationen werden auf Ubuntu 16.04 LTs mit Intel (R) Xeon (R) CPU E5-2687W V3 @ 3.10 GHz, 128 GB RAM @ 2133MHz und zwei Geforce GTX Titan X (GM200) GPUs ausgeführt. Python 3.6 wird in allen Fällen verwendet. Die Uhrzeit wurde für jeden Simulationslauf aufgezeichnet.
Wenn Sie BindsNet in Ihrer Forschung verwenden, geben Sie bitte den folgenden Artikel an:
@ARTICLE{10.3389/fninf.2018.00089,
AUTHOR={Hazan, Hananel and Saunders, Daniel J. and Khan, Hassaan and Patel, Devdhar and Sanghavi, Darpan T. and Siegelmann, Hava T. and Kozma, Robert},
TITLE={BindsNET: A Machine Learning-Oriented Spiking Neural Networks Library in Python},
JOURNAL={Frontiers in Neuroinformatics},
VOLUME={12},
PAGES={89},
YEAR={2018},
URL={https://www.frontiersin.org/article/10.3389/fninf.2018.00089},
DOI={10.3389/fninf.2018.00089},
ISSN={1662-5196},
}
Mit Beitragsröcken gemacht.
GNU Affero Allgemeine öffentliche Lizenz v3.0