ResNeSt
1.0.0
Split-Attention Network、新しいResnetバリアント。 Mask R-CNN、Cascade R-CNN、DeepLabv3などのダウンストリームモデルのパフォーマンスを大幅に向上させます。
# using github url
pip install git+https://github.com/zhanghang1989/ResNeSt
# using pypi
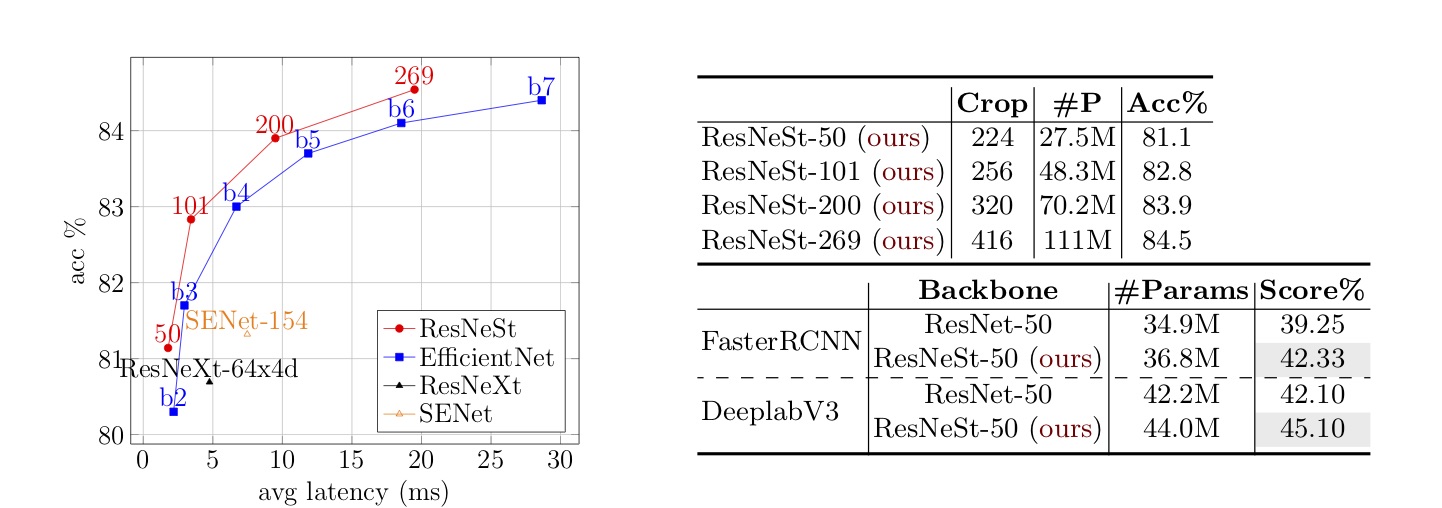
pip install resnest --pre| 作物サイズ | Pytorch | グルオン | |
|---|---|---|---|
| resnest-50 | 224 | 81.03 | 81.04 |
| resnest-101 | 256 | 82.83 | 82.81 |
| Resnest-200 | 320 | 83.84 | 83.88 |
| Resnest-269 | 416 | 84.54 | 84.53 |
Tensorflow、Caffe、Jax:Tensorflow、Jaxの第三者の実装が利用可能です。
追加のアブレーション研究モデルはリンクで利用できます
import torch
# get list of models
torch . hub . list ( 'zhanghang1989/ResNeSt' , force_reload = True )
# load pretrained models, using ResNeSt-50 as an example
net = torch . hub . load ( 'zhanghang1989/ResNeSt' , 'resnest50' , pretrained = True ) # using ResNeSt-50 as an example
from resnest . torch import resnest50
net = resnest50 ( pretrained = True ) # using ResNeSt-50 as an example
from resnest . gluon import resnest50
net = resnest50 ( pretrained = True )D2で逆バックボーンを備えたTraining detectron2モデルのラッパーを提供します。トレーニング構成と前提条件のモデルがリリースされます。 D2の詳細を参照してください。
最新の骨格は、MMDeTectionによって採用されています。
注:論文で報告されている推論速度は、Recordioデータを使用したGluon実装を使用してテストされています。
ここでは、Raw Image Data Formatを使用してSimplicityを使用して、Recordio形式を使用する場合はGluonCVチュートリアルをフォローしてください。
cd scripts/dataset/
# assuming you have downloaded the dataset in the current folder
python prepare_imagenet.py --download-dir ./ # use resnest50 as an example
cd scripts/torch/
python verify.py --model resnest50 --crop-size 224 # use resnest50 as an example
cd scripts/gluon/
python verify.py --model resnest50 --crop-size 224オブジェクトの検出およびインスタンスセグメンテーションモデルについては、detectron2-resnestフォークにアクセスしてください。
resnest:分割出席ネットワーク[arxiv]
ハングチャン、チョンルオウ、Zhongyue Zhang、Yi Zhu、Zhi Zhang、Haibin Lin、Yue Sun、Tong He、Jonas Muller、R。Manmatha、Mu Li、Alex Smola
@article{zhang2020resnest,
title={ResNeSt: Split-Attention Networks},
author={Zhang, Hang and Wu, Chongruo and Zhang, Zhongyue and Zhu, Yi and Zhang, Zhi and Lin, Haibin and Sun, Yue and He, Tong and Muller, Jonas and Manmatha, R. and Li, Mu and Smola, Alexander},
journal={arXiv preprint arXiv:2004.08955},
year={2020}
}