
multiarm offre des fonctions pour aider à la conception d'essais cliniques multi-armes multiples à un et multi-étages. Dans les deux cas, les fonctions disponibles permettent la détermination de la taille de l'échantillon, la simulation d'essai, le calcul des caractéristiques de fonctionnement analytique et la production de plusieurs parcelles informatives.
Notez qu'une interface utilisateur graphique R Shiny est également disponible pour la conception d'essais à un stade; Il est accessible dans R à l'aide de multiarm::gui() , ou en ligne sur https://mjgrayling.shinyapps.io/multiarm. Des informations supplémentaires sur cette application peuvent être trouvées dans Grayling et Wason (2020).
Vous pouvez installer la dernière version de développement de multiarm à partir de GitHub avec:
devtools :: install_github( " mjg211/multiarm " ) Un exemple d'introduction de la façon d'utiliser certaines des fonctionnalités de base du package peut être trouvée ci-dessous. Une prise en charge plus détaillée, pour les conceptions à un étage, est disponible dans le package Vignette, qui peut être accessible avec vignette("single_stage", package = "multiarm") . Voir aussi Grayling et Wason (2020). Pour plus d'aide, veuillez envoyer un courriel à [email protected].
Au total, 46 fonctions sont actuellement disponibles. Leurs conventions de dénomination sont telles que plusieurs chaînes de caractère sont réunies, séparées par des soulignements. La première chaîne indique le but de la fonction (c'est-à-dire quel type de calcul il effectue):
build_###_###() : Créer des objets de conception d'essai cliniques multi-bras, comme ceux renvoyés par les fonctions des_###_###() . Pour une utilisation lorsqu'un design spécifique est intéressant.des_###_###() : Déterminez la taille de l'échantillon requise par un type particulier de conception d'essai cliniques multi-bras.gui() : fournit une interface utilisateur graphique à la détermination de la conception.opchar_###_###() : Déterminez les caractéristiques de fonctionnement (puissance, taux d'erreur familiale, etc.) d'une conception d'essai cliniques multi-bras fournie, via l'intégration normale multivariée.plot.multiarm_des_###_###() : Produisez des parcelles informatives (puissance, courbes de taux de fausses découvertes, etc.) concernant une conception d'essai cliniques multi-bras fournie.sim_###_###() : Estimez empiriquement les caractéristiques de fonctionnement (puissance, taux d'erreur familiale, etc.) d'une conception d'essai cliniques multi-bras fournie, via la simulation.La seconde indique la conception:
###_dtl_###() : se rapporter aux conceptions de drop-the-losers en plusieurs étapes. Voir, par exemple, Wason et al (2017).###_gs_###() : se rapportent aux conceptions multi-stage multi-bras sequentielles de groupe. Voir, par exemple, Magirr et al (2012).###_ss_###() : se rapportent aux conceptions à un étage. Voir, par exemple, Grayling et Wason (2020).Le troisième indique à quel type de résultat la fonction est:
###_###_bern() : Supposons un résultat primaire distribué à Bernoulli.###_###_norm() : Supposons un résultat primaire normalement distribué.###_###_pois() : Supposons un résultat primaire distribué à Poisson. En règle générale, une fonction des_###_###() serait d'abord utilisée pour identifier une conception pour les paramètres d'essai d'intérêt. Par exemple, envisagez de concevoir un essai en un étage pour:
K ).alpha et correction ).beta , delta1 et power ).sigma ).ratio ).Pour calculer la conception, nous courions:
des <- des_ss_norm( K = 3 ,
alpha = 0.05 ,
beta = 0.2 ,
delta1 = 1 ,
sigma = rep( 1 , 4 ),
ratio = rep( 1 , 3 ),
correction = " dunnett " ,
power = " marginal " )Ensuite, la taille totale de l'échantillon requise est:
des $ N
# > [1] 67.42534De plus, les caractéristiques de fonctionnement sous la Global Null , Global Alternative et chacune des configurations les moins favorables sont accessibles à:
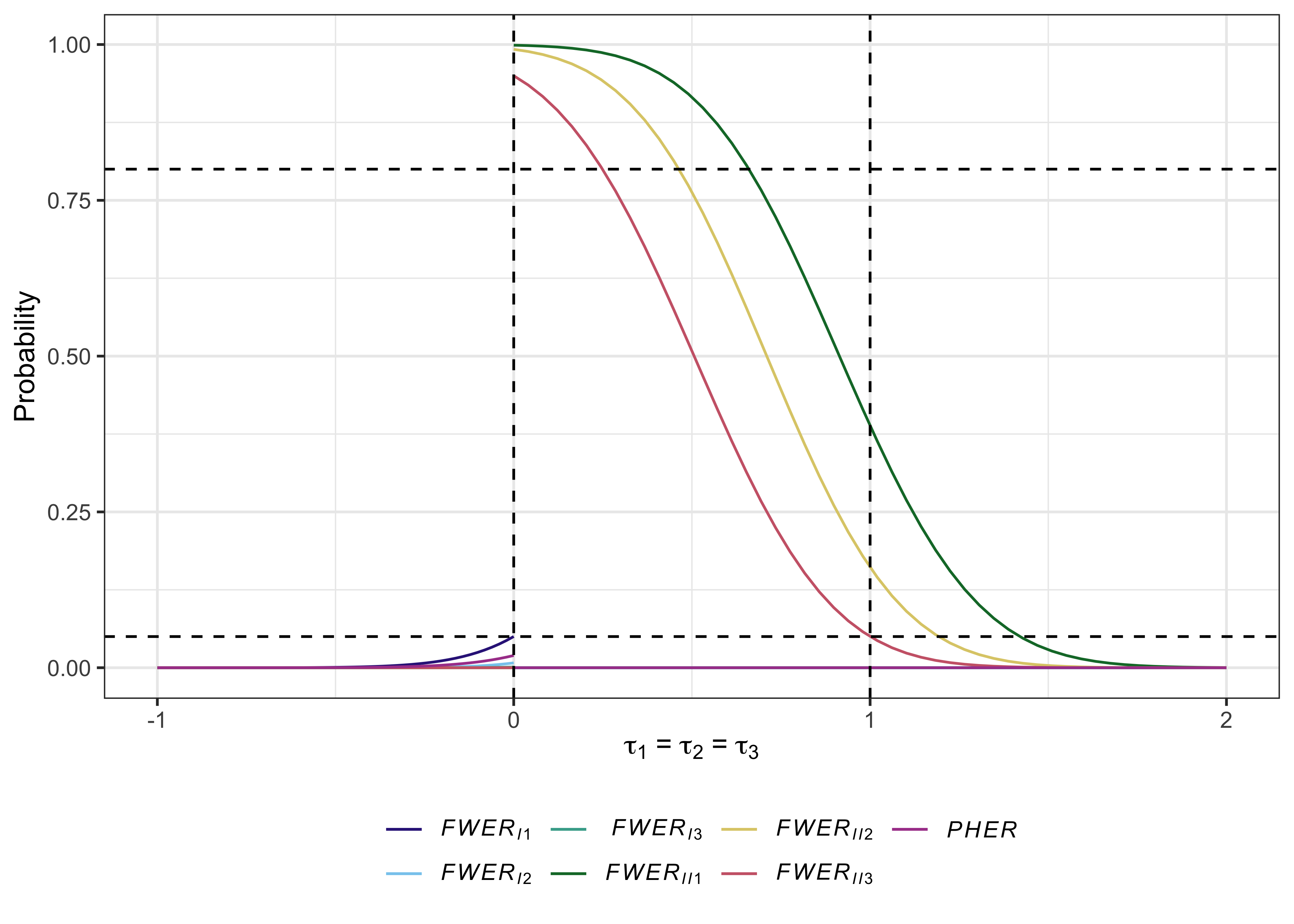
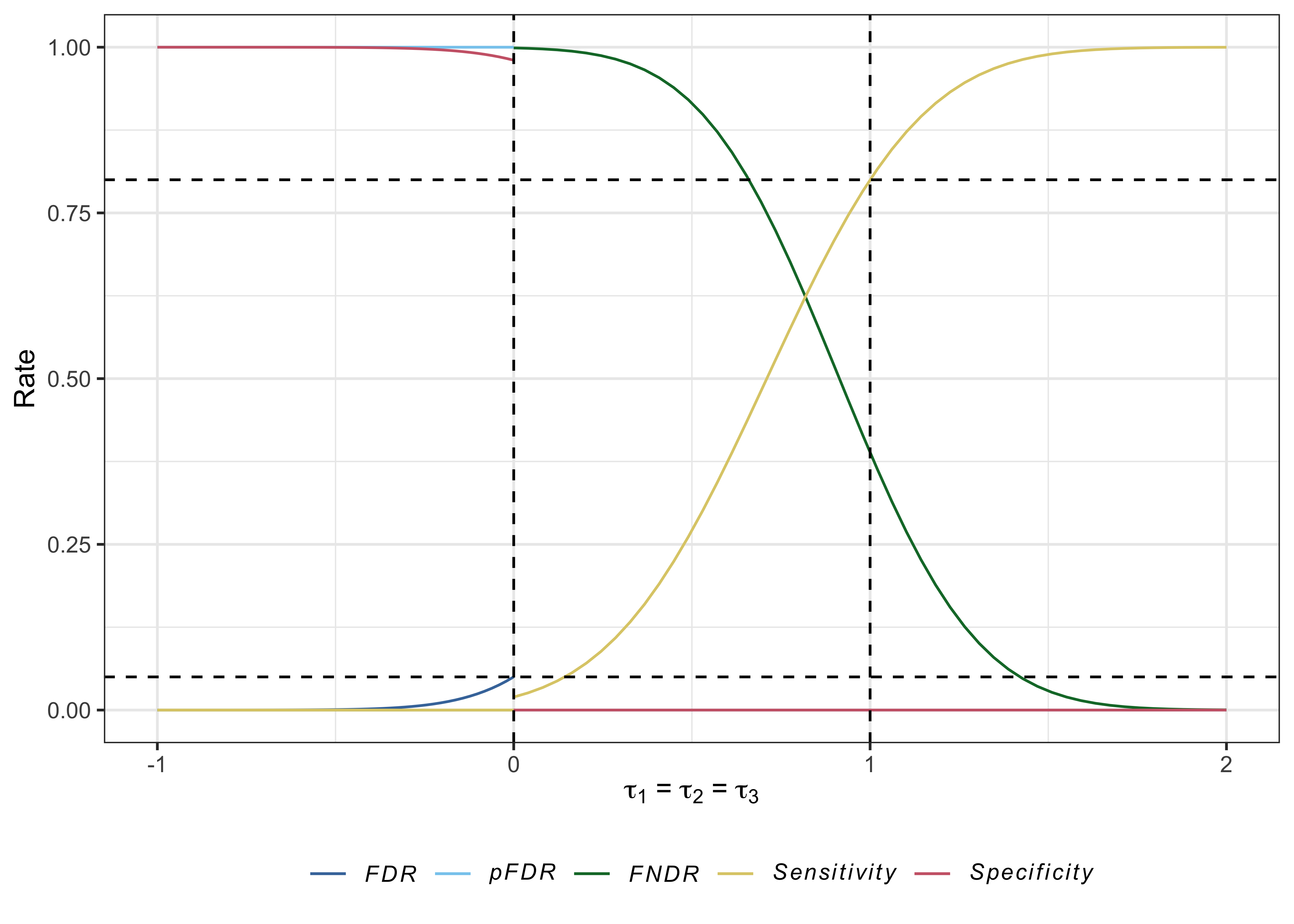
des $ opchar
# > # A tibble: 5 x 20
# > tau1 tau2 tau3 Pdis Pcon P1 P2 P3 FWERI1 FWERI2 FWERI3
# > <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
# > 1 0 0 0 0.0501 0.00109 0.0196 0.0196 0.0196 0.0501 0.00774 0.00109
# > 2 1 1 1 0.950 0.611 0.800 0.800 0.800 0 0 0
# > 3 1 0 0 0.800 0.00330 0.800 0.0196 0.0196 0.0360 0.00330 0
# > 4 0 1 0 0.800 0.00330 0.0196 0.800 0.0196 0.0360 0.00330 0
# > 5 0 0 1 0.800 0.00329 0.0196 0.0196 0.800 0.0360 0.00330 0
# > # … with 9 more variables: FWERII1 <dbl>, FWERII2 <dbl>, FWERII3 <dbl>,
# > # PHER <dbl>, FDR <dbl>, pFDR <dbl>, FNDR <dbl>, Sens <dbl>, Spec <dbl> Des tracés utiles peuvent également être produits avec plot.multiarm_des_ss_norm() comme suit:
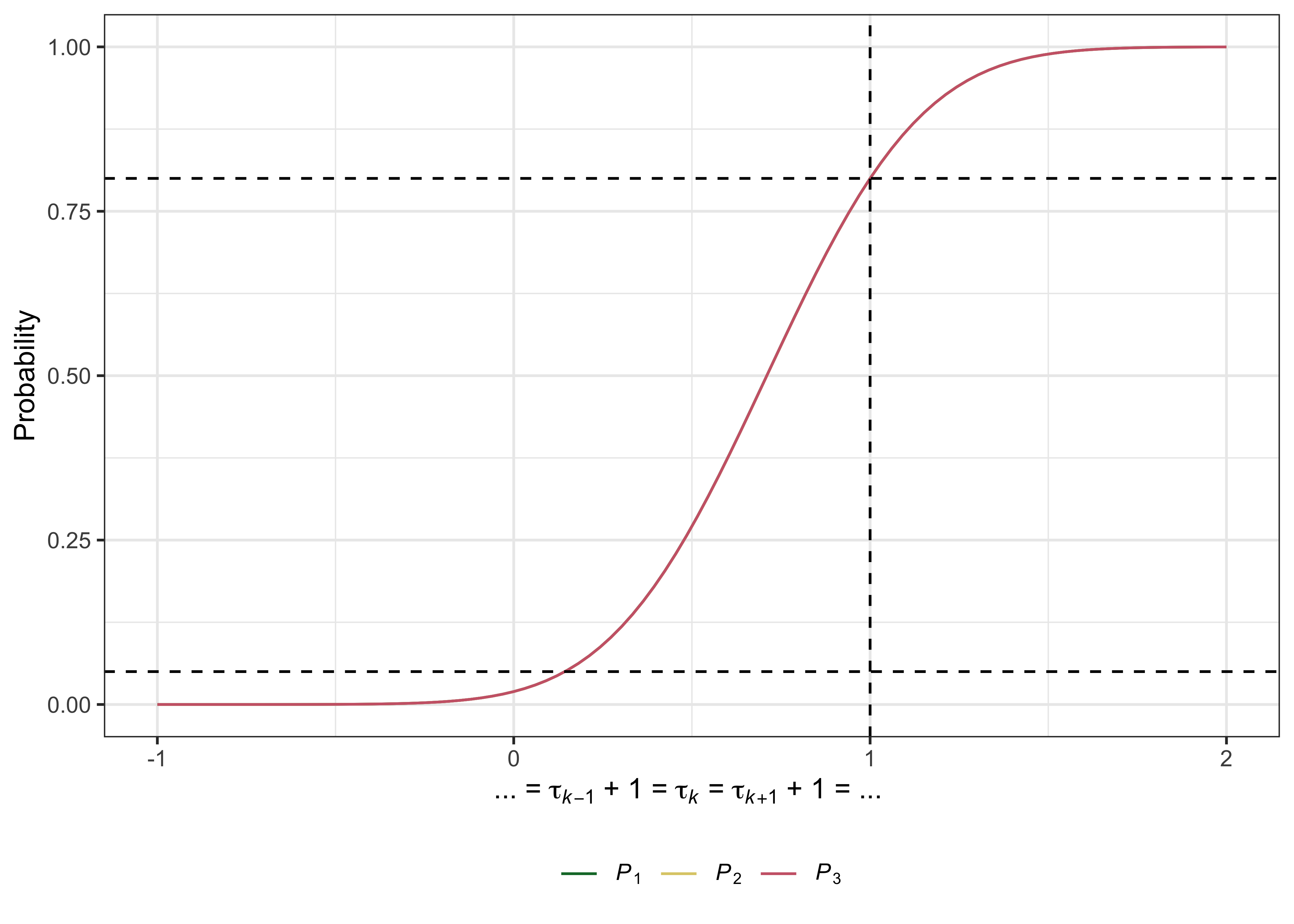
plot( des )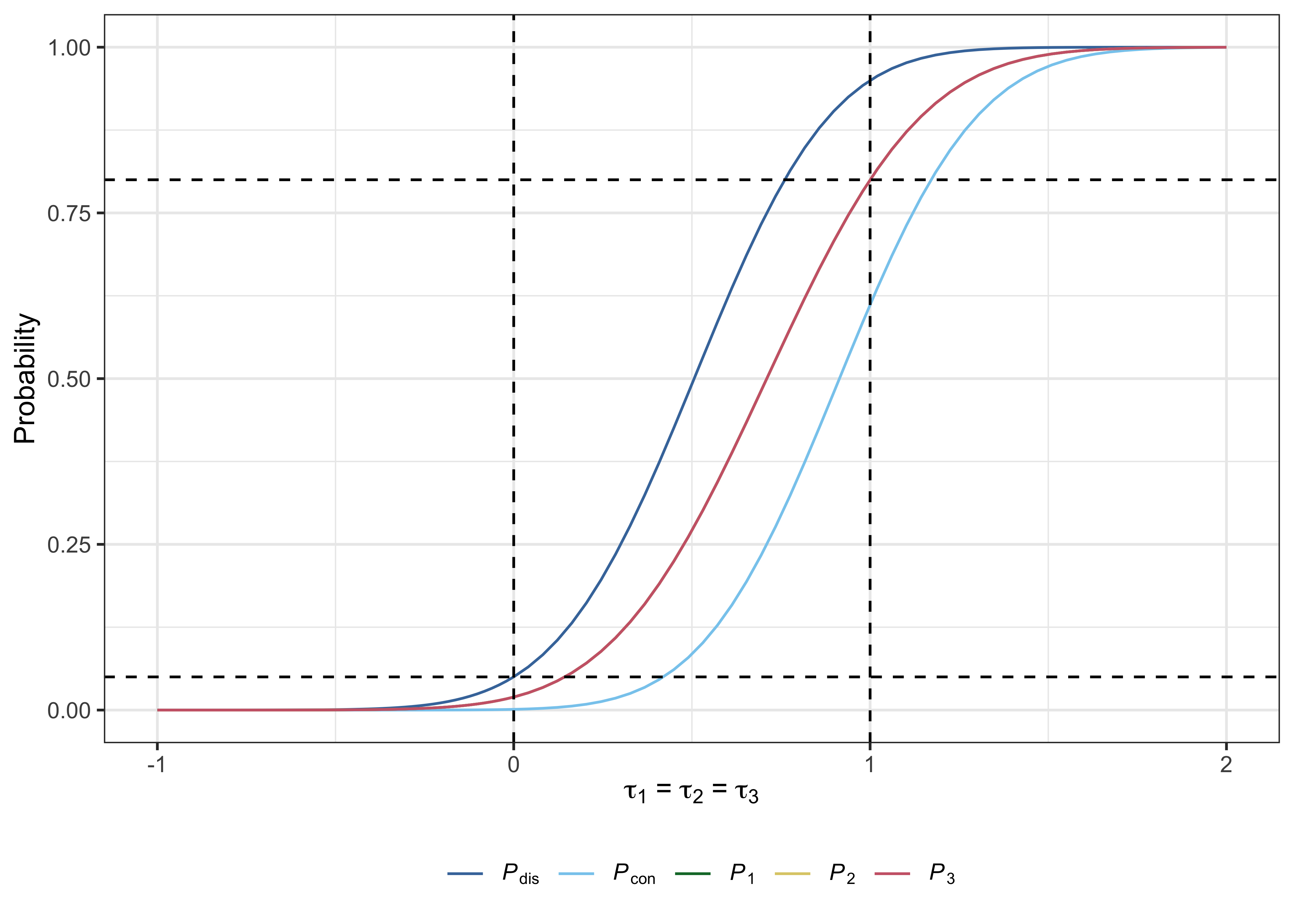
Grayling MJ, Wason JMS (2020) Une application Web pour la conception d'essais cliniques multi-bras. BMC Cancer 20:80. Doi: 10.1186 / s12885-020-6525-0. PMID: 32005187.
Magirr D, Jaki T, Whitehead J (2012) Un test de Dunnett généralisé pour les études cliniques multi-armes à plusieurs bras avec sélection de traitement. Biometrika 99 (2): 494–501. Doi: 10.1093 / biomet / ass002.
Wason J, Stallard N, Bowden J, Jennison C (2017) Une conception de drop-the-losers en plusieurs étapes pour les essais cliniques multi-bras. STAT Meth Med Res 26 (1): 508–524. Doi: 10.1177 / 0962280214550759. PMID: 25228636.