
multiarm proporciona funciones para ayudar con el diseño de ensayos clínicos multi-arm de una etapa múltiple. En cualquier caso, las funciones disponibles permiten la determinación del tamaño de la muestra, la simulación de prueba, el cálculo de las características operativas analíticas y la producción de varias parcelas informativas.
Tenga en cuenta que una interfaz gráfica de usuario brillante también está disponible para diseñar pruebas de una sola etapa; Se puede acceder dentro de R usando multiarm::gui() , o en línea en https://mjgrayling.shinyapps.io/multiarm. Se puede encontrar información adicional sobre esta aplicación en Grayling y Wason (2020).
Puede instalar la última versión de desarrollo de multiarm de GitHub con:
devtools :: install_github( " mjg211/multiarm " ) A continuación se puede encontrar un ejemplo introductorio de cómo utilizar la funcionalidad central del paquete. El soporte más detallado, para los diseños de una sola etapa, está disponible en la viñeta de paquetes, a la que se puede acceder con vignette("single_stage", package = "multiarm") . Ver también Grayling y Wason (2020). Para obtener más ayuda, envíe un correo electrónico a [email protected].
En total, 46 funciones están actualmente disponibles. Sus convenciones de nombres son tales que se unen varias cadenas de personajes, separadas por subrayos. La primera cadena indica el propósito de la función (es decir, qué tipo de cálculo realiza):
build_###_###() : Construya objetos de diseño de ensayos clínicos múltiples, como los devueltos por las funciones des_###_###() . Para su uso cuando un diseño específico es de interés.des_###_###() : Determine el tamaño de la muestra requerido por un tipo particular de diseño de ensayos clínicos múltiples.gui() : proporciona una interfaz gráfica de usuario para la determinación del diseño.opchar_###_###() : Determine las características operativas (potencia, tasas de error de forma familiar, etc.) de un diseño de ensayo clínico múltiple suministrado, a través de una integración normal multivariada.plot.multiarm_des_###_###() : Producir gráficos informativos (potencia, curvas de tasa de descubrimiento falso, etc.) relacionados con un diseño de ensayo clínico múltiple suministrado.sim_###_###() : Estime empíricamente las características operativas (potencia, tasas de error en forma de familia, etc.) de un diseño de ensayo clínico múltiple suministrado, a través de la simulación.El segundo indica el diseño:
###_dtl_###() : Relacionar con los diseños de Drop-the-Losers de múltiples etapas. Véase, por ejemplo, Wason et al (2017).###_gs_###() : Relacionarse con diseños de múltiples etapas múltiples secuenciales de grupo. Véase, por ejemplo, Magirr et al (2012).###_ss_###() : Relacionarse con diseños de una sola etapa. Ver, por ejemplo, Grayling y Wason (2020).El tercero indica para qué tipo de resultado es la función:
###_###_bern() : Suponga un resultado primario distribuido Bernoulli.###_###_norm() : Suponga un resultado primario normalmente distribuido.###_###_pois() : Suponga un resultado primario distribuido por Poisson. Por lo general, una función des_###_###() se utilizaría primero para identificar un diseño para los parámetros de interés de interés. Por ejemplo, considere diseñar una prueba de una sola etapa para:
K ).alpha y correction ).beta , delta1 y power ).sigma ).ratio ).Para calcular el diseño, ejecutaríamos:
des <- des_ss_norm( K = 3 ,
alpha = 0.05 ,
beta = 0.2 ,
delta1 = 1 ,
sigma = rep( 1 , 4 ),
ratio = rep( 1 , 3 ),
correction = " dunnett " ,
power = " marginal " )Luego, el tamaño total de la muestra requerido es:
des $ N
# > [1] 67.42534Además, se puede acceder a las características operativas bajo la alternativa global nulo , global y cada una de las configuraciones menos favorables con:
des $ opchar
# > # A tibble: 5 x 20
# > tau1 tau2 tau3 Pdis Pcon P1 P2 P3 FWERI1 FWERI2 FWERI3
# > <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
# > 1 0 0 0 0.0501 0.00109 0.0196 0.0196 0.0196 0.0501 0.00774 0.00109
# > 2 1 1 1 0.950 0.611 0.800 0.800 0.800 0 0 0
# > 3 1 0 0 0.800 0.00330 0.800 0.0196 0.0196 0.0360 0.00330 0
# > 4 0 1 0 0.800 0.00330 0.0196 0.800 0.0196 0.0360 0.00330 0
# > 5 0 0 1 0.800 0.00329 0.0196 0.0196 0.800 0.0360 0.00330 0
# > # … with 9 more variables: FWERII1 <dbl>, FWERII2 <dbl>, FWERII3 <dbl>,
# > # PHER <dbl>, FDR <dbl>, pFDR <dbl>, FNDR <dbl>, Sens <dbl>, Spec <dbl> Las gráficas útiles también se pueden producir con plot.multiarm_des_ss_norm() como sigue:
plot( des )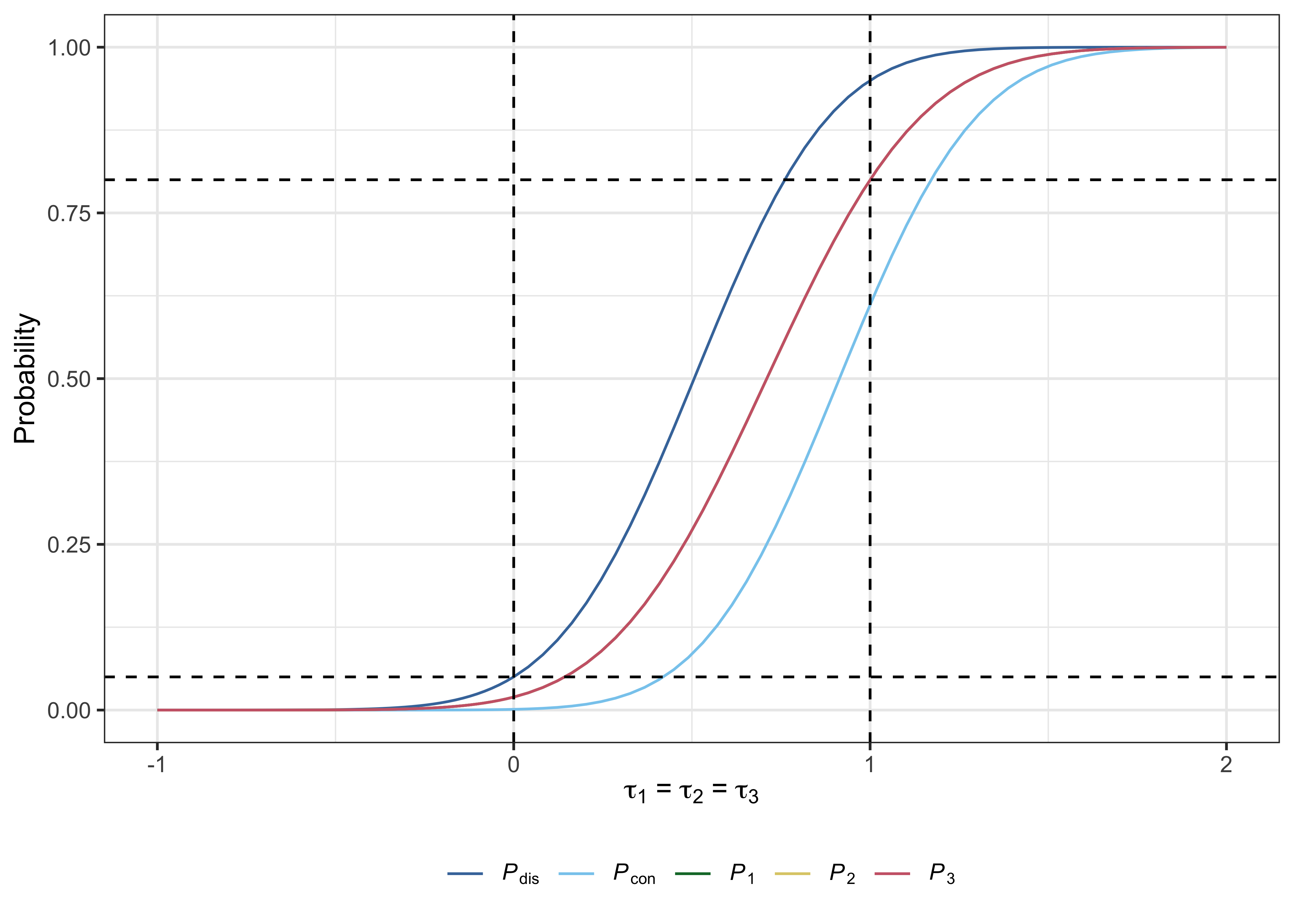
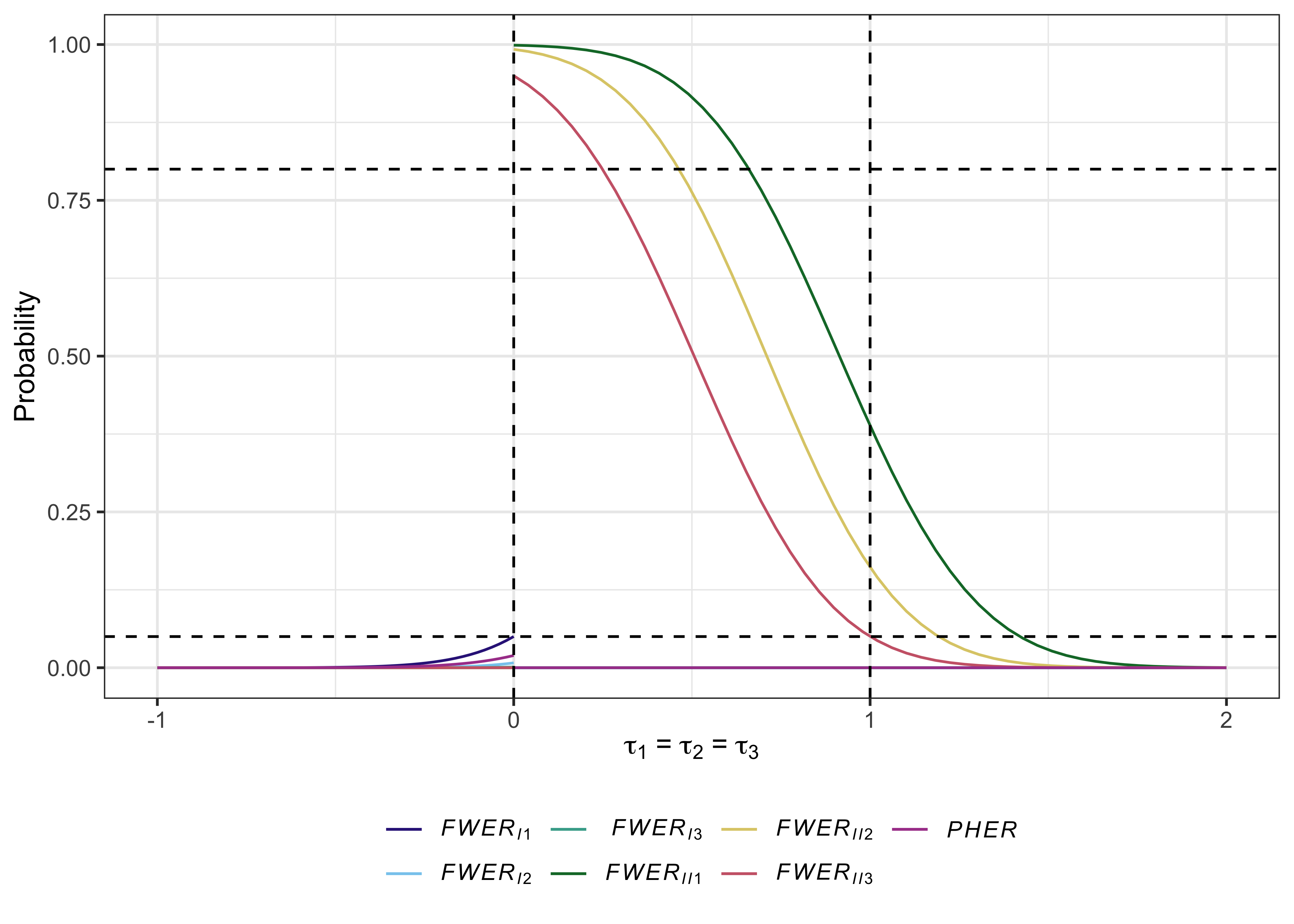
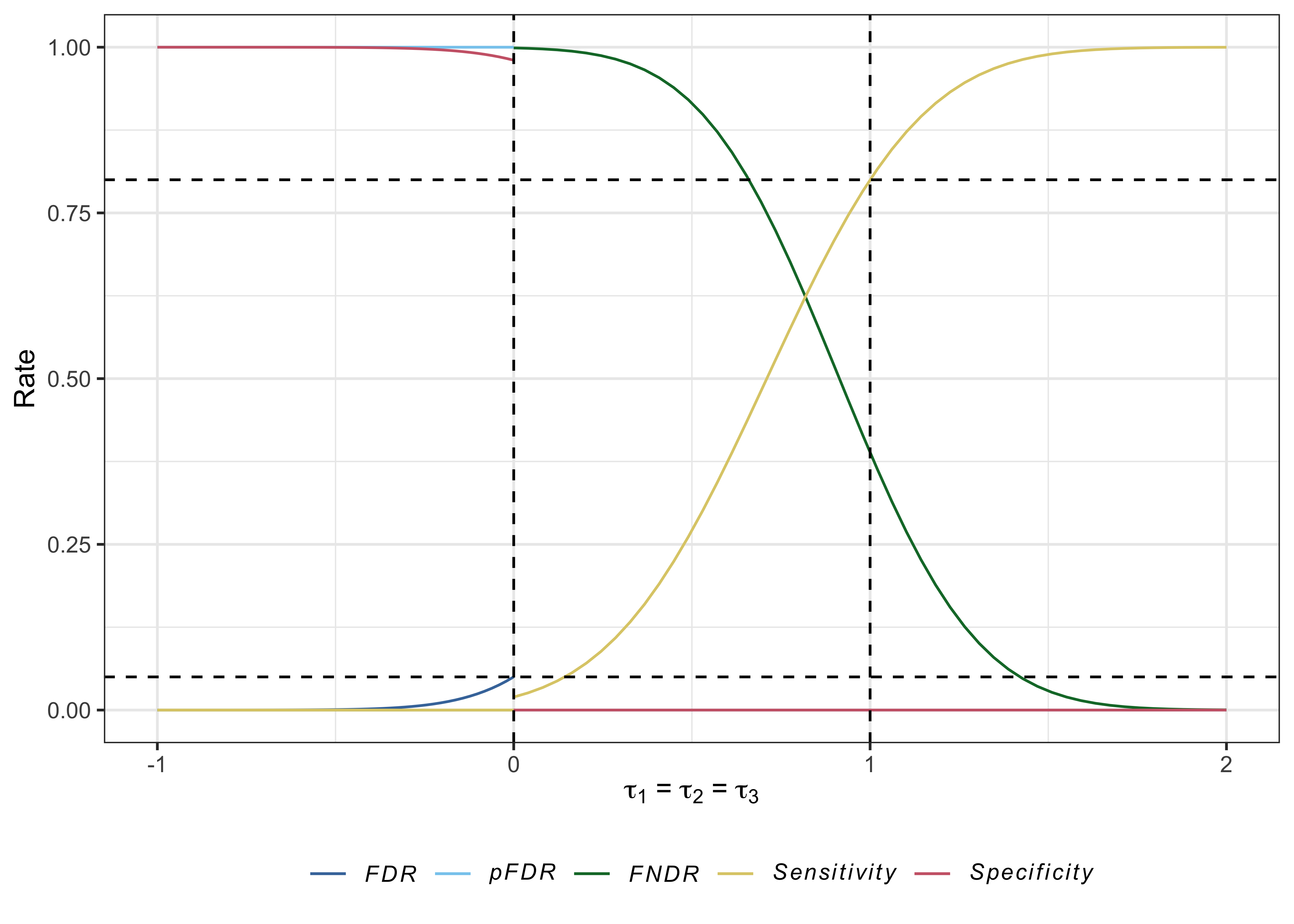
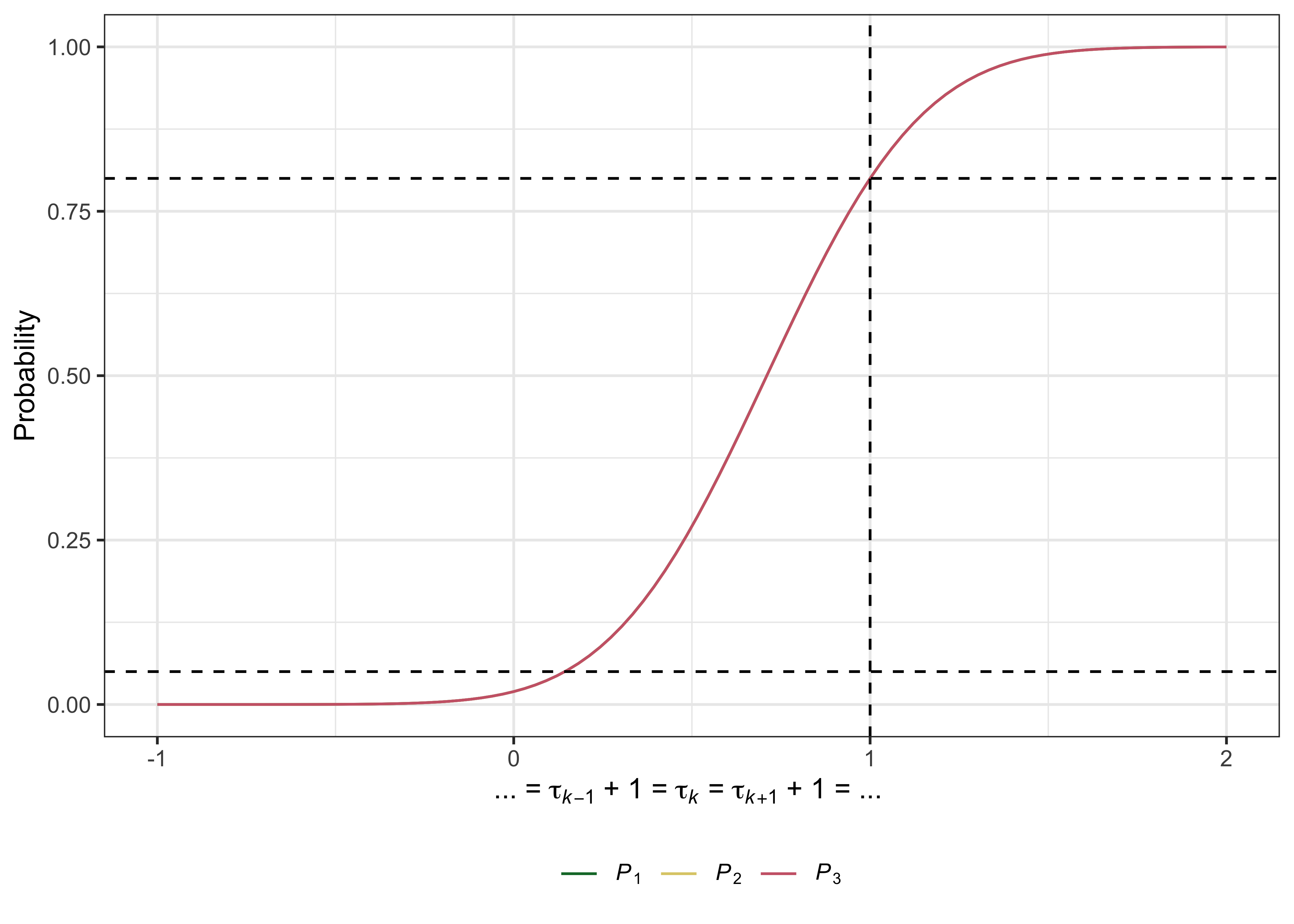
Grayling MJ, Wason JMS (2020) Una aplicación web para el diseño de ensayos clínicos múltiples. BMC Cancer 20:80. Doi: 10.1186/s12885-020-6525-0. PMID: 32005187.
Magirr D, Jaki T, Whitehead J (2012) Una prueba de Dunnett generalizada para estudios clínicos de múltiples etapas múltiples con selección de tratamiento. Biometrika 99 (2): 494–501. Doi: 10.1093/biomet/ass002.
Wason J, Stallard N, Bowden J, Jennison C (2017) Un diseño de Drop-the Losers de varias etapas para ensayos clínicos de múltiples brazos. Stat Meth Med Res 26 (1): 508–524. Doi: 10.1177/0962280214550759. PMID: 25228636.