Este repositorio contiene nuestro código para el documento "Prácticas: Aprovechar modelos de lenguaje grandes para la vinculación de conceptos biomédicos de código cruzado".
En este artículo, abordamos la tarea de vinculación de concepto biomédico, que tiene como objetivo vincular conceptos biomédicos entre fuentes/sistemas en función de sus significados semánticos y conocimiento biomédico. Se basa únicamente en los nombres de conceptos y, por lo tanto, puede cubrir una gama mucho más amplia de aplicaciones del mundo real. Esta tarea difiere de las tareas existentes, como la vinculación de la entidad, la alineación de la entidad y la coincidencia de ontología, que dependen de la información contextual o topológica adicional. En la siguiente figura se describe un ejemplo de juguete de la tarea de vinculación del concepto biomédico.
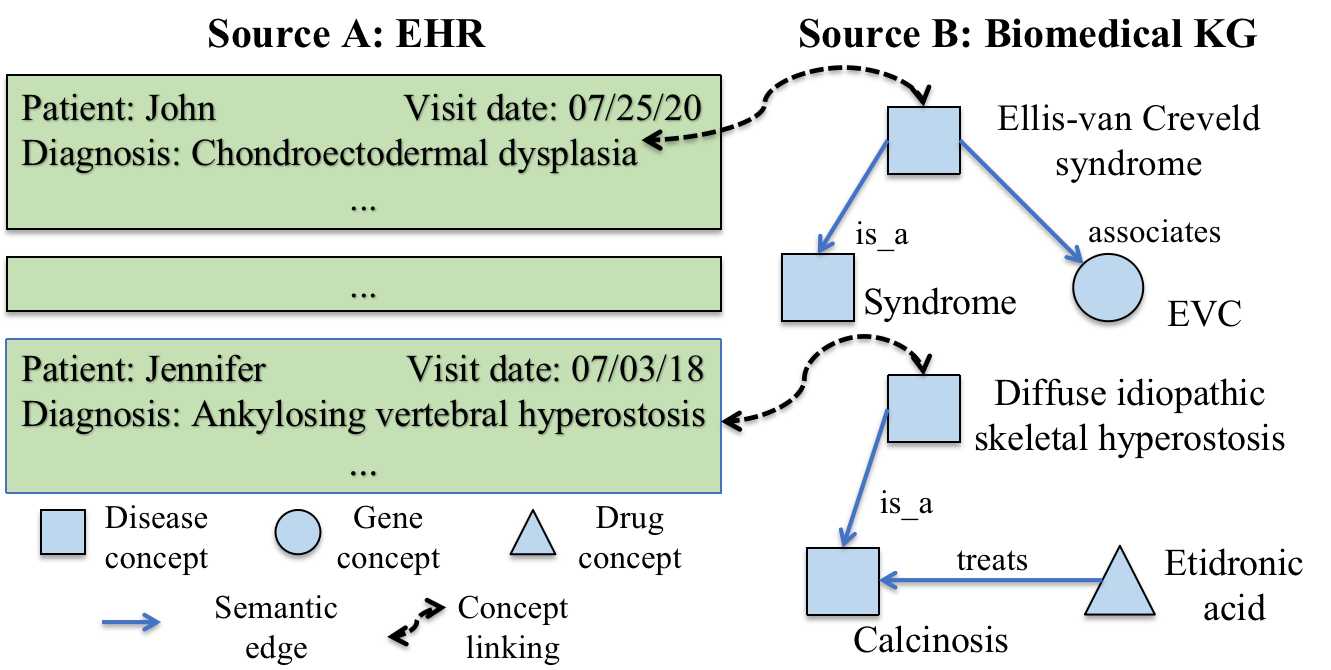
Figura 1: Un ejemplo de juguete. Izquierda: Conceptos en el EHR. Derecha: Conceptos en el KG biomédico.
PractLink es un nuevo marco de vinculación de concepto biomédico que aprovecha los modelos de lenguaje grandes (LLM). Primero emplea un modelo de lenguaje previamente capacitado especializado en biomedicina para generar conceptos candidatos que se ajustan a las ventanas de contexto LLM. Luego, utiliza un LLM para vincular conceptos a través de indicaciones de dos etapas. El indicador de la primera etapa tiene como objetivo provocar el conocimiento previo biomédico de la LLM para la tarea de vinculación del concepto, mientras que el indicador de la segunda etapa obliga a la LLM a reflexionar sobre sus propias predicciones para mejorar aún más su confiabilidad. La descripción general del marco de Lidlink se ilustra en la siguiente figura.
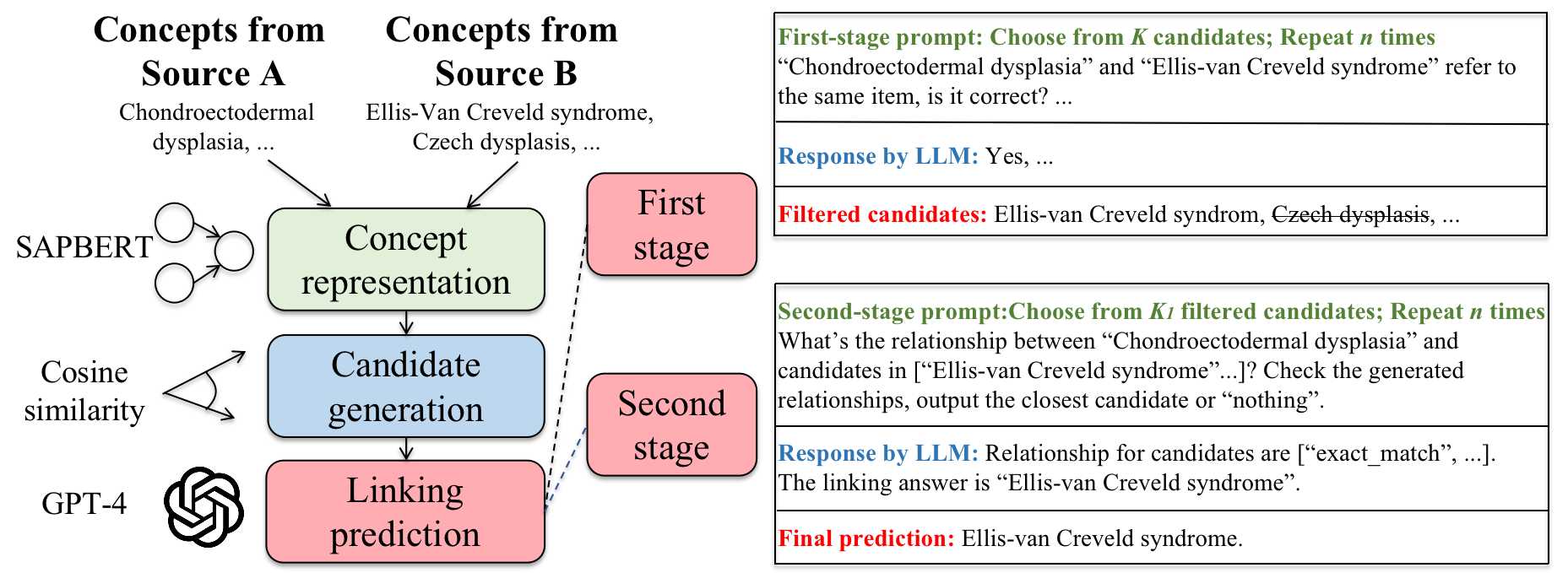
Figura 2: Descripción general de nuestro marco PRoClink propuesto.
[El archivo "requisitos.txt" podría usarse para descargar los paquetes de Python automáticamente]
Python == 3.8.10
editDistance == 0.6.2
Fuego == 0.5.0
numpy == 1.19.5
OPERAI == 0.28.1
pandas == 1.3.4
rank_bm25 == 0.2.2
SciPy == 1.12.0
Simstring-Fast == 0.3.0
TextDistance == 4.6.1
Antorcha == 1.10.0+CU111
TQDM == 4.66.1
Transformers == 4.33.3
Curamos dos conjuntos de datos de referencia de concepto biomédico que vincula: MIID (MIMIC-III-IBKH-Disease) y Cise (CRADLE-IBKH-Side-Effect), utilizando datos del conjunto de datos MIMIC-III EHR MIMIC MIMIC LINK, CRADLE EHR DataSet (un DataSet privado COTASET de un gran sistema de salud en el Estados Unidos), iBkh DataSet (IBKH Kgkh (un conjunto de datos privado EHR recopilado de un gran sistema de salud en el estado de la salud de la gran salud), iBkh DataSet (DataSet privado). enlace IBKH y enlace UMLS System UMLS. Debido a la naturaleza confidencial de los datos médicos y las consideraciones de privacidad, existen restricciones al intercambio de datos. Para obtener acceso a estos conjuntos de datos médicos, se requieren capacitación y credenciales apropiadas. Para obtener más ayuda con el acceso a los datos u otras consultas relacionadas, no dude en comunicarse con nuestro equipo de autor.
La mayor parte del código se almacena en tres carpetas: "gen_candidates", "gen_gpt_responses" y "líneas de base". Se pueden encontrar más detalles dentro de estas carpetas respectivamente.
Carpeta "Gen_candidates": esta carpeta contiene el código para la representación conceptual de PromptLink y el proceso de generación de candidatos.
Carpeta "gen_gpt_responses": esta carpeta muestra cómo la lista de apuración aprovecha el LLM para recuperar la respuesta de predicción final.
Carpeta "líneas de base": esta carpeta contiene el código para ejecutar todos los métodos de referencia comparados, incluidos BM25, Levenshtein Distance, BioBert y Sapbert.