Dieses Repo enthält unseren Code für Papier "promptLink: Nutzung großer Sprachmodelle für das biomedizinische Cross-Source-Konzept" Verknüpfung ".
In diesem Artikel befassen wir uns mit der biomedizinischen Konzeptverknüpfungsaufgabe, die darauf abzielt, biomedizinische Konzepte über Quellen/Systeme hinweg auf der Grundlage ihrer semantischen Bedeutungen und ihres biomedizinischen Wissens zu verknüpfen. Es stützt sich ausschließlich auf Konzeptnamen und kann somit eine viel breitere Palette realer Anwendungen abdecken. Diese Aufgabe unterscheidet sich von vorhandenen Aufgaben wie Entitätsverbinden, Entitätsausrichtung und Ontologie -Übereinstimmung, die von zusätzlichen kontextuellen oder topologischen Informationen abhängen. In der folgenden Abbildung wird ein Spielzeugbeispiel für das biomedizinische Konzeptverknüpfungsaufgabe beschrieben.
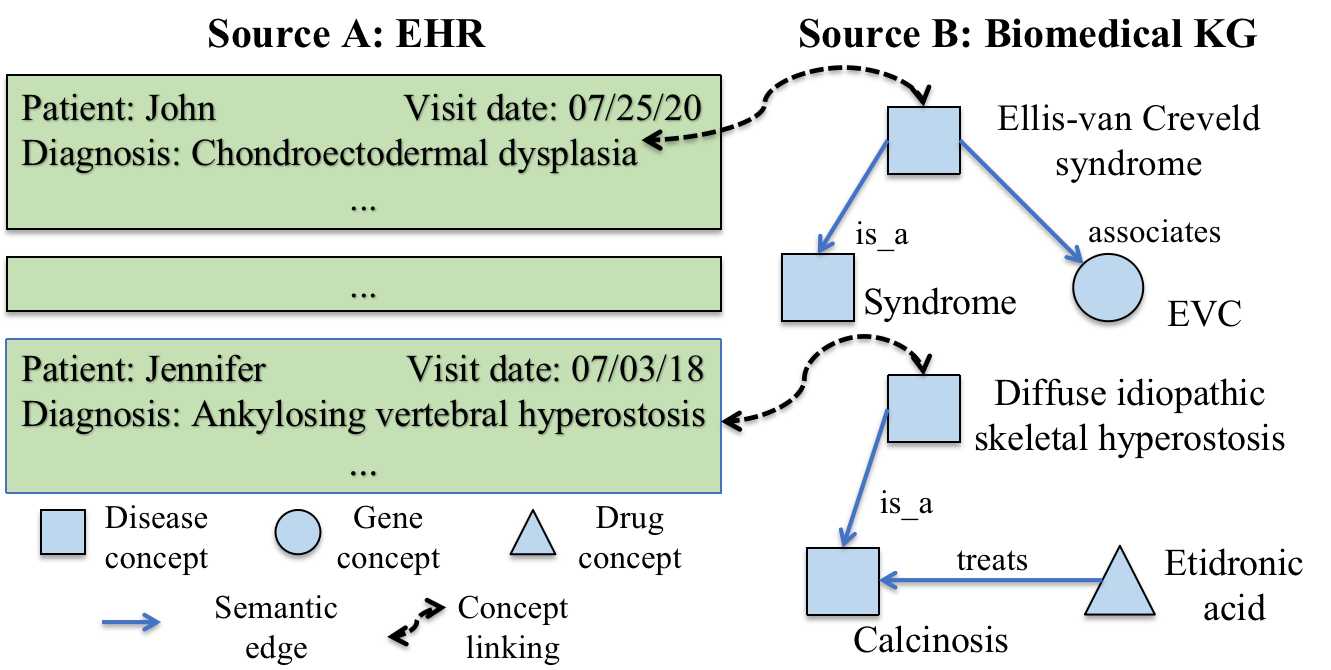
Abbildung 1: Ein Spielzeugbeispiel. Links: Konzepte im EHR. Rechts: Konzepte im biomedizinischen kg.
PromptLink ist ein neuartiges biomedizinisches Konzept, das Rahmen für die Verknüpfung von Großsprachen (LLMs) nutzt. Es wird zunächst ein vorgebildetes Sprachmodell verwendet, das auf Biomedizin spezialisiert ist, um Kandidatenkonzepte zu generieren, die in die LLM-Kontextfenster passen. Anschließend wird ein LLM verwendet, um Konzepte durch zweistufige Eingabeaufforderungen zu verknüpfen. Die Eingabeaufforderung in der ersten Stufe zielt darauf ab, aus dem LLM biomedizinischen Vorkenntnissen für die Konzeptverknüpfungsaufgabe auszulösen, während die Eingabeaufforderung in der zweiten Stufe die LLM dazu zwingt, über seine eigenen Vorhersagen nachzudenken, um ihre Zuverlässigkeit weiter zu verbessern. Der Überblick über das promptLink -Framework ist in der folgenden Abbildung dargestellt.
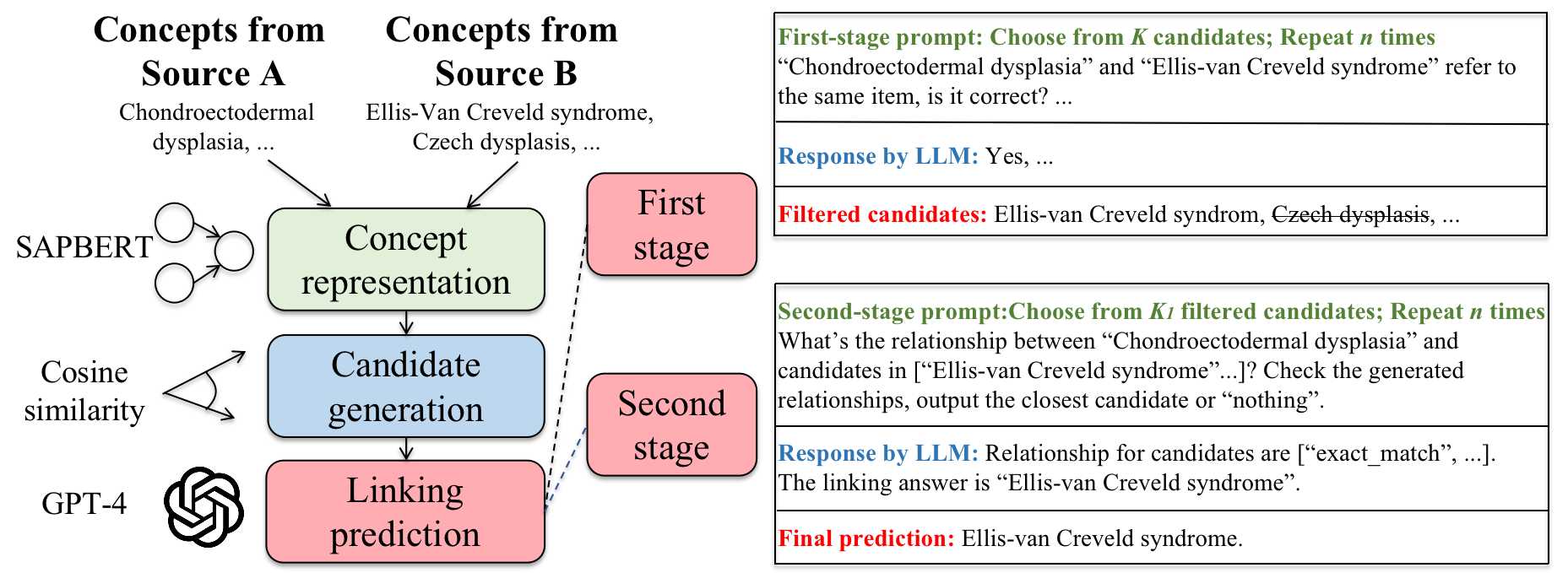
Abbildung 2: Überblick über unser vorgeschlagenes promptLink -Framework.
["Anforderungen.txt" -Datei könnte verwendet werden, um die Python -Pakete automatisch herunterzuladen]
Python == 3.8.10
editDistance == 0.6.2
Feuer == 0.5.0
Numpy == 1.19.5
OpenAI == 0,28.1
pandas == 1.3.4
RANK_BM25 == 0.2.2
scipy == 1.12.0
Simstring-Fast == 0.3.0
textDistance == 4.6.1
Fackel == 1.10.0+Cu111
tqdm == 4.66.1
Transformatoren == 4.33.3
Wir kuratieren zwei biomedizinische Konzept, die Benchmark-Datensätze verknüpfen: MIID (Mimic-iii-ibkh-disase) und Cise (Cradle-Ibkh-Side-Effect) unter Verwendung von Daten aus Mimic-III-EHR-EHR-Datensatz-Link, Cradle EHR-Datensatz (einem privaten EHR-Datensatz, das aus einem großen Gesundheitssystem aus einem großen Gesundheitssystem aus dem US-amp; IBKH Link und UMLS -Codierungssystem UMLS -Link. Aufgrund der sensiblen Natur von medizinischen Daten und Datenschutzüberlegungen gibt es Einschränkungen bei der Datenaustausch. Um Zugang zu diesen medizinischen Datensätzen zu erhalten, können geeignete Schulungen und Anmeldeinformationen erforderlich sein. Für weitere Unterstützung beim Datenzugriff oder andere damit verbundene Anfragen können Sie sich gerne an unser Autorenteam wenden.
Der größte Teil des Codes wird in drei Ordnern gespeichert: "Gen_Candidates", "Gen_gpt_responses" und "Baselines". Weitere Details finden Sie in diesen Ordnern.
Ordner "Gen_Candidates": Dieser Ordner enthält den Code für die Konzeptrepräsentation und den Prozess der Kandidatengenerierung.
Ordner "Gen_gpt_responses": Dieser Ordner zeigt, wie promptLink das LLM nutzt, um die endgültige Vorhersage -Antwort abzurufen.
Ordner "Baselines": Dieser Ordner enthält den Code zum Ausführen aller verglichenen Basismethoden, einschließlich BM25, Levenshtein Distanz, Biobert und Sapbert.