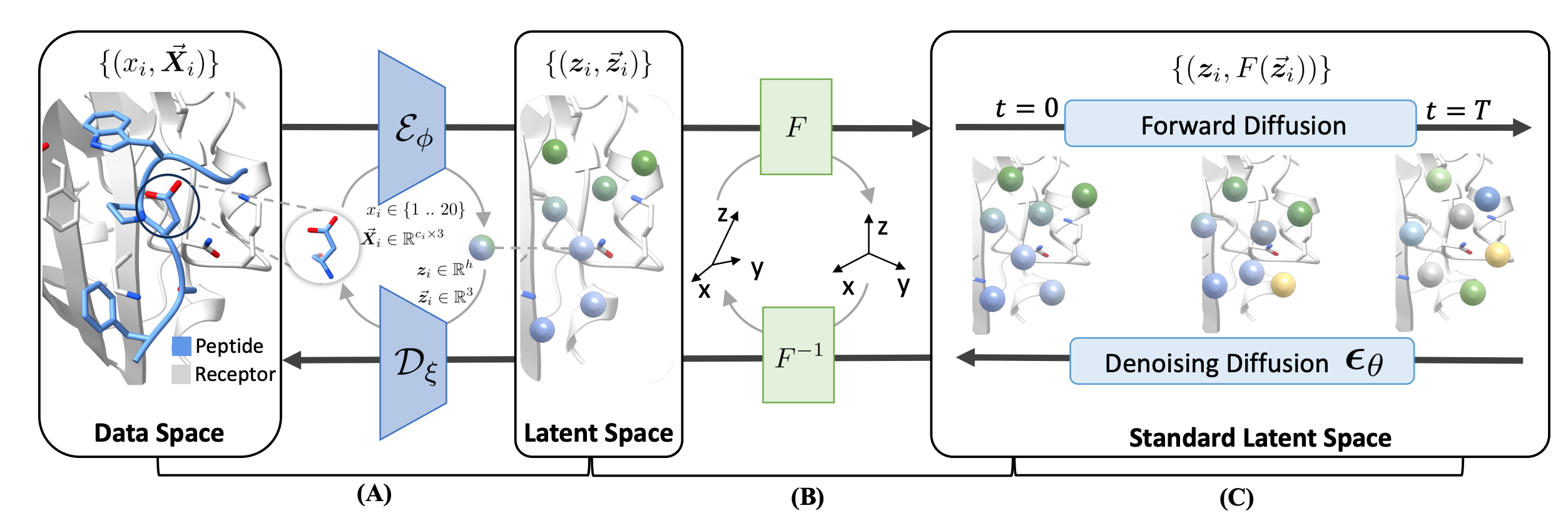
Conda環境は、構成env.yamlで構築できます。
conda env create -f env.yamlコードは、CUDAバージョン11.7およびPytorchバージョン1.13.1でテストされています。
コードを実行する前に環境をアクティブにすることを忘れないでください。
conda activate PepGLADピロセッタは、生成されたペプチドの界面エネルギーを計算するために使用されます。あなたがそれに興味があるなら、ここの指示に従ってインストールしてください。
これらのデータセットは、ベンチマークモデルにのみ使用されます。訓練されたウェイトを使用してケースの推論を使用する場合は、これらのデータセットをダウンロードする必要はありません。
このペーパーでは最初に導入されたデータセットは、このURLでZenodoにアップロードされます。次のようにダウンロードできます。
mkdir datasets # all datasets will be put into this directory
wget https://zenodo.org/records/13373108/files/train_valid.tar.gz ? download=1 -O ./datasets/train_valid.tar.gz # training/validation
wget https://zenodo.org/records/13373108/files/LNR.tar.gz ? download=1 -O ./datasets/LNR.tar.gz # test set
wget https://zenodo.org/records/13373108/files/ProtFrag.tar.gz ? download=1 -O ./datasets/ProtFrag.tar.gz # augmentation datasettar zxvf ./datasets/train_valid.tar.gz -C ./datasets
tar zxvf ./datasets/LNR.tar.gz -C ./datasets
tar zxvf ./datasets/ProtFrag.tar.gz -C ./datasetspython -m scripts.data_process.process --index ./datasets/train_valid/all.txt --out_dir ./datasets/train_valid/processed # train/validation set
python -m scripts.data_process.process --index ./datasets/LNR/test.txt --out_dir ./datasets/LNR/processed # test set
python -m scripts.data_process.process --index ./datasets/ProtFrag/all.txt --out_dir ./datasets/ProtFrag/processed # augmentation datasetトレーニング/検証スプリットの処理されたデータのインデックスは次のように生成する必要があります。これにより、 datasets/train_valid/processed/train_index.txtとdatasets/train_valid/processed/valid_index.txtになります。
python -m scripts.data_process.split --train_index datasets/train_valid/train.txt --valid_index datasets/train_valid/valid.txt --processed_dir datasets/train_valid/processed/wget http://huanglab.phys.hust.edu.cn/pepbdb/db/download/pepbdb-20200318.tgz -O ./datasets/pepbdb.tgztar zxvf ./datasets/pepbdb.tgz -C ./datasets/pepbdbpython -m scripts.data_process.pepbdb --index ./datasets/pepbdb/peptidelist.txt --out_dir ./datasets/pepbdb/processed
python -m scripts.data_process.split --train_index ./datasets/pepbdb/train.txt --valid_index ./datasets/pepbdb/valid.txt --test_index ./datasets/pepbdb/test.txt --processed_dir datasets/pepbdb/processed/
mv ./datasets/pepbdb/processed/pdbs ./dataset/pepbdb # re-locate./checkpoint/codesign.ckpt CheckPoint/Codesign.ckpt./checkpoints/fixseq.ckpt CheckPoints/fixSeq.ckptどちらもリリースページでダウンロードできます。これらのチェックポイントはペプベンチで訓練されました。
./assets/1ssc_A_B.pdbを例として、チェーンAはターゲットタンパク質です。
# obtain the binding site, which might also be manually crafted or from other ligands (e.g. small molecule, antibodies)
python -m api.detect_pocket --pdb assets/1ssc_A_B.pdb --target_chains A --ligand_chains B --out assets/1ssc_A_pocket.json
# sequence-structure codesign with length in [8, 15)
CUDA_VISIBLE_DEVICES=0 python -m api.run
--mode codesign
--pdb assets/1ssc_A_B.pdb
--pocket assets/1ssc_A_pocket.json
--out_dir ./output/codesign
--length_min 8
--length_max 15
--n_samples 10その後、10世代がフォルダー./output/codesignの下に出力されます。
./assets/1ssc_A_B.pdbを例として、チェーンAはターゲットタンパク質です。
# obtain the binding site, which might also be manually crafted or from other ligands (e.g. small molecule, antibodies)
python -m api.detect_pocket --pdb assets/1ssc_A_B.pdb --target_chains A --ligand_chains B --out assets/1ssc_A_pocket.json
# generate binding conformation
CUDA_VISIBLE_DEVICES=0 python -m api.run
--mode struct_pred
--pdb assets/1ssc_A_B.pdb
--pocket assets/1ssc_A_pocket.json
--out_dir ./output/struct_pred
--peptide_seq PYVPVHFDASV
--n_samples 10次に、10個のコンフォメーションがフォルダー./output/struct_predの下に出力されます。
各タスクには、スクリプトに統合された次の手順が必要です./scripts/run_exp_pipe.sh :
一方、既存のチェックポイントを評価する場合は、以下の指示(例えば、立体構造の生成)に従ってください。
# generate results on the test set and save to ./results/fixseq
python generate.py --config configs/pepbench/test_fixseq.yaml --ckpt checkpoints/fixseq.ckpt --gpu 0 --save_dir ./results/fixseq
# calculate metrics
python cal_metrics.py --results ./results/fixseq/results.jsonlペプベンチでのコード設計実験:
GPU=0 bash scripts/run_exp_pipe.sh pepbench_codesign configs/pepbench/autoencoder/train_codesign.yaml configs/pepbench/ldm/train_codesign.yaml configs/pepbench/ldm/setup_latent_guidance.yaml configs/pepbench/test_codesign.yamlPepbenchでの立体構造生成実験:
GPU=0 bash scripts/run_exp_pipe.sh pepbench_fixseq configs/pepbench/autoencoder/train_fixseq.yaml configs/pepbench/ldm/train_fixseq.yaml configs/pepbench/ldm/setup_latent_guidance.yaml configs/pepbench/test_fixseq.yaml私たちの仕事にあなたの興味をありがとう!
アルゴリズム、コード、およびそれらを実行する際に遭遇する問題について質問して、それをより明確かつより良くすることができるようにしてください。 Github Repoで問題を作成するか、[email protected]でお問い合わせください。
@article { kong2024full ,
title = { Full-atom peptide design with geometric latent diffusion } ,
author = { Kong, Xiangzhe and Huang, Wenbing and Liu, Yang } ,
journal = { arXiv preprint arXiv:2402.13555 } ,
year = { 2024 }
}