La métabolomique non ciblée joue un rôle crucial dans la vague de médecine de précision et de découverte de biomarqueurs. Cependant, l'identification des composés reste difficile en raison de l'incomplétude de la bibliothèque de référence du spectre existante. Pour résoudre ce problème, l'Institut fédéral allemand de la recherche et des tests de matériaux (BAM) et une équipe de recherche de l'Université libre de Berlin ont développé conjointement FIORA, un réseau neuronal de graphe open source (GNN), conçu pour simuler le processus de spectrométrie de masse en tandem pour aider à améliorer la précision de la reconnaissance de la spectrométrie de masse.
Le cœur du modèle FIORA est qu'il utilise les informations locales de quartier des liaisons dans les molécules pour apprendre les modèles de rupture des composés, dérivant ainsi la probabilité d'ions fragmentés. Par rapport aux algorithmes de fragmentation traditionnels iceberg et CFM-ID, la FIORA fonctionne excellente en prédiction de qualité et peut prédire d'autres caractéristiques telles que le temps de rétention (RT) et la section de collision (CCS). Ce résultat de recherche pionnière a été publié dans Nature Communications le 7 mars 2025.
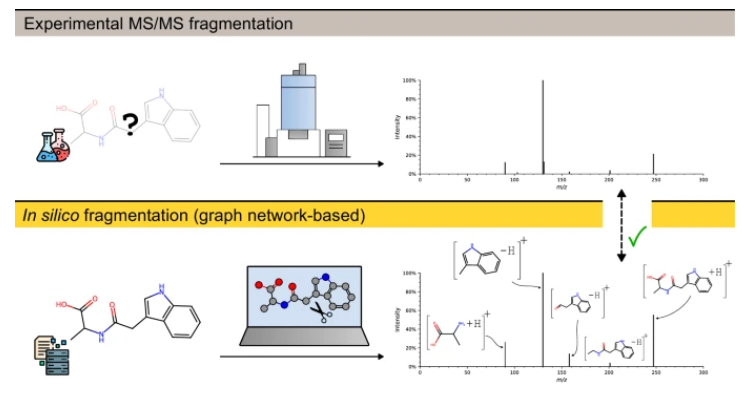
La conception de Fiora utilise pleinement des GPU haute performance, valide rapidement les annotations composées putatives et étend considérablement la bibliothèque de référence spectrale avec des prédictions de haute qualité. Ce progrès est d'une grande importance pour promouvoir la recherche sur la métabolomique non ciblée, en particulier lors de l'analyse des composés inconnus. La recherche dans ce domaine a été lente au cours de la dernière décennie en raison de la rareté du spectre de référence de haute qualité. Par exemple, le défi Casmi 2016 a montré que le taux de rappel des méthodes de simulation informatique n'était que de 34%, alors qu'il n'était même pas 30% en 2022. Cela montre le besoin urgent d'une nouvelle solution.
La FIORA est unique en ce qu'elle peut évaluer indépendamment les événements de dissociation des liaisons en fonction de la structure locale de chaque composé. Cette méthode simule le processus de fragmentation physique dans la spectrométrie de masse plus directement que de nombreux algorithmes existants. De plus, la FIORA fonctionne non seulement bien lorsque le ciblage des composés similaires, mais aussi sa capacité à promouvoir des structures inconnues est impressionnante.
Pour garantir son efficacité, FIORA a testé sur plusieurs ensembles de données et les résultats ont montré que la similitude médiane entre ses spectres de masse prédits et le spectre de référence atteignait plus de 0,8, et dans certains cas, 10% à 49% plus élevé que l'algorithme de compétition. De plus, la conception modulaire de Fiora lui permet de s'adapter de manière flexible à différents objectifs prédictifs, montrant une excellente polyvalence.
Le lancement de la FIORA comble non seulement l'écart dans l'analyse de la spectrométrie de masse, mais fournit également un outil puissant pour l'identification et la recherche des composés futurs.