واجهة R لعائلة Meme Suite of Tools ، والتي توفر عدة أدوات لتحليل الحمام على DNA ، RNA ، وتسلسل البروتين. تعمل الميمات من خلال اكتشاف تثبيت محلي لمجموعة MEME ، وتشغيل الأوامر ، ثم استيراد النتائج مباشرة إلى R.
الميمات متوفرة حاليًا على فرع devel Bioconductor:
if ( ! requireNamespace( " BiocManager " , quietly = TRUE ))
install.packages( " BiocManager " )
# The following initializes usage of Bioc devel
BiocManager :: install( version = ' devel ' )
BiocManager :: install( " memes " )يمكنك تثبيت إصدار تطوير الميمات من GitHub مع:
if ( ! requireNamespace( " remotes " , quietly = TRUE ))
install.packages( " remotes " )
remotes :: install_github( " snystrom/memes " )
# To temporarily bypass the R version 4.1 requirement, you can pull from the following branch:
remotes :: install_github( " snystrom/memes " , ref = " no-r-4 " ) # Get development version from dockerhub
docker pull snystrom/memes_docker:devel
# the -v flag is used to mount an analysis directory,
# it can be excluded for demo purposes
docker run -e PASSWORD= < password > -p 8787:8787 -v < path > / < to > / < project > :/mnt/ < project > snystrom/memes_docker:develتعتمد الميمات على تثبيت محلي لجناح الميم. للحصول على تعليمات التثبيت لجناح MEME ، راجع دليل تثبيت Meme Suite.
تحتاج الميمات إلى معرفة موقع meme/bin/ الدليل على الجهاز المحلي الخاص بك. يمكنك إخبار الميمات بموقع تثبيت Meme Suite الخاص بك بـ 4 طرق. تفضل الميمات دائمًا التعريف الأكثر تحديدًا إذا كان مسارًا صالحًا. هنا يتم تصنيفها من أكثر- إلى الأقل خاصة:
meme_path لجميع وظائف الميماتoptions(meme_bin = "/path/to/meme/bin/") داخل البرنامج النصي r الخاص بكMEME_BIN=/path/to/meme/bin/ في ملف .Renviron الخاص بك~/meme/bin/إذا فشلت الميمات في اكتشاف التثبيت في الموقع المحدد ، فسوف يعود إلى الخيار التالي.
للتحقق من الميمات يمكنها اكتشاف تثبيت MEME الخاص بك ، استخدم check_meme_install() الذي يستخدم Herirarchy Search أعلاه للعثور على تثبيت ميمي صالح. سيبلغ عن ما إذا كانت أي أدوات مفقودة ، وطباعة المسار إلى Meme الذي يراه. يمكن أن يكون هذا مفيدًا لاستكشاف مشكلات استكشاف الأخطاء وإصلاحها مع التثبيت.
library( memes )
# Verify that memes detects your meme install
# (returns all green checks if so)
check_meme_install()
# > checking main install
# > ✓ /opt/meme/bin
# > checking util installs
# > ✓ /opt/meme/bin/dreme
# > ✓ /opt/meme/bin/ame
# > ✓ /opt/meme/bin/fimo
# > ✓ /opt/meme/bin/tomtom
# > ✓ /opt/meme/bin/meme
# > x /opt/meme/bin/streme # You can manually input a path to meme_path
# If no meme/bin is detected, will return a red X
check_meme_install( meme_path = ' bad/path ' )
# > checking main install
# > x bad/path | اسم الوظيفة | يستخدم | إدخال التسلسل | مدخلات عزر | الإخراج |
|---|---|---|---|---|
runStreme() | اكتشاف الحافز (الزخارف القصيرة) | نعم | لا | universalmotif_df |
runDreme() | اكتشاف الحافز (الزخارف القصيرة) | نعم | لا | universalmotif_df |
runAme() | إثراء الحافز | نعم | نعم | data.frame (اختياري: عمود sequences ) |
runFimo() | المسح الحافز | نعم | نعم | Granges of motif |
runTomTom() | عزر مقارنة | لا | نعم | universalmotif_df w/ best_match_motif و tomtom Columns* |
runMeme() | اكتشاف الحافز (الزخارف الطويلة) | نعم | لا | universalmotif_df |
* ملاحظة: إذا تم تشغيل runTomTom() باستخدام universalmotif_df فسيتم ربط النتائج بنتائج universalmotif_df كأعمدة إضافية. هذا يسمح بالمقارنة السهلة مع DE-NOVO المكتشفة الزخارف مع مبارياتها.
يمكن أن تكون مدخلات التسلسل أي من:
Biostrings::XStringSet (يمكن إنشاء من Granges باستخدام وظيفة المساعد get_sequence() )Biostrings::XStringSet (التي تم إنشاؤها بواسطة get_sequence() )يمكن أن تكون مدخلات الحافز أيًا من:
universalmotif ، أو قائمة كائنات universalmotifrunDreme() (يسمح لنتائج runDreme() بالمرور مباشرة إلى runTomTom() )list() (على سبيل المثال list("path/to/database.meme", "dreme_results" = dreme_res) )أنواع الإخراج :
runDreme() ، runStreme() ، runMeme() و runTomTom() إرجاع كائنات universalmotif_df التي هي data.frames مع أعمدة خاصة. يحتوي عمود motif على كائن universalmotif ، مع إدخال واحد لكل صف. تصف الأعمدة المتبقية خصائص كل عزر عائد. تكون أسماء الأعمدة التالية خاصة من خلال استخدام قيمها عند تشغيل update_motifs() و to_list() لتغيير خصائص الزخارف المخزنة في العمود motif . كن حذرًا بشأن تغيير هذه القيم لأن هذه التغييرات ستنتشر إلى عمود motif عند استدعاء update_motifs() أو to_list() .
تم بناء MEMES حول حزمة Universalmotif التي يوفر إطار عمل لمعالجة الزخارف في كائنات R. universalmotif_df يمكن أن تتداخل بين تنسيق Data.Frame و universalmotif باستخدام وظائف to_df() و to_list() على التوالي. يتيح ذلك استخدام نتائج memes مع جميع حزم مواد الموصلات الحيوية الأخرى ، حيث يمكن أن تتحول كائنات universalmotif إلى أي نوع عزر آخر باستخدام convert_motifs() .
runTomTom() إرجاع عمود خاص: tomtom وهو data.frame إطار جميع بيانات المطابقة لكل عزر إدخال. يمكن توسيع هذا باستخدام tidyr::unnest(tomtom_results, "tomtom") ، وإعادة تعبئته مع nest_tomtom() . تشير الأعمدة المسبقة best_match_ التي يتم إرجاعها بواسطة runTomTom() إلى قيم الفكرة التي كانت أفضل تطابق مع عزر الإدخال.
suppressPackageStartupMessages(library( magrittr ))
suppressPackageStartupMessages(library( GenomicRanges ))
# Example transcription factor peaks as GRanges
data( " example_peaks " , package = " memes " )
# Genome object
dm.genome <- BSgenome.Dmelanogaster.UCSC.dm6 :: BSgenome.Dmelanogaster.UCSC.dm6 تأخذ وظيفة get_sequence GRanges أو GRangesList كمدخلات وتُرجع التسلسلات باعتبارها BioStrings::XStringSet ، أو قائمة كائنات XStringSet ، على التوالي. سوف get_sequence تسمية كل إدخال fasta بواسطة الإحداثيات الجينية كل تسلسل من.
# Generate sequences from 200bp about the center of my peaks of interest
sequences <- example_peaks % > %
resize( 200 , " center " ) % > %
get_sequence( dm.genome ) runDreme() يقبل XStringset أو مسار إلى ملف fasta كمدخل. يمكنك استخدام تسلسلات أخرى أو تسلسل الإدخال المخلل كمجموعة بيانات التحكم.
# runDreme accepts all arguments that the commandline version of dreme accepts
# here I set e = 50 to detect motifs in the limited example peak list
# In a real analysis, e should typically be < 1
dreme_results <- runDreme( sequences , control = " shuffle " , e = 50 ) تم بناء الميمات حول حزمة Universalmotif. يتم إرجاع النتائج بتنسيق universalmotif_df ، وهو بيانات R. الإطار الذي يمكن أن يتداخل بسلاسة بين تنسيق البيانات. الإطار وتنسيق universalmotif باستخدام to_list() للتحويل إلى تنسيق قائمة universalmotif ، و to_df() لتحويل مرة أخرى إلى تنسيق data.frame. يتيح استخدام to_list() استخدام نتائج memes مع جميع وظائف universalmotif مثل SO:
library( universalmotif )
dreme_results % > %
to_list() % > %
view_motifs()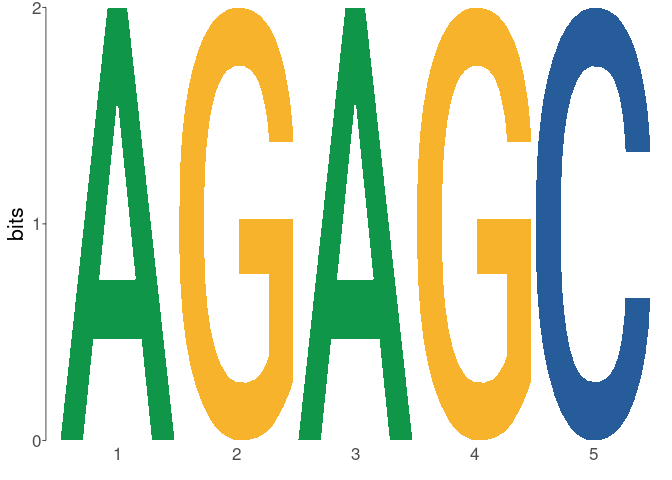
يمكن مطابقة الزخارف المكتشفة مع أشكال TF المعروفة باستخدام runTomTom() ، والتي يمكن أن تقبل كإدخال مسار إلى ملف تنسيق .meme ، أو قائمة universalmotif ، أو نتائج runDreme() .
يستخدم TomTom قاعدة بيانات للزخارف المعروفة التي يمكن تمريرها إلى معلمة database كمسار إلى ملف تنسيق .meme ، أو كائن universalmotif .
اختياريا ، يمكنك تعيين متغير البيئة MEME_DB في .Renviron إلى ملف على القرص ، أو قيمة meme_db في options إلى ملف تنسيق .meme صالح وستستخدم الميمات هذا الملف كقاعدة البيانات. تفضل الميمات دائمًا إدخال المستخدم على استدعاء الوظيفة عبر إعداد متغير عالمي.
options( meme_db = system.file( " extdata/flyFactorSurvey_cleaned.meme " , package = " memes " ))
m <- create_motif( " CMATTACN " , altname = " testMotif " )
tomtom_results <- runTomTom( m ) tomtom_results
# > motif name altname consensus alphabet strand icscore type
# > 1 <mot:motif> motif testMotif CMATTACN DNA +- 13 PPM
# > bkg best_match_name best_match_altname
# > 1 0.25, 0.25, 0.25, 0.25 prd_FlyReg prd
# > best_db_name best_match_offset best_match_pval best_match_eval
# > 1 flyFactorSurvey_cleaned 0 9.36e-05 0.052
# > best_match_qval best_match_strand
# > 1 0.0353 +
# > best_match_motif
# > 1 <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>
#> tomtom
#> 1 prd_FlyReg, tup_SOLEXA_10, CG13424_Cell, CG11085_Cell, BH2_Cell, CG13424_SOLEXA_2, Tup_Cell, Tup_SOLEXA, Bsh_Cell, Exex_SOLEXA, Odsh_SOLEXA, Unc4_Cell, Ubx_FlyReg, Unc4_SOLEXA, E5_Cell, inv_SOLEXA_5, BH2_SOLEXA, Zen_SOLEXA, CG33980_SOLEXA_2_10, BH1_SOLEXA, CG33980_SOLEXA_2_0, Hgtx_Cell, NK7.1_Cell, Slou_Cell, CG13424_SOLEXA, Zen2_Cell, AbdA_SOLEXA, Antp_SOLEXA, Btn_Cell, Dfd_SOLEXA, Eve_SOLEXA, Ftz_Cell, Hmx_SOLEXA, Hmx_Cell, CG34031_Cell, zen2_SOLEXA_2, En_Cell, Pb_SOLEXA, Slou_SOLEXA, Unpg_Cell, inv_SOLEXA_2, ovo_FlyReg, lim_SOLEXA_2, C15_SOLEXA, Ems_Cell, Btn_SOLEXA, Unpg_SOLEXA, Pb_Cell, Bsh_SOLEXA, Scr_SOLEXA, Zen2_SOLEXA, CG34031_SOLEXA, Eve_Cell, Pph13_Cell, BH1_Cell, CG11085_SOLEXA, CG32532_Cell, en_FlyReg, Dll_SOLEXA, Dfd_Cell, Dr_SOLEXA, Ap_Cell, Ro_Cell, CG4136_SOLEXA, CG33980_SOLEXA, Hbn_SOLEXA, Lbl_Cell, Otp_Cell, Rx_Cell, CG32532_SOLEXA, NK7.1_SOLEXA, Dr_Cell, Odsh_Cell, Al_SOLEXA, Antp_Cell, Hgtx_SOLEXA, Ftz_SOLEXA, Lab_SOLEXA, Dfd_FlyReg, Ap_SOLEXA, Awh_SOLEXA, CG11294_SOLEXA, CG4136_Cell, E5_SOLEXA, Ro_SOLEXA, PhdP_SOLEXA, CG12361_SOLEXA_2, Ind_Cell, Scr_Cell, CG9876_Cell, CG18599_Cell, CG9876_SOLEXA, Otp_SOLEXA, Lbl_SOLEXA, Ubx_Cell, Ubx_SOLEXA, en_SOLEXA_2, Pph13_SOLEXA, Rx_SOLEXA, CG15696_SOLEXA, CG18599_SOLEXA, Ems_SOLEXA, Repo_Cell, Dll_Cell, C15_Cell, CG12361_SOLEXA, Abd-A_FlyReg, Repo_SOLEXA, Zen_Cell, Inv_Cell, En_SOLEXA, Lim3_Cell, Lim1_SOLEXA, CG15696_Cell, Crc_CG6272_SANGER_5, Lab_Cell, CG32105_SOLEXA, Bap_SOLEXA, CG9437_SANGER_5, AbdA_Cell, pho_FlyReg, CG33980_Cell, Cad_SOLEXA, CG4328_SOLEXA, CG4328_Cell, Gsc_Cell, vri_SANGER_5, AbdB_SOLEXA, Xrp1_CG6272_SANGER_5, Al_Cell, Exex_Cell, br-Z4_FlyReg, CG11294_Cell, Aef1_FlyReg, CG7745_SANGER_5, PhdP_Cell, Awh_Cell, prd, tup, lms, CG11085, B-H2, lms, tup, tup, bsh, exex, OdsH, unc-4, Ubx, unc-4, E5, inv, B-H2, zen, CG33980, B-H1, CG33980, HGTX, NK7.1, slou, lms, zen2, abd-A, Antp, btn, Dfd, eve, ftz, Hmx, Hmx, CG34031, zen2, en, pb, slou, unpg, inv, ovo, Lim1, C15, ems, btn, unpg, pb, bsh, Scr, zen2, CG34031, eve, Pph13, B-H1, CG11085, CG32532, en, Dll, Dfd, Dr, ap, ro, CG4136, CG33980, hbn, lbl, otp, Rx, CG32532, NK7.1, Dr, OdsH, al, Antp, HGTX, ftz, lab, Dfd, ap, Awh, CG11294, CG4136, E5, ro, PHDP, CG12361, ind, Scr, CG9876, CG18599, CG9876, otp, lbl, Ubx, Ubx, en, Pph13, Rx, CG15696, CG18599, ems, repo, Dll, C15, CG12361, abd-A, repo, zen, inv, en, Lim3, Lim1, CG15696, crc, lab, CG32105, bap, CG9437, abd-A, pho, CG33980, cad, CG4328, CG4328, Gsc, vri, Abd-B, Xrp1, al, exex, br, CG11294, Aef1, CG7745, PHDP, Awh, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, <S4 class 'universalmotif' [package "universalmotif"] with 20 slots>, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, flyFactorSurvey_cleaned, 0, 1, 0, 1, 1, 1, 0, 0, 1, 0, 0, 0, 0, 0, 0, 1, 0, 0, 1, 0, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 1, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 3, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, -1, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 1, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, -2, 0, 0, 0, -3, 0, 4, 1, 0, 0, 0, 0, -2, 0, -2, 1, 0, -2, 1, 0, 2, -1, 0, 9.36e-05, 0.000129, 0.00013, 0.000194, 0.000246, 0.000247, 0.000319, 0.000364, 0.000427, 0.000435, 0.000473, 0.000473, 0.000556, 0.000643, 0.000694, 0.000728, 0.000748, 0.000748, 0.000785, 0.000828, 0.000849, 0.000868, 0.000868, 0.000868, 0.00102, 0.00113, 0.00116, 0.00116, 0.00116, 0.00116, 0.00116, 0.00116, 0.00118, 0.00126, 0.00134, 0.00141, 0.00144, 0.00145, 0.00145, 0.00154, 0.00161, 0.00165, 0.00173, 0.00177, 0.00177, 0.00179, 0.00179, 0.0019, 0.00191, 0.00191, 0.00191, 0.00203, 0.00203, 0.00203, 0.00212, 0.00214, 0.00219, 0.00232, 0.00242, 0.00247, 0.0026, 0.00264, 0.00264, 0.00268, 0.00282, 0.00282, 0.00282, 0.00282, 0.00282, 0.00286, 0.00286, 0.00296, 0.00305, 0.00316, 0.0032, 0.0032, 0.00326, 0.00326, 0.00341, 0.00348, 0.00348, 0.00348, 0.00348, 0.00348, 0.00348, 0.0035, 0.00359, 0.00359, 0.00359, 0.00364, 0.00372, 0.00372, 0.00372, 0.00387, 0.00387, 0.00412, 0.00415, 0.00424, 0.00424, 0.00439, 0.00452, 0.00452, 0.00467, 0.00483, 0.00497, 0.00497, 0.00528, 0.00549, 0.0059, 0.00597, 0.00624, 0.00635, 0.00709, 0.00761, 0.0079, 0.00856, 0.00859, 0.00912, 0.00935, 0.0097, 0.00974, 0.0101, 0.0109, 0.0116, 0.0123, 0.0123, 0.0127, 0.0138, 0.0138, 0.014, 0.014, 0.0147, 0.0148, 0.0155, 0.0165, 0.0166, 0.0177, 0.052, 0.0718, 0.0725, 0.108, 0.137, 0.137, 0.177, 0.202, 0.238, 0.242, 0.263, 0.263, 0.309, 0.357, 0.386, 0.405, 0.416, 0.416, 0.436, 0.46, 0.472, 0.483, 0.483, 0.483, 0.566, 0.629, 0.646, 0.646, 0.646, 0.646, 0.646, 0.646, 0.654, 0.702, 0.745, 0.785, 0.8, 0.808, 0.808, 0.858, 0.897, 0.92, 0.961, 0.985, 0.985, 0.993, 0.993, 1.05, 1.06, 1.06, 1.06, 1.13, 1.13, 1.13, 1.18, 1.19, 1.22, 1.29, 1.34, 1.38, 1.45, 1.47, 1.47, 1.49, 1.57, 1.57, 1.57, 1.57, 1.57, 1.59, 1.59, 1.65, 1.7, 1.76, 1.78, 1.78, 1.81, 1.81, 1.9, 1.94, 1.94, 1.94, 1.94, 1.94, 1.94, 1.95, 2, 2, 2, 2.02, 2.07, 2.07, 2.07, 2.15, 2.15, 2.29, 2.31, 2.36, 2.36, 2.44, 2.52, 2.52, 2.6, 2.68, 2.76, 2.76, 2.94, 3.05, 3.28, 3.32, 3.47, 3.53, 3.94, 4.23, 4.39, 4.76, 4.77, 5.07, 5.2, 5.39, 5.41, 5.61, 6.06, 6.42, 6.81, 6.81, 7.03, 7.66, 7.65, 7.78, 7.78, 8.15, 8.26, 8.59, 9.18, 9.2, 9.85, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0353, 0.0368, 0.0369, 0.0369, 0.0369, 0.0369, 0.0369, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0372, 0.0379, 0.0379, 0.0381, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0396, 0.0404, 0.0404, 0.0424, 0.0424, 0.0424, 0.0424, 0.0435, 0.0439, 0.0439, 0.0449, 0.046, 0.0464, 0.0464, 0.0489, 0.0504, 0.0536, 0.0538, 0.0557, 0.0562, 0.0622, 0.0662, 0.0681, 0.0727, 0.0727, 0.0765, 0.0778, 0.0797, 0.0797, 0.0819, 0.0877, 0.0923, 0.0964, 0.0964, 0.0987, 0.106, 0.106, 0.106, 0.106, 0.11, 0.111, 0.114, 0.121, 0.121, 0.128, +, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, -, +, -, -, -, -, -, -, -, +, -, -, -, +, -, -, -, -, -, -, -, +, -, +, -, -, -, +, +, +, -, -
# >
# > [Hidden empty columns: family, organism, nsites, bkgsites, pval, qval,
# > eval.] سيضيف runTomTom() نتائجه كأعمدة إلى بيانات runDreme() .
full_results <- dreme_results % > %
runTomTom() يستخدم AME لاختبار إثراء الزخارف المعروفة في التسلسلات المستهدفة. سوف يستخدم runAme() إدخال MEME_DB في .Renviron أو options(meme_db = "path/to/database.meme") كقاعدة بيانات motif. بالتناوب ، سوف يقبل جميع المدخلات الصحيحة مماثلة لـ runTomTom() .
# here I set the evalue_report_threshold = 30 to detect motifs in the limited example sequences
# In a real analysis, evalue_report_threshold should be carefully selected
ame_results <- runAme( sequences , control = " shuffle " , evalue_report_threshold = 30 )
ame_results
# > # A tibble: 2 x 17
# > rank motif_db motif_id motif_alt_id consensus pvalue adj.pvalue evalue tests
# > <int> <chr> <chr> <chr> <chr> <dbl> <dbl> <dbl> <int>
# > 1 1 /usr/lo… Eip93F_… Eip93F ACWSCCRA… 5.14e-4 0.0339 18.8 67
# > 2 2 /usr/lo… Cf2-PB_… Cf2 CSSHNKDT… 1.57e-3 0.0415 23.1 27
# > # … with 8 more variables: fasta_max <dbl>, pos <int>, neg <int>,
# > # pwm_min <dbl>, tp <int>, tp_percent <dbl>, fp <int>, fp_percent <dbl> تسمح view_tomtom_hits بمقارنة زخارف الإدخال بأعلى الزيارات من TomTom. يعد التفتيش اليدوي لهذه المباريات أمرًا مهمًا ، حيث في بعض الأحيان لا تكون المباراة العليا هي المهمة الصحيحة دائمًا. يتيح لك تغيير top_n إظهار مباريات إضافية بترتيب تنازلي من رتبهم.
full_results % > %
view_tomtom_hits( top_n = 1 )
# > $m01_AGAGC 
قد يكون من المفيد عرض النتائج من runAme() كخريطة حرارية. يمكن أن يخلق plot_ame_heatmap() تصورات معقدة لتحليل الإثراء بين أنواع المناطق المختلفة (انظر المقالات القصيرة للحصول على التفاصيل). هنا مثال بسيط على خريطة الحرارة.
ame_results % > %
plot_ame_heatmap()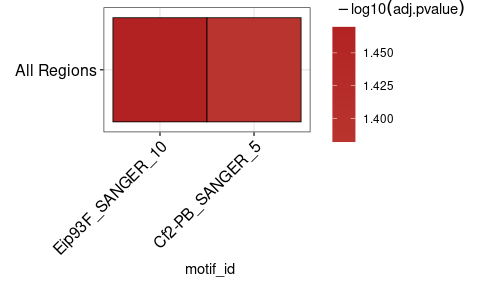
يتم استخدام أداة FIMO لتحديد التطابقات مع الزخارف المعروفة. سيقوم runFimo بإرجاع هذه الزيارات ككائن GRanges الذي يحتوي على الإحداثيات الجينية لمطابقة الحافز.
# Query MotifDb for a motif
e93_motif <- MotifDb :: query( MotifDb :: MotifDb , " Eip93F " ) % > %
universalmotif :: convert_motifs()
# > See system.file("LICENSE", package="MotifDb") for use restrictions.
# Scan for the E93 motif within given sequences
fimo_results <- runFimo( sequences , e93_motif , thresh = 1e-3 )
# Visualize the sequences matching the E93 motif
plot_sequence_heatmap( fimo_results $ matched_sequence ) 
تدعم MEMES أيضًا استيراد النتائج التي تم إنشاؤها باستخدام مجموعة MEME خارج R (على سبيل المثال ، تشغيل الوظائف على meme-suite.org ، أو التشغيل على سطر الأوامر). هذا يتيح استخدام نتائج جناح MEME مسبقًا مع وظائف الميمات المصب.
| أداة الميم | اسم الوظيفة | نوع الملف |
|---|---|---|
| ستريم | importStremeXML() | streme.xml |
| دري | importDremeXML() | dreme.xml |
| تومتون | importTomTomXML() | tomtom.xml |
| آمي | importAme() | ame.tsv* |
| فيمو | importFimo() | fimo.tsv |
| ميمي | importMeme() | Meme.txt |
* يمكن أن تستخدم أيضًا importAME() إخراج "sequences.tsv" عندما يستخدم AME method = "fisher" ، وهذا اختياري.
لا يدعم مجموعة MEME Windows حاليًا ، على الرغم من أنه يمكن تثبيته تحت Cygwin أو Windows Linux Subsytem (WSL). يرجى ملاحظة أنه إذا تم تثبيت MEME على Cygwin أو WSL ، فيجب عليك أيضًا تشغيل R داخل Cygwin أو WSL لاستخدام الميمات.
يتمثل الحل البديل في استخدام Docker لتشغيل بيئة افتراضية مع تثبيت Suite Meme. نحن نقدم حاوية ميمات ميناء
هذا السفن مع جناح Meme ، R studio ، وجميع تبعيات memes مثبتة مسبقًا.
MEMES عبارة عن غلاف لبعض الأدوات المختارة من مجموعة MEME ، والتي تم تطويرها بواسطة مجموعة أخرى. بالإضافة إلى الإشارة إلى الميمات ، يرجى الاستشهاد بأدوات Meme Suite المقابلة للأدوات التي تستخدمها.
إذا كنت تستخدم runDreme() في تحليلك ، يرجى الاستشهاد:
Timothy L. Bailey ، "DREME: MOTIF Discovery في بيانات Chip-Seq من عامل النسخ" ، المعلوماتية الحيوية ، 27 (12): 1653-1659 ، 2011. النص الكامل
إذا كنت تستخدم runTomTom() في تحليلك ، يرجى الاستشهاد:
Shobhit Gupta ، Ja Stamatoyannopolous ، Timothy Bailey و William Stafford Noble ، "تحديد التشابه بين الزخارف" ، بيولوجيا الجينوم ، 8 (2): R24 ، 2007. النص الكامل
إذا كنت تستخدم runAme() في تحليلك ، يرجى الاستشهاد:
Robert McLey و Timothy L. Bailey ، "تحليل إثراء العزر: إطار موحد وتقييم الطريقة" ، BMC Bioinformatics ، 11: 165 ، 2010 ، doi: 10.1186/1471-2105-11-165. النص الكامل
إذا كنت تستخدم runFimo() في تحليلك ، يرجى الاستشهاد:
تشارلز إ. جرانت ، تيموثي ل. بيلي ، وويليام ستافورد نوبل ، "فيمو: المسح الضوئي لأحداث عزر معين" ، المعلوماتية الحيوية ، 27 (7): 1017-1018 ، 2011. النص الكامل
يعد Meme Suite مجانيًا للاستخدام غير الربحي ، ولكن يجب على المستخدمين الربحية شراء ترخيص. انظر صفحة حقوق الطبع والنشر لمجموعة Meme Suite للحصول على التفاصيل.